Enterobacter
Enterobacter ist eine Gattung von Bakterien, die zu der Familie der Enterobacteriaceae gehört und etwa 15 Arten umfasst. Vertreter der Gattung Enterobacter kommen in fast allen Lebensräumen einschließlich des menschlichen Darms vor. Dort gehören sie zur normalen Darmflora. Die stäbchenförmigen Bakterienzellen werden in der Gram-Färbung rot angefärbt und zählen daher zu der Gruppe der gramnegativen Bakterien. Sie sind in der Lage, sich mit Hilfe von Flagellen aktiv zu bewegen. Sie können mit oder ohne Sauerstoff leben und werden somit als fakultativ anaerobe Lebensformen bezeichnet. Wenn kein Sauerstoff vorhanden ist, führen sie zur Energiegewinnung eine Gärung durch. Die für Enterobacter typische Gärung ist die 2,3-Butandiol-Gärung – dies ist ein wichtiges Unterscheidungsmerkmal zu verwandten Gattungen.
| Enterobacter | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
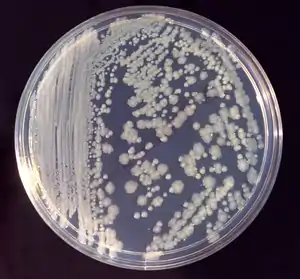
Enterobacter cloacae | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Enterobacter | ||||||||||||
| Hormaeche & Edwards 1960 emend. Brady et al. 2013 |
Generelle Aussagen zur Pathogenität von Enterobacter als Krankheitserreger sind schwierig: Es gibt Arten, die als nicht pathogen angesehen werden, aber ebenso Arten, die eine Krankheit beim Menschen hervorrufen können (Einstufung in die Risikogruppe 2 gemäß Biostoffverordnung). Früher nahm man an, dass Vertreter der Gattung allenfalls als opportunistische Krankheitserreger zu sehen sind, die bei Patienten mit einem bereits geschwächten Immunsystem Infektionskrankheiten verursachen können. Die seit wenigen Jahrzehnten beobachtete Antibiotikaresistenz von einigen Enterobacter-Arten (vor allem Enterobacter cloacae) führt dazu, dass sie mittlerweile als Erreger von im Krankenhaus erworbenen Infektionen – nosokomialen Infektionen – von zunehmender Bedeutung sind. Darüber hinaus ist bemerkenswert, dass sich die Systematik der Gattung bzw. verwandter Gattungen aufgrund phylogenetischer Untersuchungen verschiedener Gene beständig ändert. Dies hat dazu geführt, dass einige Bakterien-Arten inzwischen nicht mehr der Gattung Enterobacter zugerechnet werden, sondern anderen Gattungen, die zum Teil neu beschrieben wurden. So gehört beispielsweise der bei einigen Pflanzen vorkommende Enterobacter agglomerans seit 1989 zu einer eigenen Gattung Pantoea und die medizinisch relevanten Arten Enterobacter aerogenes und Enterobacter sakazakii werden heute als Klebsiella aerogenes beziehungsweise Cronobacter sakazakii geführt.
Merkmale
Erscheinungsbild
Die Zellen von Enterobacter-Arten sind stäbchenförmig und durch Flagellen aktiv beweglich (motil), Letzteres unterscheidet sie von der verwandten Gattung Klebsiella. Die Flagellen (auch als Geißeln bezeichnet) sind peritrich angeordnet. Eine einzelne Zelle hat eine Breite von etwa 0,5 µm bei einer Länge von 2–3 µm. In der Gram-Färbung verhalten sich die Zellen gramnegativ, werden also durch die verwendeten Farbstoffe rosa bis rot angefärbt. Es werden keine Überdauerungsformen wie Endosporen gebildet.[1][2]
Bei Enterobacter cloacae ist die Bakterienzellwand von einer Kapsel umgeben, die aus Polysacchariden besteht. Sie sind an der Ausbildung von Biofilmen beteiligt und verleihen den auf einem Nährboden gewachsenen Bakterienkolonien ein schleimiges Aussehen. Manchmal gibt es auch Zellen, denen die Kapsel fehlt,[3] die gewachsenen Kolonien sehen dann eher rau aus, dies ist auf dem Bild oben zu erkennen. Auch andere Enterobacter-Arten bilden eine Kapsel aus,[4] jedoch ergibt sich innerhalb der Gattung kein einheitliches Bild.[5] Vertreter der Gattung Enterobacter zeigen gutes Wachstum auf gängigen Nährmedien.[1] Ihre Kolonien sind meist nicht besonders gefärbt, im Unterschied zu den gelblich gefärbten Kolonien von Cronobacter sakazakii.[6]
Wachstum und Stoffwechsel
Die Angehörigen der Gattung Enterobacter sind chemoorganotroph, d. h., sie bauen zur Energiegewinnung organische Stoffe ab. Sie sind fakultativ anaerob: Wenn Sauerstoff vorhanden ist, können sie einen oxidativen Energiestoffwechsel durchführen, sie oxidieren die organischen Stoffe zu Kohlenstoffdioxid (CO2) und Wasser; wenn kein Sauerstoff vorhanden ist, also unter anoxischen Bedingungen, nutzen sie die 2,3-Butandiol-Gärung zur Energiegewinnung. Hierbei entstehen als Endprodukte vor allem in großen Mengen der Alkohol 2,3-Butandiol und CO2, daneben in geringen Mengen u. a. verschiedene Säuren. Bei anderen Gattungen der Familie Enterobacteriaceae wie z. B. Escherichia und Salmonella ist die Gemischte Säuregärung der anaerobe Energiestoffwechselweg, wobei im Gegensatz zu der Butandiolgärung große Mengen von Säuren (Essigsäure, Milchsäure und Bernsteinsäure) als Endprodukte entstehen, aber kein Butandiol. Dieses Merkmal wird zur Unterscheidung der Enterobakterien-Gattungen genutzt[7] (siehe Abschnitt Nachweise). Wie bei den Enterobacteriaceae typisch, verlaufen der Katalase-Test positiv und der Oxidase-Test negativ.[7]
Für die Kultivierung sind einfache Nährmedien geeignet, es sind keine besonderen Wachstumsfaktoren notwendig. Die Bakterien lassen sich beispielsweise auf Casein-Soja-Pepton-Agar (CASO-Agar) anzüchten, auch Blutagar ist geeignet sowie Selektivnährmedien, die zur Isolierung und Unterscheidung von Vertretern der Enterobakterien geeignet sind, beispielsweise MacConkey-Agar oder Eosin-Methylen-Blau-Agar (EMB).[6] Enterobacter-Arten sind mesophil, optimales Wachstum erfolgt in einem Temperaturbereich von 30 bis 37 °C, dabei sind nach eintägiger Inkubation bereits Kolonien mit einem Durchmesser von 2–3 mm sichtbar. Einige Arten, z. B. E. cloacae wachsen auch bei niedrigeren Temperaturen (15–25 °C), beispielsweise im Brackwasser von Küstenregionen mit gemäßigtem Klima.[3]
Chemotaxonomie
Der GC-Gehalt, also der Anteil der Nukleinbasen Guanin und Cytosin in der Bakterien-DNA, liegt bei 52–60 Molprozent.[7] Bestandteile der Bakterienzelle wirken als Antigene, von diagnostischer Bedeutung sind die somatischen O-Antigene und die H-Antigene[8] (man vergleiche das bei den Salmonellen angewendete Kauffmann-White-Schema).
Pathogenität
Generelle Aussagen zur Pathogenität von Enterobacter sind schwierig, da sich die Systematik der Gattung bzw. verwandter Gattungen beständig ändert. Weiterhin liegt in älteren Berichten häufig eine Verwechslung oder nicht klare Unterscheidung von Enterobacter und Klebsiella vor.[9] In der Vergangenheit als medizinisch relevant betrachtete Arten gehören mittlerweile nicht mehr der Gattung an (die bekannten Beispiele E. aerogenes und E. sakazakii).[10][11] In der Fachliteratur werden oftmals gemeinsame Aussagen für E. cloacae und E. aerogenes gemacht,[5][12] so dass mit Überführung der zuletzt genannten Bakterienart in die Gattung Klebsiella (2017) eine eindeutige Zuordnung von Aussagen zur Pathogenität problematisch ist.
Mitte des 20. Jahrhunderts wurden Enterobacter-Arten selten als pathogen bezeichnet, dies änderte sich durch den zunehmenden Verbrauch an Antibiotika. Mittlerweile spielen mehrere Vertreter der Gattung bei nosokomialen Infektionen („Krankenhausinfektionen“) eine Rolle, vor allem da sie gegen einige Antibiotika resistent sind.[9] Durch Infektionen, die im Krankenhaus erfolgen, sind besonders immunsupprimierte Patienten gefährdet,[12] in dem Zusammenhang wird von Harnwegsinfekten und Bakteriämien berichtet. E. cancerogenus (E. taylorae) wurde 1987 bei einem Fall von Osteomyelitis, einer infektiösen Entzündung des Knochenmarks, als Erreger genannt.[9]
E. asburiae, E. bugandensis, E. cancerogenus, E. hormaechei, E. kobei, E. ludwigii und E. cloacae subsp. cloacae werden durch die Biostoffverordnung in Verbindung mit der TRBA (Technische Regeln für Biologische Arbeitsstoffe) 466 der Risikogruppe 2 zugeordnet. Die Arten E. mori, E. muelleri, E. siamensis, E. soli, E. tabaci, E. xiangfangensis und E. cloacae subsp. dissolvens gehören der Risikogruppe 1 an (sie werden als apathogen angesehen).[13]
Nachweise

Für die Isolierung der Bakterien aus Umweltproben oder klinischem Material gibt es kein spezielles Nährmedium, das selektiv für Enterobacter-Arten ist. Stattdessen verwendet man Selektivnährmedien für Enterobakterien, mit dann folgender weiterer Identifizierung.
Biochemische Nachweise
Biochemische Merkmale, wie beispielsweise die vorhandenen Enzyme und die daraus resultierenden Stoffwechseleigenschaften, können in einer Bunten Reihe zur Identifizierung von Enterobacter-Arten bzw. Unterscheidung dieser von anderen Vertretern der Enterobacteriaceae genutzt werden.[6] Typischerweise wird der Voges-Proskauer-Test eingesetzt.[1] Durch den Test wird Acetoin, ein Zwischenprodukt der 2,3-Butandiol-Gärung, nachgewiesen. Enterobacter und Klebsiella reagieren hierbei positiv, Escherichia hingegen negativ. Auch weitere Reaktionen im sogenannten IMViC-Testverfahren dienen der Unterscheidung von Escherichia und Enterobacter:[7]
| Mikroorganismus | Indolbildung | Methylrotprobe | Acetoinbildung | Citratverwertung |
|---|---|---|---|---|
| Escherichia coli | + | + | − | − |
| Enterobacter cloacae | − | − | + | + |
Vertreter der Gattung Enterobacter verwerten die Kohlenhydrate Glucose und Lactose in einer Gärung unter Bildung von Gas und Säure, bei der Vergärung von Glucose entsteht viel Kohlenstoffdioxid (CO2), mindestens doppelt so viel wie Wasserstoff (H2).[1] Bei einzelnen Bakterienstämmen ergibt sich in Bezug auf die Lactoseverwertung kein einheitliches Bild, bei einigen Arten erfolgt daher die Angabe „variabel“, d. h., dass es sowohl Stämme gibt, die Lactose abbauen können, wie auch Stämme, die dies nicht tun. Bei Selektivnährmedien erfolgt der Nachweis des Lactoseabbaus häufig über die Säurebildung beim fermentativen Abbau des Kohlenhydrats, durch die Säurebildung verändert der im Nährmedium enthaltene pH-Indikator seine Farbe. Manche Stämme zeigen möglicherweise ein negatives oder nur schwach positives Ergebnis, da zu wenig Säure produziert wird.[6] Hingegen verläuft der ONPG-Test positiv, auch wenn der Lactosenachweis durch Säurebildung nach 48-stündiger Inkubation negativ war.[14] Dieser biochemische Nachweis zeigt, dass die Bakterien über das Enzym β-Galactosidase verfügen, mit dem Lactose in die beiden Bestandteile Glucose und Galactose hydrolysiert wird.
Zu den weiteren Kohlenhydraten, die viele Vertreter der Gattung verwerten können, gehören beispielsweise die Monosaccharide L-Arabinose, L-Rhamnose und D-Xylose, das Disaccharid D-Cellobiose, das Trisaccharid Raffinose sowie der Zuckeralkohol D-Mannitol.[1][6] Es erfolgt keine Bildung von Schwefelwasserstoff (H2S), hingegen verläuft die Äskulinspaltung positiv, Äskulin wird hydrolysiert. Das Enzym Urease ist nicht vorhanden, somit kann Harnstoff nicht abgebaut werden.[1] Zwar ist E. gergoviae Urease-positiv,[6] wird aber nicht mehr zur Gattung gezählt (siehe Abschnitt Systematik). In Bezug auf weitere Enzyme, die in biochemischen Testsystemen geprüft werden, ist Ornithindecarboxylase (ODC) vorhanden – ein wichtiges Kriterium zur Unterscheidung der ODC-negativen Klebsiellen[15] –, während die Enzyme Lysindecarboxylase (LDC) und Arginindihydrolase (ADH) nur bei einigen Arten vorkommen und somit zu deren Unterscheidung benutzt werden.[1][6]

Um die einzelnen Enterobacter-Arten zu identifizieren, eignen sich biochemische Tests, die auf dem Abbau verschiedener organischer Verbindungen beruhen und dabei gebildete Stoffwechselprodukte anzeigen, dafür können miniaturisierte Testsysteme verwendet werden. Bei den dafür zweckdienlichen Verbindungen handelt es sich beispielsweise um L-Fucose, D-Lyxose, D-Maltitol, D-Melibiose, Saccharose und D-Sorbitol.[6]
Weitere Nachweise
Eine serologische Unterscheidung verschiedener Stämme von E. cloacae ist möglich. Dabei werden Antikörper gegen die 53 somatischen O-Antigene und die 56 durch die Flagellen begründeten H-Antigene verwendet. So lassen sich knapp 80 verschiedene Serotypen unterscheiden. Das Verfahren findet aber noch keine Anwendung bei der epidemiologischen Untersuchung.[8][16]
Molekularbiologische Methoden sind gut geeignet zur Unterscheidung und damit zur Identifizierung verschiedener Enterobacter-Arten. Spezifisch ist der Nachweis bestimmter Teile des bakteriellen Genoms mit Hilfe des PCR-Verfahrens (Polymerase-Kettenreaktion). Dabei werden Genabschnitte, die typisch für die Bakterienart sind, vervielfältigt (amplifiziert) und nachgewiesen. Ein 2012 entwickeltes Verfahren beruht auf der Real Time Quantitative PCR (q-PCR) von einer als dnaJ bezeichneten Nukleotidsequenz. Dazu werden die Bakterien zunächst auf dem Selektivnährmedium Endo-Agar kultiviert und dann wird ihre DNA extrahiert. Ein zu dem Genabschnitt passendes Primer-Paar ermöglicht die Vervielfältigung und quantitative Bestimmung der vorhandenen Genabschnitte und somit eine Identifizierung des Bakteriums. Das in Deutschland entwickelte Verfahren zielt auf den Nachweis von E. cloacae ab, der damit von den anderen Arten des sogenannten E. cloacae Komplexes (bestehend aus E. asburiae, E. cloacae, E. hormaechei, E. kobei, E. ludwigii und E. nimipressuralis) unterschieden werden kann.[17] Weitere Abwandlungen des PCR-Verfahrens werden ebenfalls zur Identifizierung verwendet, beispielsweise mit zufällig vervielfältigter polymorpher DNA (RAPD), deren Variante AP-PCR (arbitrary primed PCR, PCR mit arbiträr – willkürlich – gewählten Primern) oder die repetitive sequenzbasierte PCR (rep-PCR). Ein ebenfalls auf molekularbiologischen Methoden basierendes Untersuchungsverfahren ist die Pulsed-Field-Gelelektrophorese (PFGE), welche bei epidemischen Ausbrüchen in Neugeborenen-Intensivstationen zur Aufklärung über die beteiligten Erreger verwendet wird.[12] PFGE wird ebenfalls zur Identifizierung von Cronobacter spp. benutzt.[18]
Die Identifizierung mit Hilfe der MALDI-TOF-Methode in Kombination mit Massenspektrometrie (MS) ist zwar geeignet, Enterobacter nachzuweisen, die Unterscheidung nah verwandter Arten (sogenannter E. cloacae Komplex) gelingt jedoch nicht zuverlässig.[17] Auch eine weitere Untersuchung zeigt, dass zwar die Unterscheidung von E. cloacae Komplex und E. aerogenes (mittlerweile Klebsiella aerogenes) mittels MALDI-TOF MS möglich ist, die Abgrenzung innerhalb des Komplexes aber nicht eindeutig ist.[19]
Systematik und Taxonomie
Beschreibungen von Bakterien, die sich auf Enterobacter-Arten zurückführen lassen, gibt es seit Ende des 19. Jahrhunderts („Bacillus cloacae“ Jordan 1890). Synonyme für Enterobacter sind „Cloaca“ Castellani & Chalmers 1919 sowie „Aerobacter“ Hormaeche & Edwards 1958,[20] diese Namen sind jedoch nicht mehr gültig.[21] Die Gattung Enterobacter ist nicht die Typusgattung der Familie Enterobacteriaceae, dies ist die Gattung Escherichia. Damit wird von der Regel 21a in Verbindung mit Regel 9 der Nomenklatur gemäß dem Bakteriologischen Code (International Code of Nomenclature of Bacteria) abgewichen, was 1958 durch Festlegung in der Judicial Opinion 15 der Judicial Commission (etwa „richterliche oder unparteiische Kommission“) der Internationalen Kommission für die Systematik der Prokaryoten (International Committee on Systematics of Prokaryotes, ICSP) festgesetzt wurde.[22] Typusart der Gattung ist Enterobacter cloacae (Jordan 1890) Hormaeche & Edwards 1960.
Äußere Systematik
Die Gattung Enterobacter zählt zu der Familie der Enterobacteriaceae in der Ordnung Enterobacterales Adeolu et al. 2016, die zur Klasse der Gammaproteobacteria gehört.[22] Zu der 2016 etablierten Ordnung der Enterobacterales gehören acht Familien mit insgesamt etwa 60 Gattungen. Die neu festgelegte und damit den Regeln des Bakteriologischen Codes (ICBN) entsprechende Typusgattung der Ordnung Enterobacterales Adeolu et al. 2016 ist die Gattung Enterobacter.[23][22] Die Enterobacteriaceae bilden eine große Gruppe gramnegativer Bakterien, zu denen u. a. die Gattungen Citrobacter, Escherichia, Klebsiella, Raoultella, Salmonella und Shigella gehören, von denen einige Vertreter als Krankheitserreger von Bedeutung sind.
Die Arbeit von Hormaeche und Edwards von 1960 basierte auf der Problematik, dass in der als „Aerobacter“ bezeichneten Gattung (mit den damals anerkannten Arten „Aerobacter aerogenes“ und „Aerobacter cloacae“) sowohl motile wie auch nicht-motile Bakterien eingeordnet wurden, wobei letztere der Gattung Klebsiella zuzuordnen wären, was durch serologische und biochemische Tests bereits damals bewiesen wurde. Die neue Bezeichnung als Enterobacter statt „Aerobacter“ und die Beschreibung der typischen Merkmale sollte dabei helfen, den in dem Bereich tätigen Wissenschaftlern eine eindeutige Klassifizierung neu beschriebener Arten zu ermöglichen.[1] Eine Konsequenz daraus ist die 2017 erfolgte Reklassifizierung von Enterobacter aerogenes als Klebsiella aerogenes.[10] Es war bereits 1971 erkannt worden, dass es sich bei E. aerogenes und K. mobilis um homotypische Synonyme handelt, da beide Arten den gleichen Typusstamm aufweisen.[24] Ebenfalls wurde in der Vergangenheit diskutiert, Enterobacter aufgrund phänotypischer Ähnlichkeiten mit Klebsiella und weiteren Gattungen zum Tribus der Klebsielleae zu zählen.[24] Die Rangstufe Tribus ist seit der Revision (1990) des International Code of Nomenclature of Bacteria (Bakteriologischer Code) nicht mehr üblich.
Weitere Untersuchungen zeigten, dass verschiedene Arten von Erwinia phänotypisch eher Arten der Gattung Enterobacter glichen, sie wurden daher in diese Gattung gestellt, beispielsweise Erwinia herbicola als Enterobacter agglomerans,[20] die aber seit 1989 zu einer eigenen Gattung Pantoea gehören.[25] Vor allem durch genetische Untersuchungen, z. B. DNA-DNA-Hybridisierung und Multi-Locus Sequenzanalyse (MLSA, hierbei werden nur bestimmte Gene untersucht) veränderte sich die Systematik der Enterobakterien grundlegend. So wurde bei zahlreichen Enterobacter-Arten festgestellt, dass sie zu anderen, neu beschriebenen Gattungen gehören. Die Arbeiten von Brady et al. 2013 führten so zu der Erstbeschreibung von Kosakonia, Lelliottia und Pluralibacter, sowie einer erweiterten Beschreibung der Gattungen Cronobacter und Enterobacter.[2]
Innere Systematik
Untersuchungen im Zeitraum von 2004 bis 2005 von Enterobacter dissolvens ergaben, dass es sich um eine Unterart (Subspezies) von E. cloacae handelt.[26] Im gleichen Zeitraum wurden drei medizinisch relevante Subspezies von E. hormaechei beschrieben,[27] die jedoch erst 2016 valide publiziert wurden.[22] Wegen der durchaus schwierigen Unterscheidung nah verwandter Arten wird in der medizinischen Mikrobiologie der Begriff des sogenannten E. cloacae Komplexes verwendet, bestehend aus E. asburiae, E. cloacae, E. hormaechei, E. kobei, E. ludwigii und E. nimipressuralis.[15][27] Andere Autoren verstehen darunter E. asburiae, E. cancerogenus, E. cloacae und E. ludwigii.[19]
Aktuell (Stand Dezember 2019) werden in der Gattung folgende Arten und Unterarten geführt,[22] E. cloacae ist die Typusart.[21] Zusätzlich werden in der Liste auch Bakterien aufgeführt, die früher der Gattung Enterobacter angehörten, nun aber zu anderen Gattungen gestellt wurden, dies ist durch einen Pfeil hinter dem Namen mit Verweis auf die aktuelle Bezeichnung kenntlich gemacht.
- Enterobacter aerogenes Hormaeche & Edwards 1960 (Synonym Klebsiella mobilis Bascomb et al. 1971) → Klebsiella aerogenes (Hormaeche & Edwards 1960) Tindall et al. 2017, comb. nov.[10]
- Enterobacter agglomerans Ewing & Fife 1972 → Pantoea agglomerans (Ewing & Fife 1972) Gavini et al. 1989, comb. nov.[25]
- Enterobacter amnigenus Izard et al. 1981 → Lelliottia amnigena (Izard et al. 1981) Brady et al. 2013, comb. nov.[2]
- Enterobacter arachidis Madhaiyan et al. 2010 → Kosakonia arachidis (Madhaiyan et al. 2010) Brady et al. 2013, comb. nov.[2]
- Enterobacter asburiae Brenner et al. 1988 emend. Hoffmann et al. 2005,[26] zuvor als “CDC Enteric group 17” bezeichnet[15]
- Enterobacter bugandensis Doijad et al. 2016, sp. nov.[4]
- Enterobacter cancerogenus (Urosevic 1966) Dickey & Zumoff 1988, comb. nov. (Synonym Erwinia cancerogena Urosevic 1966 (Approved Lists 1980)), zuvor als “CDC Enteric group 19” bezeichnet[15]
- Enterobacter cloacae (Jordan 1890) Hormaeche & Edwards 1960
- Enterobacter cloacae subsp. cloacae (Jordan 1890) Hoffmann et al. 2005 subsp. nov.
- Enterobacter cloacae subsp. dissolvens (Rosen 1922) Hoffmann et al. 2005 subsp. nov.
- Enterobacter cowanii Inoue et al. 2001 → Kosakonia cowanii (Inoue et al. 2001) Brady et al. 2013, comb. nov.[2]
- Enterobacter dissolvens (Rosen 1922) Brenner et al. 1988 → E. cloacae subsp. dissolvens (Rosen 1922) Hoffmann et al. 2005 subsp. nov.
- Enterobacter gergoviae Brenner et al. 1980 → Pluralibacter gergoviae (Brenner et al. 1980) Brady et al. 2013, comb. nov.[2]
- Enterobacter helveticus Stephan et al. 2007 → Cronobacter helveticus (Stephan et al. 2007) Brady et al. 2013, comb. nov.[2]
- Enterobacter hormaechei O'Hara et al. 1990[14] emend. Hoffmann et al. 2016, zuvor als “CDC Enteric group 75” bezeichnet[15]
- Enterobacter hormaechei subsp. hormaechei Hoffmann et al. 2016 subsp. nov.
- Enterobacter hormaechei subsp. oharae Hoffmann et al. 2016 subsp. nov.
- Enterobacter hormaechei subsp. steigerwaltii Hoffmann et al. 2016 subsp. nov.
- Enterobacter intermedius corrig. Izard et al. 1980, sp. nov. → Kluyvera intermedia (Izard et al. 1980) Pavan et al. 2005, comb. nov.
- Enterobacter kobei Kosako et al. 1988 emend. Hoffmann et al. 2005,[26] zuvor als “NIH group 21” bezeichnet[15]
- Enterobacter ludwigii Hoffmann et al. 2005 sp. nov.
- Enterobacter massiliensis Lagier et al. 2014 sp. nov.[28] → Metakosakonia massiliensis (Lagier et al. 2014) Alnajar & Gupta 2017, comb. nov.[29]
- Enterobacter mori Zhu et al. 2011 sp. nov.
- Enterobacter muelleri Kämpfer et al. 2015 sp. nov.
- Enterobacter nimipressuralis (Carter 1945) Brenner et al. 1988, comb. nov. → Lelliottia nimipressuralis (Carter 1945) Brady et al. 2013, comb. nov.[2]
- Enterobacter oryzae Peng et al. 2009, sp. nov. → Kosakonia oryzae (Peng et al. 2009) Brady et al. 2013, comb. nov.[2]
- Enterobacter oryzendophyticus Hardoim et al. 2015, sp. nov. → Kosakonia oryzendophytica (Hardoim et al. 2015) Li et al. 2016, comb. nov.
- Enterobacter oryziphilus Hardoim et al. 2015, sp. nov. → Kosakonia oryziphila (Hardoim et al. 2015) Li et al. 2016, comb. nov.
- Enterobacter pulveris Stephan et al. 2008, sp. nov. → Cronobacter pulveris (Stephan et al. 2008) Brady et al. 2013, comb. nov.[2]
- Enterobacter pyrinus Chung et al. 1993, sp. nov. → Pluralibacter pyrinus (Chung et al. 1993) Brady et al. 2013, comb. nov.[2]
- Enterobacter radicincitans Kämpfer et al. 2005, sp. nov. → Kosakonia radicincitans (Kämpfer et al. 2005) Brady et al. 2013, comb. nov.[2]
- Enterobacter sacchari Zhu et al. 2013, sp. nov. → Kosakonia sacchari (Zhu et al. 2013) Gu et al. 2014, comb. nov.
- Enterobacter sakazakii Farmer et al. 1980, sp. nov. → Cronobacter sakazakii (Farmer et al. 1980) Iversen et al. 2008, comb. nov.[11]
- Enterobacter siamensis Khunthongpan et al. 2014, sp. nov.
- Enterobacter soli Manter et al. 2011, sp. nov.
- Enterobacter tabaci Duan et al. 2016, sp. nov.
- Enterobacter taylorae Farmer et al. 1985, sp. nov. → E. cancerogenus (Urosevic 1966) Dickey & Zumoff 1988, comb. nov.
- Enterobacter turicensis Stephan et al. 2007, sp. nov. → Cronobacter zurichensis (Stephan et al. 2007) Brady et al. 2013, nom. nov.[2]
- Enterobacter xiangfangensis Gu et al. 2014, sp. nov.
Etymologie und Eponyme
Die Bezeichnung Enterobacter leitet sich von enteron (altgriechisch ἕντερον ‚Darm‘) und dem latinisierten Wort bacter für altgriechisch βακτηρΐα ‚Stab‘ ab, bedeutet folglich etwa „kleines Stäbchenbakterium im Darm“.[22] Die Idee zu dieser Bezeichnung stammt von dem deutsch-dänischen Bakteriologen Fritz Kauffmann.[1]
Für zahlreiche Enterobacter-Spezies wurde von den Wissenschaftlern, die sie erstmals beschrieben haben, ein Epitheton gewählt, das ein Eponym darstellt, also ein Wort, das aus einem Eigennamen abgeleitet ist. E. hormaechei wurde zu Ehren von Estenio Hormaeche (uruguayischer Mikrobiologe) benannt, er hat zusammen mit Philip R. Edwards die Gattung definiert.[14] Das Epitheton von E. ludwigii ist dem deutschen Mikrobiologen Wolfgang Ludwig gewidmet, der zur Verständlichkeit der Systematik der Bakterien beigetragen hat. Mary Alyce Fife-Asbury (US-amerikanische Mikrobiologin), Hans Emil Müller (deutscher Mikrobiologe) und Welton Taylor (US-amerikanischer Mikrobiologe) „standen Pate“ für E. asburiae, E. muelleri bzw. E. taylorae, die drei Wissenschaftler haben grundlegende Erkenntnisse über die Enterobacteriaceae erlangt und veröffentlicht.[22]
Aber auch die Personen, die mit der Verwendung eines Eponyms ihre Kollegen geehrt haben, wurden umgekehrt von anderen Wissenschaftlern in einem Epitheton einer Subspezies erwähnt: Caroline M. O'Hara und Arnold G. Steigerwalt, beides US-amerikanische Mikrobiologen, die E. hormaechei erstbeschrieben haben, findet man in E. hormaechei subsp. oharae und E. hormaechei subsp. steigerwaltii wieder.[22]
Vorkommen
Hormaeche und Edwards beschreiben in ihrem Artikel A proposed genus Enterobacter von 1960 das Vorkommen der damals bekannten zwei Enterobacter-Arten mit „ widely distributed in nature.“ (deutsch: ‚In der Natur weit verbreitet.‘).[1] Auch Jahrzehnte danach ist diese Aussage noch zutreffend, jedoch sollte bei Angaben zur Ökologie von Enterobacter beachtet werden, wie häufig sich die Systematik der Gattung bzw. verwandter Gattungen geändert hat.[9] So wurde beispielsweise ein mit Pflanzen assoziiertes Bakterium zunächst als Erwinia herbicola, dann als Enterobacter agglomerans und nun als Pantoea agglomerans bezeichnet.[22]
Natürliche Habitate der Vertreter der Gattung sind Gewässer, Abwasser, Brackwasser, Pflanzen und Erdboden. Mehrere Arten findet man auch auf der Haut und im Darm von Menschen und Tieren, in Fleisch, Rohmilch und im Krankenhaus.[9] In Tierställen treten sie als Bioaerosole in Größenordnungen von bis zu 104 koloniebildenden Einheiten pro Kubikmeter Luft auf.[30] Beim Fund in Lebensmitteln ist eine Kontamination im Herstellungsprozess zu vermuten, dies trifft beispielsweise auf Milcherzeugnisse wie Joghurt und Käse zu und kann auch bei pulverförmiger Säuglingsanfangsnahrung der Fall sein.[12] In medizinischen Proben wie Urin, Faeces, Blut, Sputum und Wunden wurden E. asburiae, E. cancerogenus, E. cloacae subsp. cloacae und E. hormaechei nachgewiesen. Hingegen wurde Enterobacter cloacae subsp. dissolvens nur in Umweltproben gefunden, erstmals wurde er 1922 von verrottenden Getreidehalmen isoliert.[9] E. soli wurde 2011 aus Erdboden isoliert und ist in der Lage, Lignin abzubauen.[22]
Einige der in den letzten Jahren entdeckten Mitglieder der Gattung (E. bugandensis, E. kobei, E. ludwigii und E. massiliensis) wurden in medizinischem Untersuchungsmaterial entdeckt, wobei es sich dabei nicht zwangsläufig um Krankheitserreger handelt. E. massiliensis wurde im Rahmen einer systematischen Untersuchung der Darmflora isoliert, aus einer Stuhlprobe eines gesunden jungen Mannes aus dem Senegal.[28] Mehrere Arten wurden neben ihrem Vorkommen in medizinischem Untersuchungsmaterial auch aus Umweltproben isoliert, E. cancerogenus von Bäumen, aus Wasser und Lebensmitteln, E. kobei aus Lebensmitteln und E. ludwigii aus Erdboden und Pflanzen.[15] Andere Enterobacter-Arten wurden von Pflanzen isoliert und sind nach jetzigem Kenntnisstand typisch für diese, z. B. E. mori aus den Wurzeln eines erkrankten Maulbeerbaums Morus alba L. (Weiße Maulbeere) oder E. muelleri aus der Rhizosphäre von Zea mays L. (Mais).[22]
Bedeutung
Biotechnologie
-Acetolactate_Structural_Formula_V1.svg.png.webp)
-Acetoin.svg.png.webp)
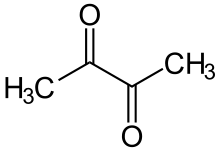
Bestimmte Enzyme von Enterobacter können zu einer biotechnologischen Nutzung des Bakteriums bzw. seiner Gene führen. So wird das Enzym α-Acetolactatdecarboxylase (Acetyllactatdecarboxylase) verwendet, das in der 2,3-Butandiol-Gärung die Decarboxylierung von Acetyllactat bewirkt (Acetyllactat wird auch als 2-Acetolactat bzw. α-Acetolactat bezeichnet, es handelt sich um das Anion der 2-Hydroxy-2-methyl-3-oxobuttersäure), wobei Acetoin entsteht. Andererseits kann Acetyllactat ohne Einwirkung von Enzymen der oxidativen Decarboxylierung unterliegen und dadurch in Diacetyl umgewandelt werden. Diacetyl ist eine organische Verbindung, die in geringer Konzentration einen ausgeprägten Geschmack und Geruch nach Butter aufweist und auch Bestandteil des natürlichen Butteraromas ist. Andererseits kann sie in Wein und Bier zu einem Fehlaroma führen. Diacetyl kann enzymatisch mit Hilfe der Diacetylreduktase zu Acetoin reduziert werden, was beim Prozess des Bierbrauens durch die Brauhefe Saccharomyces cerevisiae geschieht. Trotzdem können geringe Mengen an Diacetyl entstehen, die zu dem unerwünschten Fehlaroma führen. Daher wurde das Gen für die α-Acetolactatdecarboxylase aus Enterobacter durch Transformation in Saccharomyces cerevisiae übertragen. Durch die rekombinante Hefe konnte beim Brauprozess die Konzentration an Diacetyl in der Würze verringert werden, da nun direkt Acetyllactat in Acetoin umgewandelt wurde.[31]
Hingegen findet Diacetyl auch als Aromastoff Verwendung, so dass seine biotechnologische Produktion von Interesse ist. Dazu wurde 2015 in einem Stamm von E. cloacae subsp. dissolvens das Gen für das Enzym α-Acetolactatdecarboxylase durch Gen-Knockout „abgeschaltet“. Außerdem wurden die Gene für die Enzyme, die Diacetyl zu Acetoin reduzieren, inaktiviert. Die gentechnisch veränderten Bakterien produzierten daraufhin in einem Fermenter Diacetyl. Um die Ausbeute zu erhöhen, erwies sich der Zusatz von Eisen(III)-Ionen (Fe3+-Ionen) als hilfreich. Dadurch wurde die Diacetylkonzentration in der Fermenterbrühe auf 1,45 g/L gesteigert.[32]
Antibiotikaresistenzen
Eine Antibiotikaresistenz liegt vor, wenn ein bestimmtes Bakterium resistent („widerstandsfähig“) gegen ein bestimmtes Antibiotikum ist, durch dieses also nicht im Wachstum gehemmt wird. Andersherum ausgedrückt ist das Antibiotikum gegen das Bakterium nicht wirksam. Bei Bakterien wird manchmal zwischen einer natürlichen (engl. natural(ly)) und einer erworbenen (engl. acquired) Resistenz unterschieden.[31]
Die Vertreter der Gattung Enterobacter besitzen eine natürliche Resistenz gegen bestimmte β-Lactam-Antibiotika, da sie in ihrem Bakterienchromosom über einen induzierbaren Genabschnitt verfügen, dessen Genprodukt das Enzym Cephalosporinase ist. Cephalosporinasen sind eine Untergruppe der β-Lactamasen, die den β-Lactam-Ring dieser Antibiotikagruppe aufspalten und sie so unwirksam machen. Bakterienstämme des Enterobacter cloacae Komplexes (einschließlich E. asburiae und E. hormaechei) sind dadurch resistent gegen Aminopenicilline und Cephalosporine der 1. Generation. Gegen Carboxypenicilline hingegen sind sie sensitiv (empfindlich), bei den Cephalosporinen der 2. Generation sind einige und bei den Cephalosporinen der 3. Generation die meisten Antibiotika wirksam.[31] Das als ampC bezeichnete Gen codiert für das als AmpC-Beta-Lactamase (in diesem Fall eine Cephalosporinase) bezeichnete Enzym. Einer 2008 durchgeführten Studie zufolge ist es in allen untersuchten Enterobacter-Isolaten vorhanden. Durch eine Mutation kann es zu einer Überexpression der in dem Bakterienchromosom codierten AmpC-Beta-Lactamase kommen und dadurch bedingt zur Resistenz gegen Cephalosporine höherer Generationen und Cephamycine wie Cefoxitin, Cefotetan und Cefmetazol.[12]
Die Gene für eine erworbene Resistenz sind häufig auf einem Plasmid lokalisiert. Dies gilt beispielsweise für die plasmidcodierte Penicillinase oder die plasmidcodierte Extended Spectrum β-Lactamase (ESBL). Bakterienstämme des Enterobacter cloacae Komplexes sind durch eine erworbene Resistenz in der Lage, Carboxypenicilline (z. B. Carbenicillin) und Ureidopenicilline (Mezlocillin) sowie Cephalosporine der 3. Generation (z. B. Cefotaxim) zu inaktivieren.[31] Damit sind sie gegen zwei der vier im MRGN-System (multiresistente gramnegative Bakterien) definierten Antibiotikaklassen resistent. Eine Multiresistenz wird bei Enterobacter spp. eher durch eine Überexpression der chromosomal codierten AmpC verursacht, eine plasmidcodierte ESBL kommt daneben vor.[33] Laut eines Berichts des ECDC (Europäisches Zentrum für die Prävention und die Kontrolle von Krankheiten) wiesen im Jahr 2016 32 % der Enterobacter-Isolate eine Resistenz gegen Cephalosporine der 3. Generation auf, diese Daten stammen aus dem europäischen Surveillance-System TESSy.[34]
Zuletzt wurde auch über Vertreter der Gattung Enterobacter berichtet, die gegen Carbapeneme und somit gegen eine weitere der im MRGN-System definierten vier Antibiotikaklassen resistent sind. Die Resistenz gegenüber Carbapenemen ist noch eher selten und kann durch die zuvor erwähnte AmpC-Beta-Lactamase oder ESBL in Kombination mit einem Porinverlust verursacht werden. Aber auch Carbapenemase-produzierende Vertreter wurden bereits isoliert. In Deutschland war bei E. cloacae 2010 die VIM-1 (Verona-Integron-Metallo-β-Lactamase) die am häufigsten identifizierte Carbapenemase.[33] Nach dem Bericht des ECDC wiesen 2,6 % der Enterobacter spp. Isolate eine Resistenz gegen Carbapeneme auf, untersucht wurden 1.435 Isolate aus im Rahmen von TESSy im Jahr 2016 erhobenen Daten aus 14 Staaten.[34] Der ebenfalls vom ECDC 2013 veröffentlichte Zwischenbericht über European survey on carbapenemase-producing Enterobacteriaceae (EuSCAPE) lieferte Daten aus 38 Staaten, demnach sind in Estland und Litauen Enterobacter spp. die bei den Enterobacteriaceae dominierenden Arten in Bezug auf Carbapenemase-Produktion. Die meisten Staaten (33) gaben hingegen Klebsiella pneumoniae als wichtigsten Vertreter an.[35] Die nach dieser Bakterienart benannten Carbapenemasen (KPC-1, KPC-2 usw.) sind jedoch nicht auf Klebsiella-Arten beschränkt, sondern können – da sie plasmidcodiert sind – durch horizontalen Gentransfer zwischen verschiedenen gramnegativen Bakterienarten ausgetauscht werden.[33] 2004 wurde erstmals über Enterobacter-Isolate mit einer plasmidcodierten KPC-2 β-Lactamase (in diesem Fall eine Carbapenemase) berichtet. Der Patient, von dem die Isolate stammten, wurde 2001 in einem Krankenhaus in Boston behandelt und litt an Sepsis, die durch verschiedene Bakterien verursacht worden war. Die zunächst nicht näher spezifizierten Enterobacter-Isolate wurden mit verschiedenen Untersuchungsverfahren als E. cloacae oder E. asburiae identifiziert, wobei eine exakte Zuordnung nicht möglich war.[36]
Während 1970 noch alle E. cloacae Stämme empfindlich gegen Gentamicin waren, ein Aminoglycosidantibiotikum, änderte sich das im Verlauf der 1970er Jahre rapide. Ursache dafür sind die auf einem Plasmid lokalisierten AME-Gene, die für die Aminoglycosid-modifizierenden Enzyme codieren, die die Struktur dieser Antibiotikagruppe so verändern, dass sie nicht mehr wirksam sind. Neben Gentamicin sind u. a. Tobramycin und Amikacin betroffen.[31]
Die Antibiotikaresistenz wird im Labor durch ein Antibiogramm ermittelt. Dabei kommt häufig die Kirby-Bauer-Methode zum Einsatz, bei der in einem Agardiffusionstest auf Müller-Hinton-Agar mit einer verteilten Suspension des Bakteriums kleine kreisförmige Filterplättchen gelegt werden, die verschiedene Antibiotika in definierter Menge enthalten. Mehrere Erstbeschreibungen von Enterobacter-Arten enthalten die Ergebnisse dieser Antibiogramme, beispielsweise E. bugandensis,[4] E. hormaechei[14] und E. massiliensis.[28]
Auch das PCR-Verfahren kann verwendet werden. Es zielt auf die Gene ab, die für die Antibiotikaresistenzen der Bakterien verantwortlich sind, da durch sie die Enzyme codiert sind, die die Antibiotika abbauen oder verändern und damit unwirksam machen. Im Jahr 2015 wurden mittels PCR 77 Enterobacter-Isolate untersucht, die an nosokomialen Infektionen („Krankenhausinfektionen“) in Algerien (27 Proben) und Frankreich (50 Proben) beteiligt waren und von denen angenommen wurde, dass es sich um E. cloacae handelt. Das PCR-Verfahren ergab, dass 29 Isolate mindestens ein ESBL-codierendes Gen und 28 Isolate AME-Gene (Aminoglycosid-modifizierende Enzyme) aufwiesen. Die relative und absolute Häufigkeit positiver Befunde war bei den aus Algerien stammenden Proben (18 bzw. 20) größer als bei den aus Frankreich stammenden (11 bzw. 8).[19]
Medizinische Bedeutung
Die humanmedizinische Relevanz der in der Gattung verbliebenen Arten ist noch Gegenstand der Forschung. Man nimmt an, dass sie neben den im Abschnitt Pathogenität erwähnten Fällen von Infektionskrankheiten, die durch Enterobacter-Arten verursacht wurden, auch für Infektionskrankheiten der unteren Atemwege, Lungenentzündungen (Pneumonien), Infektionskrankheiten der Haut und des Weichgewebes, ophthalmische (das Auge betreffende) Infektionskrankheiten, Endokarditis sowie septische Arthritis verantwortlich sein können. Bei den meisten Fällen handelt es sich um im Krankenhaus erworbene (nosokomiale) Infektionen.[12] Arten, die aus medizinischen Proben im Krankenhaus oder im Zusammenhang mit nosokomialen Infektionskrankheiten isoliert wurden, werden als opportunistische Krankheitserreger angesehen.[4] Problematisch ist die verbreitete Resistenz gegen mehrere Antibiotika, so dass sie als Erreger nosokomialer Infektionskrankheiten von zunehmender Bedeutung sind.[5][15][33] Hingegen spielen Enterobacter-Arten, anders als Cronobacter sakazakii, bei Lebensmittelinfektionen epidemiologisch keine Rolle.[12]
Von Infektionen, die im Krankenhaus erfolgen, sind vor allem immunsupprimierte Patienten betroffen. Weiterhin sind Neugeborene gefährdet, bei denen insbesondere die Kriterien Frühgeburt, geringes Geburtsgewicht, Anwendung invasiver medizinischer Verfahren und zu häufige Verwendung von Antibiotika Risikofaktoren sind. Mit den neonatalen Infektionskrankheiten werden E. cloacae und E. hormaechei in Verbindung gebracht, neben den nicht mehr zur Gattung zählenden C. sakazakii (Nekrotisierende Enterokolitis bei Cronobacter-Infektionen[37]), K. aerogenes und Pluralibacter gergoviae. Dabei ist anzumerken, dass es in der Vergangenheit häufiger zu falschen Ergebnissen bezüglich der Unterscheidung von E. hormaechei und Cronobacter-Arten gekommen ist.[12][15] Von Pneumonien sind v. a. intensivmedizinisch beatmete Patienten betroffen.[16] Ein Bericht des ECDC nennt für das Jahr 2016 bei 10 % der auf einer Intensivstation erworbenen Lungenentzündungen Enterobacter-Arten als Ursache (zum Vergleich: Pseudomonas aeruginosa 21 %, Staphylococcus aureus 18 %, Klebsiella spp. 16 % und Escherichia coli 13 %).[34]
Bei Infektionen mit Enterobacter-Arten sollte zunächst ein Antibiogramm zur Abklärung der Resistenzen durchgeführt werden. Häufig werden Ureidopenicilline und Cephalosporine der 3. Generation (z. B. Cefotaxim und Ceftazidim) verwendet, die jedoch bei multiresistenten Arten, die über eine plasmidcodierte Penicillinase oder die plasmidcodierte Extended Spectrum β-Lactamase verfügen, nicht wirksam sind. Weitere verwendete Antibiotika sind Carbapeneme, Chinolone und Aminoglycoside.[16] Bei Enterobacter-Arten, die durch die chromosomal codierte AmpC-Beta-Lactamase (in diesem Fall eine Cephalosporinase) resistent sind, werden Carbapeneme empfohlen,[38] dabei ist zu beachten, dass auch Resistenzen gegen diese Antibiotikagruppe auftreten können.[33]
Die meisten Daten zur medizinischen Bedeutung finden sich zur Typusart E. cloacae. 1966 wurde berichtet, dass sie Patienten im Krankenhaus eher zufällig kolonisiert, als dass sie für eine Infektion verantwortlich ist. In den 1970er Jahren nahm man an, dass ein bestimmter E. cloacae-Stamm endemisch in einem bestimmten Krankenhaus ist und bei immunsupprimierten Patienten als opportunistischer Erreger Infektionskrankheiten verursacht. Hygieneuntersuchungen zeigten, dass der Stamm über kontaminierte Hände des Pflegepersonals verbreitet wird und es über medizinische Geräte und Flüssigkeiten (z. B. das bei der Hydrotherapie verwendete Wasser, aber auch Desinfektionsmittel mit Benzalkoniumchlorid) zu Kreuzkontaminationen kommt. Nach einer Statistik der Centers for Disease Control and Prevention (CDC) war E. cloacae 1975 für 4,6 %, 1984 hingegen für 5,9 % der nosokomialen Infektionskrankheiten in US-amerikanischen Krankenhäusern verantwortlich.[9]
Überwachungsprogramme
Um epidemiologisch nutzbare Informationen über Infektionskrankheiten und die daran beteiligten Krankheitserreger zu sammeln, gibt es verschiedene Überwachungsprogramme (sogenannte Surveillances). Das Robert Koch-Institut (RKI) hat 2015 eine Übersicht der Surveillance-Systeme für Erreger und Resistenz für Deutschland herausgegeben. Dort ist die Surveillance der Antibiotika‐Anwendung und der bakteriellen Resistenzen auf Intensivstationen (SARI) aufgeführt, die vom Nationalen Referenzzentrum für Surveillance von nosokomialen Infektionen am Institut für Hygiene und Umweltmedizin, Charité Berlin und dem Institut für Umweltmedizin und Krankenhaushygiene der Universität Freiburg organisiert wird. Im SARI-Programm werden Proben auf 13 häufige Erreger und ihre Antibiotikaresistenz untersucht, unter den aufgeführten Bakterien ist auch E. cloacae zu finden. Die Teilnahme der Krankenhäuser an dieser Surveillance ist freiwillig.[39] Hingegen sind die Vorgaben des Infektionsschutzgesetzes (IfSG) verpflichtend. So ist in § 23 IfSG festgelegt, dass nosokomiale Infektionskrankheiten und Krankheitserreger mit speziellen Resistenzen und Multiresistenzen zu erfassen sind. Die näheren Einzelheiten werden durch die beim Robert Koch-Institut eingerichtete Kommission Antiinfektiva, Resistenz und Therapie festgelegt und in einer Liste im Bundesgesundheitsblatt veröffentlicht. Dort ist unter den aufgeführten Bakterien ebenfalls E. cloacae zu finden, der zu erfassen ist, sofern eine Einzelresistenz gegen Imipenem oder Meropenem beobachtet wurde oder eine Multiresistenz gemäß der KRINKO-Definition für 3MRGN bzw. 4MRGN.[40] Die Abkürzung KRINKO steht für die ebenfalls beim RKI eingerichtete Kommission für Krankenhaushygiene und Infektionsprävention.
Auch international wurden Überwachungssysteme eingerichtet, beispielsweise das European survey on carbapenemase-producing Enterobacteriaceae (EuSCAPE). An diesem Surveillance-System sind die 28 Mitgliedstaaten der Europäischen Union, Island, Norwegen, sieben (potenzielle) Beitrittskandidaten und Israel beteiligt. Ein 2013 veröffentlichter Zwischenbericht gibt an, dass 28 dieser 38 Staaten ein nationales Surveillance-System für Carbapenemase-produzierende Enterobacteriaceae (CPE) unterhalten, davon wird in 24 nationalen Programmen auch Enterobacter spp. überwacht.[35] Ein weiteres europäisches Surveillance-System wird als TESSy (The European Surveillance System) bezeichnet und liefert Daten zu nosokomialen Infektionen, die als Surveillance-Berichte durch das ECDC jährlich veröffentlicht werden und ebenfalls Enterobacter-Arten berücksichtigen.[34]
Bei Angaben zum Auftreten von durch Enterobacter-Arten verursachte Infektionskrankheiten ist die diagnostisch schwierige Abgrenzung der Arten untereinander sowie zu den verwandten Gattungen wie Klebsiella und Cronobacter zu beachten. Nach einem Bericht des deutschen Krankenhaus-Infektions-Surveillance-Systems (KISS) waren Enterobacter-Arten 2011 für 6,5 % aller nosokomialen Infektionskrankheiten auf Intensivstationen verantwortlich.[33] Daten aus Surveillance-Programmen bzw. aus Fallstudien bei Ausbrüchen von Infektionskrankheiten auf Intensivstationen liegen für Nord- und Südamerika, Europa und Asien vor.[41]
Quellen
Literatur
- Francine Grimont, Patrick A. D. Grimont: The Genus Enterobacter (Chapter 3.3.11). In: Martin Dworkin, Stanley Falkow, Eugene Rosenberg, Karl-Heinz Schleifer, Erko Stackebrandt (Hrsg.): The Prokaryotes. A Handbook on the Biology of Bacteria, Volume 6: Proteobacteria: Gamma Subclass. 3. Auflage. Springer-Verlag, New York 2006, ISBN 978-0-387-25496-8, S. 197–214, doi:10.1007/0-387-30746-X_9.
- Estenio Hormaeche, Philip R. Edwards: A proposed genus Enterobacter. In: International Bulletin of Bacteriological Nomenclature and Taxonomy. Band 10, Nr. 2, April 1960, S. 71–74, doi:10.1099/0096266X-10-2-71.
Einzelnachweise
- E. Hormaeche, P. R. Edwards: A proposed genus Enterobacter. In: International Bulletin of Bacteriological Nomenclature and Taxonomy. Band 10, Nr. 2, April 1960, S. 71–74, doi:10.1099/0096266X-10-2-71.
- Carrie Brady, Ilse Cleenwerck, Stephanus Venter, Teresa Coutinho, Paul De Vos: Taxonomic evaluation of the genus Enterobacter based on multilocus sequence analysis (MLSA): Proposal to reclassify E. nimipressuralis and E. amnigenus into Lelliottia gen. nov. as Lelliottia nimipressuralis comb. nov. and Lelliottia amnigena comb. nov., respectively, E. gergoviae and E. pyrinus into Pluralibacter gen. nov. as Pluralibacter gergoviae comb. nov. and Pluralibacter pyrinus comb. nov., respectively, E. cowanii, E. radicincitans, E. oryzae and E. arachidis into Kosakonia gen. nov. as Kosakonia cowanii comb. nov., Kosakonia radicincitans comb. nov., Kosakonia oryzae comb. nov. and Kosakonia arachidis comb. nov., respectively, and E. turicensis, E. helveticus and E. pulveris into Cronobacter as Cronobacter zurichensis nom. nov., Cronobacter helveticus comb. nov. and Cronobacter pulveris comb. nov., respectively, and emended description of the genera Enterobacter and Cronobacter. In: Systematic and Applied Microbiology. Band 36, Nr. 5, Juli 2013, S. 309–319, doi:10.1016/j.syapm.2013.03.005.
- S. D. Salas, G. G. Geesey: Surface attachment of a sediment isolate of Enterobacter cloacae. In: Microbial Ecology. Band 9, Nr. 4, Dezember 1983, S. 307–315, doi:10.1007/BF02019020.
- Swapnil Doijad, Can Imirzalioglu, Yancheng Yao, Niladri Bhusan Pati, Linda Falgenhauer, Torsten Hain, Bärbel U. Foesel, Birte Abt, Jörg Overmann, Mariam M. Mirambo, Stephen E. Mshana, Trinad Chakraborty: Enterobacter bugandensis sp. nov., isolated from neonatal blood. In: International Journal of Systematic and Evolutionary Microbiology. Band 66, Februar 2016, S. 968–974, doi:10.1099/ijsem.0.000821.
- Herbert Hof, Rüdiger Dörries: Duale Reihe: Medizinische Mikrobiologie. 3. Auflage. Thieme Verlag, Stuttgart 2005, ISBN 978-3-13-125313-2, S. 397–398.
- F. Grimont, P. A. D. Grimont: The Genus Enterobacter. Isolation, Identification. In: The Prokaryotes. A Handbook on the Biology of Bacteria, Volume 6. 2006, S. 205–208.
- Michael T. Madigan, John M. Martinko, Jack Parker: Brock Mikrobiologie. Deutsche Übersetzung herausgegeben von Werner Goebel, 1. Auflage. Spektrum Akademischer Verlag GmbH, Heidelberg/Berlin 2000, ISBN 3-8274-0566-1, S. 531–536.
- F. Grimont, P. A. D. Grimont: The Genus Enterobacter. Serotyping. In: The Prokaryotes. A Handbook on the Biology of Bacteria, Volume 6. 2006, S. 209–210.
- F. Grimont, P. A. D. Grimont: The Genus Enterobacter. Ecology and Epidemiology. In: The Prokaryotes. A Handbook on the Biology of Bacteria, Volume 6. 2006, S. 200–202.
- B. J. Tindall, G. Sutton, G. M. Garrity: Enterobacter aerogenes Hormaeche and Edwards 1960 (Approved Lists 1980) and Klebsiella mobilis Bascomb et al. 1971 (Approved Lists 1980) share the same nomenclatural type (ATCC 13048) on the Approved Lists and are homotypic synonyms, with consequences for the name Klebsiella mobilis Bascomb et al. 1971 (Approved Lists 1980). In: International Journal of Systematic and Evolutionary Microbiology. Band 67, Februar 2017, S. 502–504, doi:10.1099/ijsem.0.001572.
- Carol Iversen, Niall Mullane, Barbara McCardell, Ben D. Tall, Angelika Lehner, Séamus Fanning, Roger Stephan, Han Joosten: Cronobacter gen. nov., a new genus to accommodate the biogroups of Enterobacter sakazakii, and proposal of Cronobacter sakazakii gen. nov., comb. nov., Cronobacter malonaticus sp. nov., Cronobacter turicensis sp. nov., Cronobacter muytjensii sp. nov., Cronobacter dublinensis sp. nov., Cronobacter genomospecies 1, and of three subspecies, Cronobacter dublinensis subsp. dublinensis subsp. nov., Cronobacter dublinensis subsp. lausannensis subsp. nov. and Cronobacter dublinensis subsp. lactaridi subsp. nov.. In: International Journal of Systematic and Evolutionary Microbiology. Band 58, Juni 2008, S. 1442–1447, doi:10.1099/ijs.0.65577-0.
- S. Cooney, S. O’Brien, C. Iversen, S. Fanning: Bacteria: Other Pathogenic Enterobacteriaceae – Enterobacter and Other Genera. In: Yamine Motarjemi, Gerald Moy, Ewen Todd (Hrsg.): Encyclopedia of Food Safety. 1. Auflage. Academic Press, San Diego, CA 2014, ISBN 978-0-12-378612-8, S. 433–436.
- TRBA (Technische Regeln für Biologische Arbeitsstoffe) 466: Einstufung von Prokaryonten (Bacteria und Archaea) in Risikogruppen. In: Webseite der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin (BAuA). 25. August 2015, S. 148–149, abgerufen am 26. Dezember 2019 (letzte Änderung vom 14. August 2019).
- C. M. O'Hara, A. G. Steigerwalt, B. C. Hill, J. J. Farmer III, G. R. Fanning, D. J. Brenner: Enterobacter hormaechei, a new species of the family Enterobacteriaceae formerly known as enteric group 75. In: Journal of Clinical Microbiology. Band 27, Nr. 9, September 1989, S. 2046–2049, PMID 2778068, PMC 267735 (freier Volltext).
- Carol Iversen: Enterobacter. In: Carl A. Batt, Mary-Lou Tortorello (Hrsg.): Encyclopedia of Food Microbiology. 2. Auflage. Academic Press, San Diego, CA 2014, ISBN 978-0-12-384730-0, S. 653–658.
- Uwe Ullmann: Enterobacter. In: Gholamreza Darai, Michaela Handermann, Hans-Günther Sonntag, L. Zöller (Hrsg.): Lexikon der Infektionskrankheiten des Menschen: Erreger, Symptome, Diagnose, Therapie und Prophylaxe. 4. Auflage. Springer-Verlag, Berlin 2012, ISBN 978-3-642-17157-4, S. 274–275.
- Melanie Pavlovic, Regina Konrad, Azuka N. Iwobi, Andreas Sing, Ulrich Busch, Ingrid Huber: A dual approach employing MALDI-TOF MS and real-time PCR for fast species identification within the Enterobacter cloacae complex. In: FEMS Microbiology Letters. Band 328, Nr. 1, März 2012, S. 46–53, PMID 22150997, doi:10.1111/j.1574-6968.2011.02479.x.
- I. Proudy, D. Bouglé, E. Coton, M. Coton, R. Leclercq, M. Vergnaud: Genotypic characterization of Enterobacter sakazakii isolates by PFGE, BOX-PCR and sequencing of the fliC gene. In: Journal of Applied Microbiology. Band 104, Nr. 1, Januar 2008, S. 26–34, doi:10.1111/j.1365-2672.2007.03526.x, PMID 17850301.
- Nour Chems el Houda Khennouchi, Lotfi Loucif, Nafissa Boutefnouchet, Hamoudi Allag, Jean-Marc Rolain: MALDI-TOF MS as a Tool To Detect a Nosocomial Outbreak of Extended-Spectrum-β-Lactamase- and ArmA Methyltransferase-Producing Enterobacter cloacae Clinical Isolates in Algeria. In: Antimicrobial Agents and Chemotherapy. Band 59, Nr. 10, Oktober 2015, S. 6477–6483, doi:10.1128/AAC.00615-15, PMID 26239991, PMC 4576089 (freier Volltext).
- F. Grimont, P. A. D. Grimont: The Genus Enterobacter. Introduction. In: The Prokaryotes. A Handbook on the Biology of Bacteria, Volume 6. 2006, S. 197–200.
- Approved Lists of Bacterial Names. In: V. B. D. Skerman, Vicki McGowan, P. H. A. Sneath (Hrsg.): International Journal of Systematic Bacteriology. Band 30, Nr. 1, 1980, S. 362, doi:10.1099/00207713-30-1-225.
- Jean Euzéby, Aidan C. Parte: Genus Enterobacter. In: List of Prokaryotic names with Standing in Nomenclature (LPSN). Abgerufen am 27. Dezember 2019.
- M. Adeolu, S. Alnajar, S. Naushad, R. S. Gupta: Genome-based phylogeny and taxonomy of the ‘Enterobacteriales’: proposal for Enterobacterales ord. nov. divided into the families Enterobacteriaceae, Erwiniaceae fam. nov., Pectobacteriaceae fam. nov., Yersiniaceae fam. nov., Hafniaceae fam. nov., Morganellaceae fam. nov., and Budviciaceae fam. nov. In: International Journal of Systematic and Evolutionary Microbiology. Band 66, Dezember 2016, S. 5575–5599, doi:10.1099/ijsem.0.001485.
- S. Bascomb u. a.: Numerical Classification of the Tribe Klebsielleae. In: Journal of General Microbiology. Band 66, Nr. 3, Juni 1971, S. 279–295, doi:10.1099/00221287-66-3-279.
- Francoise Gavini, Joris Mergaert, Amor Beji, Christine Mielcarek, Daniel Izard, Karel Kersters, Jozef De Ley: Transfer of Enterobacter agglomerans (Beijerinck 1888) Ewing and Fife 1972 to Pantoea gen. nov. as Pantoea agglomerans comb. nov. and Description of Pantoea dispersa sp. nov.. In: International Journal of Systematic Bacteriology. Band 39, Nr. 3, Juli 1989, S. 337–345, doi:10.1099/00207713-39-3-337.
- Harald Hoffmann, Sibylle Stindl, Wolfgang Ludwig, Anita Stumpf, André Mehlen, Jürgen Heesemann, Daniel Monget, Karl H. Schleifer, Andreas Roggenkamp: Reassignment of Enterobacter dissolvens to Enterobacter cloacae as E. cloacae subspecies dissolvens comb. nov. and emended description of Enterobacter asburiae and Enterobacter kobei. In: Systematic and Applied Microbiology. Band 28, Nr. 3, April 2005, S. 196–205, doi:10.1016/j.syapm.2004.12.010.
- Harald Hoffmann, Sibylle Stindl, Wolfgang Ludwig, Anita Stumpf, André Mehlen, Daniel Monget, D. Pierard, S. Ziesing, Jürgen Heesemann, Andreas Roggenkamp, Karl H. Schleifer: Enterobacter hormaechei subsp. oharae subsp. nov., E. hormaechei subsp. hormaechei comb. nov., and E. hormaechei subsp. steigerwaltii subsp. nov., Three New Subspecies of Clinical Importance. In: Journal of Clinical Microbiology. Band 43, Nr. 7, Juli 2005, S. 3297–3303, doi:10.1128/JCM.43.7.3297-3303.2005.
- Jean-Christophe Lagier, Khalid El Karkouri, Ajay Kumar Mishra, Catherine Robert, Didier Raoult, Pierre-Edouard Fournier: Non contiguous-finished genome sequence and description of Enterobacter massiliensis sp. nov. In: Standards in Genomic Sciences. Band 7, Nr. 3, Februar 2013, S. 399–412, doi:10.4056/sigs.3396830.
- Seema Alnajar, Radhey S. Gupta: Phylogenomics and comparative genomic studies delineate six main clades within the family Enterobacteriaceae and support the reclassification of several polyphyletic members of the family. In: Infection, Genetics and Evolution. Band 54, Oktober 2017, S. 108–127, doi:10.1016/j.meegid.2017.06.024.
- VDI 4255 Blatt 2:2009-12 Bioaerosole und biologische Agenzien; Emissionsquellen und -minderungsmaßnahmen in der landwirtschaftlichen Nutztierhaltung; Übersicht (Bioaerosols and biological agents; Emission sources and control measures in livestock operations; Overview). Beuth Verlag, Berlin. S. 11.
- F. Grimont, P. A. D. Grimont: The Genus Enterobacter. Properties Relevant to Pathogenicity for Humans, Biotechnology. In: The Prokaryotes. A Handbook on the Biology of Bacteria, Volume 6. 2006, S. 203–204.
- L. Zhang, Y. Zhang, Q. Liu, L. Meng, M. Hu, M. Lv, K. Li, C. Gao, P. Xu, C. Ma: Production of diacetyl by metabolically engineered Enterobacter cloacae. In: Scientific Reports. Band 5, März 2015, S. 9033, doi:10.1038/srep09033, PMID 25761989, PMC 4357014 (freier Volltext).
- Empfehlung der Kommission für Krankenhaushygiene und Infektionsprävention (KRINKO) beim Robert Koch-Institut (RKI): Hygienemaßnahmen bei Infektionen oder Besiedlung mit multiresistenten gramnegativen Stäbchen. In: Bundesgesundheitsblatt - Gesundheitsforschung - Gesundheitsschutz. Band 55, 2012, S. 1311–1354, doi:10.1007/s00103-012-1549-5.
- Healthcare-associated infections acquired in intensive care units - Annual Epidemiological Report for 2016. (PDF; 962 KB) ECDC Surveillance Report. Europäisches Zentrum für die Prävention und die Kontrolle von Krankheiten (ECDC), Mai 2018, S. 1–11, abgerufen am 24. Mai 2018 (englisch).
- Hajo Grundmann, Corinna Glasner, Anna-Pelagia Magiorakos, Liselotte Högberg-Diaz, Dominique L. Monnet, Barbara Albiger: Carbapenemase-producing bacteria in Europe. (PDF; 3,3 MB) ECDC Technical Report. Europäisches Zentrum für die Prävention und die Kontrolle von Krankheiten (ECDC), November 2013, S. 1–18, abgerufen am 24. Mai 2018 (englisch).
- Ashfaque Hossain, M. J. Ferraro u. a.: Plasmid-Mediated Carbapenem-Hydrolyzing Enzyme KPC-2 in an Enterobacter sp. In: Antimicrobial Agents and Chemotherapy. Band 48, Nr. 11, November 2004, S. 4438–4440, doi:10.1128/AAC.48.11.4438-4440.2004, PMID 15504876, PMC 525415 (freier Volltext).
- Marianne Abele-Horn: Antimikrobielle Therapie. Entscheidungshilfen zur Behandlung und Prophylaxe von Infektionskrankheiten. Unter Mitarbeit von Werner Heinz, Hartwig Klinker, Johann Schurz und August Stich, 2., überarbeitete und erweiterte Auflage. Peter Wiehl, Marburg 2009, ISBN 978-3-927219-14-4, S. 262.
- Patrick N. A. Harris, Jane Y. Wei u. a.: Carbapenems versus alternative antibiotics for the treatment of bloodstream infections caused by Enterobacter, Citrobacter or Serratia species: a systematic review with meta-analysis. In: Journal of Antimicrobial Chemotherapy. Band 71, Nr. 2, Februar 2016, S. 296–306, doi:10.1093/jac/dkv346, PMID 26542304 (Review).
- Robert Koch-Institut (Hrsg.): Übersicht der Surveillance-Systeme für Erreger und Resistenz. 29. Januar 2015, S. 1–3 (Online + PDF, 141 KB [abgerufen am 30. März 2018]).
- Bekanntmachung des Robert Koch-Institutes: Surveillance nosokomialer Infektionen sowie die Erfassung von Krankheitserregern mit speziellen Resistenzen und Multiresistenzen. In: Bundesgesundheitsblatt - Gesundheitsforschung - Gesundheitsschutz. Band 56, 2013, S. 580–583, doi:10.1007/s00103-013-1705-6.
- Susan L. Fraser, Christian P. Sinave: Enterobacter Infections. In: Medscape. 5. September 2017, abgerufen am 24. Mai 2018.