DNA-Extraktion
Eine DNA-Extraktion beschreibt die Extraktion von DNA aus Zellen. Die DNA-Extraktion ist eine der Methoden zur DNA-Reinigung und vermutlich die am häufigsten verwendete.
Zellaufschluss
Die DNA eines Organismus kann auf mehreren Wegen isoliert werden. Die meisten Verfahren beginnen mit einer Konzentration der Zellen durch Zentrifugation und einem für die jeweilige Gruppe geeigneten Zellaufschluss des Zell-Niederschlags. So müssen z. B. bei pflanzlichen, pilzlichen oder bakteriellen Zellen, welche im Vergleich zu tierischen Zellen, Mycoplasmen und einigen Archaeenarten zusätzlich zur Zellmembran eine Zellwand besitzen, meist zusätzliche enzymatische (z. B. Lysozym bei Bakterien oder Proteinase K zur Proteolyse) oder mechanische Zerkleinerungsschritte (Standmixer) erfolgen. Bei der Plasmidpräparation aus Bakterien wird oftmals als chemischer Zellaufschluss die alkalische Lyse verwendet.[1]
Nach einem Zellaufschluss folgt meist eine Klärung des Homogenates durch Filtration oder Zentrifugation. DNA aus Mitochondrien oder Chloroplasten können durch eine Zellfraktionierung von der DNA des Zellkerns getrennt werden. Für eine Isolierung extrachromosomaler DNA wie beispielsweise virale DNA wird die Hirt-Extraktion verwendet.[2] Zur Entfernung der RNA kann ein RNase-Verdau durchgeführt werden.
Extraktionen
DNA ist ein polares Biopolymer mit relativ hoher molarer Masse, weshalb sie in unpolarer Umgebung (z. B. in organischen Lösungsmitteln) durch die verkleinerte Hydrathülle und die daraus folgende Absenkung ihrer Löslichkeit ausfällt. Daneben ist DNA in sauren, wässrigen Lösungen unlöslich aufgrund des Desoxyribosephosphat-Rückgrates mit negativen Ladungen proportional zur Kettenlänge, denn bei niedrigen pH-Werten sind die Phosphatgruppen und somit die negativen Ladungen der DNA mit Protonen abgesättigt, wodurch die Hydrathülle sich ebenfalls verkleinert.
Die meisten DNA-Extraktionen basieren nach dem Zellaufschluss auf zwei unterschiedlichen Verfahren, der Zwei-Phasen-Extraktion oder der Fällung, letztere eventuell mit zusätzlicher selektiver Adsorption an eine DNA-bindende Matrix. Die Extraktionsverfahren werden zum Teil auch miteinander kombiniert. Bei den folgenden Methoden folgt meistens eine abschließende Ethanolfällung. Es existieren verschiedene Kits zur DNA-Extraktion, mit unterschiedlichen Ausbeuten.[3] Daneben gibt es verschiedene Varianten der Methoden je nach Ursprung der DNA, beispielsweise für pflanzliche,[4][5][6][7] pilzliche[8][9] oder bakterielle DNA.[10][11][12][13]
Zwei-Phasen-Extraktion
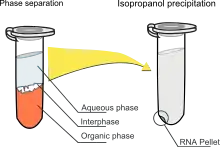
Die Zwei-Phasen-Extraktion basiert auf der unterschiedlichen Verteilung von Biomolekülen in einer organischen Phase, einer wässrigen Phase und der dazwischenliegenden Interphase. Hierzu zählen z. B. die Phenol-Chloroform-Extraktion, die Phenol-Chloroform-Isoamylalkohol-Extraktion (im Volumenverhältnis 25:24:1)[14] und die Trizol-Extraktion.[15] Die DNA sammelt sich bei der Extraktion nach Sacchi (mit Trizol, TRI Reagent, Trisure, STAT-60, RNAzol oder DNAzol[16] als Handelsnamen und Varianten) in der Interphase, die RNA und die Kohlenhydrate in der wässrigen Phase und die Proteine und die Lipide in der organischen Phase. Dabei sorgt das Chaotrop Guanidiniumthiocyanat für eine Denaturierung aller Proteine, einschließlich DNasen, RNasen und Peptidasen. Die Interphase wird mit einer Pipette in ein neues Gefäß überführt. Anschließend wird die enthaltene DNA einer Ethanol- oder Isopropanolfällung unterzogen. Vorteile der Zwei-Phasen-Extraktion sind geringe Kosten und keine Notwendigkeit einer Zentrifuge. Die Nachteile sind die Verwendung giftiger und flüchtiger Reagenzien bzw. die Notwendigkeit einer Laborabzugshaube sowie erhöhter Zeitbedarf.
Adsorption an Silicagel

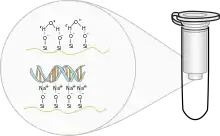
Bei dieser Fällungsmethode und Festphasenextraktion wird die DNA in leicht saurer Umgebung an eine Matrix aus Kieselgel (englisch silica gel),[17][18] Glaspuder,[19] Glasfaserfilter[20] oder Diethyl-Aminoethyl-Dextran (DEAE-Dextran) adsorbiert. Dabei kann auch selektiv einzel- und doppelsträngige DNA getrennt werden.[21] Die Adsorption erfolgt unter DNA-fällenden Pufferbedingungen (mit Alkoholen und leicht saurem pH-Wert) bei Silica und Glasfaser über polare Wechselwirkungen, bei DEAE-Dextran durch ionische Wechselwirkungen. Die Probe kann mehrfach auf die Matrix aufgetragen werden, um die Ausbeute zu erhöhen. Anschließend wird die in der Chromatographiesäule enthaltene Matrix mit chaotropen Pufferlösungen von den verunreinigenden DNA-bindenden Proteinen befreit. Die chaotropen Salze denaturieren die Proteine und halten sie in Lösung, während die DNA an die Matrix gebunden bleibt.[20] Bei einer chemischen Gelextraktion, also der Extraktion von DNA aus Agarosegelen durch Auflösung des Gels, wird oftmals als alternatives Chaotrop Natriumiodid verwendet.[22] Die Elution der DNA von der Matrix erfolgt durch Zugabe von Wasser oder leicht basischem Tris-Puffer mit EDTA (TE-Puffer). Durch eine optionale zweite Elution wird die absolute extrahierte DNA-Menge zu Lasten der Konzentration erhöht. Nach der Elution der DNA von der Kieselgel-Matrix folgt meistens ebenfalls die Ethanolfällung. Vorteile der Adsorption sind der geringere Zeitaufwand und keine Notwendigkeit einer Abzugshaube. Nachteile sind höhere Kosten und die Notwendigkeit einer Zentrifuge. Bei magnetischen Varianten wird die Zentrifuge vermieden, indem die Matrix magnetische Partikel enthält, die mit einem Magneten sedimentiert werden können.[23]
Lyse durch Kochen
Bei einer Lyse durch Kochen (engl. boiling lysis) werden bakterielle Zellen kurze Zeit nach Zugabe von Lysozym und Triton X-100 für 45 Sekunden gekocht, anschließend werden mit einem sterilen Zahnstocher die verklumpten Zelltrümmer (mit dem Großteil der chromosomalen DNA) entfernt.[24][25] Nach einer Zentrifugation wird der nun geklärte Überstand einer Ethanolfällung unterzogen. Diese Methode wird meistens zur Plasmidisolation aus Bakterien verwendet, da die chromosomale DNA mit anderen Bestandteilen verklumpt. Diese Methode eignet sich bei erhöhter Probenanzahl, erzeugt jedoch eine vergleichsweise etwas geringere Ausbeute und Reinheit, da Plasmide teilweise mit den verklumpten Bestandteilen entfernt werden und keine selektive Entfernung der Proteine erfolgt.
CTAB-Fällung
RNA und DNA bilden mit kationischen Tensiden unlösliche Komplexe.[26] Die CTAB-Fällung nutzt die Extraktion von DNA mit Cetyltrimethylammoniumbromid (CTAB), welche anschließend mit einer Chloroform-Extraktion und der Ethanolfällung kombiniert wird.[27] Sie wird meistens bei pflanzlichen Zellen angewendet. Nach einer Zerkleinerung im Mörser erfolgt der Zellaufschluss durch Zugabe von CTAB, Polyvinylpyrrolidon (zur Bindung von Polyphenolen) und Mercaptoethanol (zur Spaltung von Disulfidbrücken in Proteinen) in einem TRIS-Puffer bei einem pH-Wert von 8. Anschließend erfolgt meistens eine Chloroform-Isoamylalkohol- oder eine Chloroform-Octanol-Extraktion, bei der die DNA aufgrund des basischen pH-Werts in der wässrigen Phase verbleibt. Die wässrige Phase wird in einem neuen Reaktionsgefäß einer Ethanolfällung unterzogen. Analog erfolgt auch die Extraktion mit Catrimox-14, eine Methode der RNA-Extraktion.
Ethanolfällung

Bei der Ethanol- oder Isopropanolfällung wird die DNA unter leicht sauren Bedingungen (pH 5,2) in einer weniger polaren (alkoholischen) Umgebung aufgrund der Verringerung der Löslichkeit ausgefällt.[28] Wasser besitzt eine Dielektrizitätskonstante von 80,1 bei 25 °C, während diejenige von Ethanol bei etwa 24,3 liegt. Durch die Zugabe von Ethanol wird der DNA Wasser entzogen, wodurch die Löslichkeit sinkt. Der niedrige pH-Wert bei der Ethanolfällung wird durch die Zugabe von saurer Kaliumacetat- oder Natriumacetat-Lösung erzielt. Dies hält die sonst negativ geladene DNA in einem teilweise protonierten, teilweise mit Kalium- oder Natriumionen gesättigten und ungeladenen Zustand, wodurch die Löslichkeit weiter gesenkt wird. Bei einer alternativen Verwendung von Ammoniumacetat können die Proteine vor der Ethanolzugabe separat gefällt und durch einen zusätzlichen Zentrifugationsschritt abgetrennt werden, was eine Extraktion von Plasmid-DNA ohne eine Verwendung von Silica erlaubt.[29]
Nach Zugabe des Ethanols und der Kaliumacetatlösung wird der Versuchsansatz für 15 min. bei 10.000 g zentrifugiert. Bei niedrigen DNA-Konzentrationen oder kurzen DNA-Fragmenten unter 100 Basen oder Basenpaaren können der Fällungszeitraum auf mehrere Stunden verlängert und als Fällungshilfe Polymere wie z. B. Glykogen, tRNA oder lineares Polyacrylamid hinzugegeben werden. Der Überstand wird nach der Zentrifugation abgenommen und verworfen. Meistens wird im Anschluss 70%iges Ethanol als Waschlösung zugesetzt, erneut zentrifugiert und der Überstand wieder verworfen. Dadurch wird die DNA entsalzt und von Resten an Extraktionsreagenzien befreit. Der Niederschlag wird getrocknet und anschließend in TE-Puffer gelöst. Da die Ethanolfällung relativ unsauber trennt und einige Proteine, Polysaccharide und RNA gleichzeitig mit der DNA präzipitieren, wird sie meist nur als abschließender Reinigungsschritt und mehrfach hintereinander an derselben Probe eingesetzt.
Endotoxinentfernung
In manchen Fällen muss die DNA von kontaminierenden Endotoxinen befreit werden, z. B. zur Vermeidung einer Aktivierung des TLR-4 bei einer Anwendung gereinigter Plasmid-DNA in TLR4-positiven Organismen. Hierzu werden unter anderem die Zwei-Phasen-Wolkenpunktextraktion mit Triton-X114 oder die Chromatographie verwendet,[30][31] beide Methoden nacheinander[32][33] oder in Kombination.[34]
Die Wolkenpunktextraktion basiert auf der Phasentrennung einer einprozentigen (m/V) Triton-X114-Lösung oberhalb von 22 °C oder nach der Zugabe von Salzen und Natriumlaurylsulfat.[35]
Als Material in einer Chromatographiesäule zur Endotoxinentfernung wird unter anderem Hydroxylapatit, Polystyrol, Dowex 1-X2 (stark basischer Anionenaustauscher), Aktivkohle oder Polymyxin-B-modifiziertes Säulenmaterial verwendet.[36][37]
Quantifizierung
Die Mengenbestimmung gereinigter DNA erfolgt am einfachsten per Photometrie. Soll dagegen die Menge einer bestimmten DNA-Sequenz in einer Mischung von DNA-Sequenzen bestimmt werden, kommt meistens die QPCR zum Einsatz. Bei der Photometrie entspricht eine Extinktion E von 1 einer gereinigten DNA-Lösung bei doppelsträngiger DNA und einer Wellenlänge von 260 nm einer Konzentration von 50 Mikrogramm pro Milliliter, bei einzelsträngiger RNA entspricht dies 40 Mikrogramm pro Milliliter und bei einzelsträngiger DNA 33 Mikrogramm pro Milliliter.[38]
| Nukleinsäure | E260 nm = 1 entspricht[38] |
|---|---|
| dsDNA | 50 μg/ml |
| ssRNA | 40 μg/ml |
| ssDNA | 33 μg/ml |
Bei einzelsträngigen Oligonukleotiden kann die entsprechende Konzentration anhand der Anzahl der verschiedenen Nukleinbasen (nA für die Anzahl an Adeninen) berechnet werden:[39]
Die Berechnung des Extinktionskoeffizienten kann für einzelsträngige und doppelsträngige DNA mit der Nearest-Neighbor-Heuristik erfolgen.[40] Ein Extinktionskoeffizient erlaubt die Bestimmung der Konzentration ohne Standardreihe nach dem Lambert-Beerschen Gesetz. Der Extinktionskoeffizient ϵ bei einer Wellenlänge von 260 nm berechnet sich wie folgt:[41]
Reinheit
Das Verhältnis aus der Extinktion bei 260 nm und der Extinktion bei 280 nm wird als Indikator der Reinheit der DNA verwendet. Proteine absorbieren relativ stark bei 280 nm aufgrund der enthaltenen Aminosäuren Phenylalanin, Tyrosin und Tryptophan. Reine DNA besitzt ein Verhältnis der Extinktionen von zwei, eine reine Proteinprobe liegt dagegen bei 0,57. Da aber der Extinktionskoeffizient von Proteinen über eine Zehnerpotenz niedriger als der von DNA liegt,[42] bedeutet ein Verhältnis von 1,9 einen Proteinanteil von 40 % und ein Verhältnis von 1,8 einen Proteinanteil von 60 %.[43] Eine Berechnung der DNA-Konzentration anhand des Extinktionskoeffizienten ist ohne eine Berücksichtigung der Reinheit daher nur bei einem Verhältnis von 2 sinnvoll.
| % Protein | % Nukleinsäure | E260 nm/E280 nm |
|---|---|---|
| 100 | 0 | 0,57 |
| 95 | 5 | 1,06 |
| 90 | 10 | 1,32 |
| 70 | 30 | 1,73 |
| 60 | 40 | 1,8 |
| 40 | 60 | 1,9 |
| 30 | 70 | 1,94 |
| 10 | 90 | 1,98 |
| 5 | 95 | 1,99 |
| 0 | 100 | 2,00 |
Eine Möglichkeit zur Berechnung der Konzentration unter Einbeziehung der Reinheit erfolgt nach folgender Formel, mit R als Verhältnis der Extinktionen bei 260 nm und 280 nm:[42][44]
Durch Einsetzen der Extinktionskoeffizienten ergibt sich:[42]
Wo technisch verfügbar, ist es sinnvoller das Verhältnis der Extinktionen bei 260 nm und 234 nm als Reinheitsindikator zu verwenden.[42] Bei 234 nm absorbiert DNA wenig im Vergleich zu 280 nm, aber die Peptidbindung der Proteine relativ stark. Da die Extinktionskoeffizienten für Protein bei 234 nm nur 1,5- bis 1,8-fach niedriger als die von DNA liegen, ist dieses Maß empfindlicher für Proteinverunreinigungen.[42] Analog kann die Konzentration unter Einbeziehung der Reinheit nach folgender Formel berechnet werden:[42]
Geschichte
Die erste Extraktion von DNA wurde 1869 von Friedrich Miescher durchgeführt.[45] Ab 1951 wurde erstmals von E. Volkin und C. E. Carter das Chaotrop Guanidiniumchlorid zur Denaturierung von Proteinen mit anschließender Chloroform-Extraktion der RNA und Ethanolfällung eingesetzt,[46] ab 1963 wurde es von R. Cox und H. Arnstein bei der RNA-Extraktion eingesetzt.[47] Die Zwei-Phasen-Extraktion, auf der die drei heutigen Methoden zur Zwei-Phasen-Extraktion basieren, entstammt einer Extraktionsmethode mit Methanol und Chloroform, welche 1959 für Lipide entwickelt wurde.[48] Phenol wurde erstmals 1953 von W. Grassman und G. Deffner zur denaturierenden Proteinextraktion eingesetzt,[49] ab 1956 wurde Phenol von K. Kirby zur RNA-Extraktion eingesetzt.[50] Die Verwendung des Chaotrops Guanidiniumthiocyanat wurde erstmals 1977 von A. Ullrich und Kollegen erwähnt und 1979 von J. Chirgwin und Kollegen beschrieben.[51][52] H. Birnboim und J. Doly kombinierten ab 1979 die Alkalische Lyse von Bakterienzellen mit der DNA-Extraktion.[1] Im Jahr 1981 beschrieben J. Feramisco und Kollegen eine RNA-Extraktion mit Guanidiniumthiocyanat und heißem Phenol.[53] J. Zeugin und J. Hartley veröffentlichten 1985 die heutige Variante der Ethanolfällung.[28] Piotr Chomczynski und Nicoletta Sacchi verwendeten 1987 erstmals Guanidiniumthiocyanat und sauren pH-Wert in Kombination mit der Zwei-Phasen-Extraktion zur Denaturierung und Lösungsvermittlung der Proteine während der Extraktion und getrennten Fällung von DNA und RNA, wodurch eine auch weniger abgebaute RNA erhalten wurde.[15] Im Jahr 1989 wurde die selektive Adsorption von DNA und ihre Festphasenextraktion von R. McCormick beschrieben[54] und 1990 von R. Boom modifiziert.[17] Um höhere Reinheiten zu erreichen, wurde 1998 von Meijer und Kollegen eine Kombination aus Zwei-Phasen- und Silicaadsorption entwickelt.[55]
Literatur
- Friedrich Lottspeich, Haralabos Zorbas: Bioanalytik. Spektrum Akademischer Verlag, Heidelberg 1998, ISBN 978-3827400413.
- Cornel Mülhardt: Der Experimentator: Molekularbiologie/Genomics. Sechste Auflage. Spektrum Akademischer Verlag, Heidelberg 2008, ISBN 3-82-742036-9.
- J. Sambrook, T. Maniatis, D. W. Russel: Molecular cloning: a laboratory manual. 3rd edition (2001), Cold Spring Harbor Laboratory Press. ISBN 0-87969-577-3.
Einzelnachweise
- H. C. Birnboim, J. Doly: A rapid alkaline extraction procedure for screening recombinant plasmid DNA. In: Nucleic Acids Res. (1979), Band 7(6), S. 1513–23. PMID 388356; PMC 342324 (freier Volltext).
- B. Hirt: Selective extraction of polyoma DNA from infected mouse cell cultures. In: Journal of molecular biology. Band 26, Nummer 2, Juni 1967, S. 365–369, PMID 4291934.
- H. Yoshikawa, F. Dogruman-Al, F. Dogruman-Ai, S. Turk, S. Kustimur, N. Balaban, N. Sultan: Evaluation of DNA extraction kits for molecular diagnosis of human Blastocystis subtypes from fecal samples. In: Parasitology research. Band 109, Nummer 4, Oktober 2011, S. 1045–1050, doi:10.1007/s00436-011-2342-3, PMID 21499752.
- J. J. Doyle, J. L. Doyle: A rapid DNA isolation procedure for small quantities of fresh leaf tissue. In: Phytochem. Bull. (1987), Band 19, S. 11–15.
- David V. Jobes, David L. Hurley, Leonard B. Thien: Plant DNA isolation: a method to efficiently remove polyphenolics, polysaccharides, and RNA. In: TAXON. 44, 2019, S. 379, doi:10.2307/1223408.
- T. Dey, S. Saha, T. N. Dhar, S. Adhikary, P. Ghosh: Optimization and comparison of efficiency between two DNA isolation protocols in Cymbopogon species. In: General and Applied Plant Physiology (2010). Band 36, S. 232–238. (PDF).
- S. Adhikari, S. K. Chattopadhyay, P. D. Ghosh: A simplified high yielding miniprep genomic DNA extraction protocol for three chemotypically different plant species. In: Indian Journal of Biotechnology (2012), Band 11, S. 337–340, (PDF).
- P. Piper: Isolation of yeast DNA. In: Methods in molecular biology. Band 53, 1996, S. 103–107, doi:10.1385/0-89603-319-8:103, PMID 8924971.
- L. Fauchery, S. Uroz, M. Buée, A. Kohler: Purification of Fungal High Molecular Weight Genomic DNA from Environmental Samples. In: Methods in molecular biology. Band 1775, 2018, S. 21–35, doi:10.1007/978-1-4939-7804-5_3, PMID 29876806.
- J. Marmur: A procedure for the isolation of deoxyribonucleic acid from micro-organisms. In: Journal of Molecular Biology. 3, 1961, S. 208, doi:10.1016/S0022-2836(61)80047-8.
- J. Marmur: This week's citation classic. In: Current Contents (1991), Band 34, Heft 36. S. 11–12. (PDF).
- K. L. Greathouse, R. Sinha, E. Vogtmann: DNA extraction for human microbiome studies: the issue of standardization. In: Genome biology. Band 20, Nummer 1, 10 2019, S. 212, doi:10.1186/s13059-019-1843-8, PMID 31639026, PMC 6802309 (freier Volltext).
- Francisco Salvà Serra, Francisco Salvà-Serra, Liselott Svensson-Stadler, Antonio Busquets, Daniel Jaén-Luchoro, Roger Karlsson, Edward R. B. Moore, Margarita Gomila: A protocol for extraction and purification of high-quality and quantity bacterial DNA applicable for genome sequencing: a modified version of the Marmur procedure.. In: Protocol Exchange. 2018, doi:10.1038/protex.2018.084.
- J. M. Graham, D. Rickwood: Subcellular Fractionation: A Practical Approach. Oxford University Press, 1997. ISBN 9780191591617.
- P. Chomczynski, N. Sacchi: Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. In: Anal Biochem. (1987) 162(1):156-9. PMID 2440339.
- P. Chomczynski, K. Mackey, R. Drews, W. Wilfinger: DNAzol: a reagent for the rapid isolation of genomic DNA. In: BioTechniques. Band 22, Nummer 3, März 1997, S. 550–553, doi:10.2144/97223pf01, PMID 9067036.
- R. Boom, C. J. Sol, M. M. Salimans, C. L. Jansen, P. M. Wertheim-van Dillen, J. van der Noordaa: Rapid and simple method for purification of nucleic acids. In: Journal of clinical microbiology. Band 28, Nummer 3, März 1990, S. 495–503, ISSN 0095-1137. PMID 1691208. PMC 269651 (freier Volltext).
- R. Boom, C. Sol, M. Beld, J. Weel, J. Goudsmit, P. Wertheim-van Dillen: Improved silica-guanidiniumthiocyanate DNA isolation procedure based on selective binding of bovine alpha-casein to silica particles. In: Journal of clinical microbiology. Band 37, Nummer 3, März 1999, S. 615–619, ISSN 0095-1137. PMID 9986822. PMC 84491 (freier Volltext).
- M. A. Marko, R. Chipperfield, H. C. Birnboim: A procedure for the large-scale isolation of highly purified plasmid DNA using alkaline extraction and binding to glass powder. In: Anal Biochem. (1982), Band 121(2), S. 382–7. PMID 6179438.
- T. A. Borodina, H. Lehrach, A. V. Soldatov: DNA purification on homemade silica spin-columns. In: Anal Biochem. (2003), Band 321(1), S. 135–7. PMID 12963065.
- M. Beld, C. Sol, J. Goudsmit, R. Boom: Fractionation of nucleic acids into single-stranded and double-stranded forms. In: Nucleic acids research. Band 24, Nummer 13, Juli 1996, S. 2618–2619, ISSN 0305-1048. PMID 8692706. PMC 145977 (freier Volltext).
- B. Vogelstein, D. Gillespie: Preparative and analytical purification of DNA from agarose. In: Proc Natl Acad Sci U S A. (1979), Band 76(2), S. 615–9. PMID 284385; PMC 382999 (freier Volltext).
- S. C. Tan, B. C. Yiap: DNA, RNA, and protein extraction: the past and the present. In: Journal of biomedicine & biotechnology. Band 2009, 2009, S. 574398, doi:10.1155/2009/574398, PMID 20011662, PMC 2789530 (freier Volltext).
- D. S. Holmes, M. A. Quigley: A rapid boiling method for the preparation of bacterial plasmids. In: Anal Biochem. (1981), Band 114, Nr. 1, S. 193–197. PMID 6269464.
- L. V. Andreou: Isolation of plasmid DNA from bacteria. In: Methods Enzymol. (2013), Band 529, S. 135–142. doi:10.1016/B978-0-12-418687-3.00010-0. PMID 24011041.
- S. K. Dutta, A. S. Jones, M. Stacey: The separation of desoxypentosenucleic acids and pentosenucleic acids. In: Biochimica et Biophysica Acta. Band 10, Nummer 4, April 1953, S. 613–622, PMID 13059025.
- J. C. Murphy, M. A. Winters, S. L. Sagar: Large-scale, nonchromatographic purification of plasmid DNA. In: Methods Mol Med. (2006), Band 127, S. 351–62. PMID 16988465.
- J. A. Zeugin, J. L. Hartley: Ethanol Precipitation of DNA. In: Focus (1985), Band 7(4). PDF.
- J. Crouse, D. Amorese: Ethanol Precipitation: Ammonium Acetate as an Alternative to Sodium Acetate. In: Focus (1987), Band 9(2), S. 3–5. PDF (Memento vom 22. November 2009 im Internet Archive).
- Pérola O. Magalhães, André M. Lopes, Priscila G. Mazzola, Carlota Rangel-Yagui, Thereza C. V. Penna, Adalberto Pessoa Jr.: Methods of Endotoxin Removal from Biological Preparations: a Review. In: J Pharm Pharmaceut Sci. (2007), Band 10, Nummer 3, S. 388–404. PDF
- US-Patent US20130004562.
- A. Rozkov, B. Larsson, S. Gillström, R. Björnestedt, S. R. Schmidt: Large-scale production of endotoxin-free plasmids for transient expression in mammalian cell culture. In: Biotechnology and bioengineering. Band 99, Nummer 3, Februar 2008, S. 557–566, ISSN 1097-0290. doi:10.1002/bit.21603. PMID 17680665.
- W. Pi, C. Sun, Z. Song, L. Ma, S. Liu, D. Bai, Y. Cai, S. Liu: [Purification of plasmid DNA using anion-exchange chromatography and removal of endotoxin]. In: Se pu = Chinese journal of chromatography / Zhongguo hua xue hui. Band 25, Nummer 6, November 2007, S. 809–813, ISSN 1000-8713. PMID 18257294.
- Patent WO 95/21177, US-Patent US5747663 A, US6953686, Europäisches Patent EP211968.
- R. Ma, J. Zhao, H. C. Du, S. Tian, L. W. Li: Removing endotoxin from plasmid samples by Triton X-114 isothermal extraction. In: Analytical biochemistry. Band 424, Nummer 2, Mai 2012, S. 124–126, ISSN 1096-0309. doi:10.1016/j.ab.2012.02.015. PMID 22370278.
- S. K. Maitra, T. T. Yoshikawa, L. B. Guze, M. C. Schotz: Properties of binding of Escherichia coli endotoxin to various matrices. In: Journal of clinical microbiology. Band 13, Nummer 1, Januar 1981, S. 49–53, ISSN 0095-1137. PMID 7007429. PMC 273719 (freier Volltext).
- M. Cotten, A. Baker, M. Saltik, E. Wagner, M. Buschle: Lipopolysaccharide is a frequent contaminant of plasmid DNA preparations and can be toxic to primary human cells in the presence of adenovirus. In: Gene therapy. Band 1, Nummer 4, Juli 1994, S. 239–246, ISSN 0969-7128. PMID 7584087.
- Wolfgang Hennig: Genetik. Springer-Verlag, 2013, ISBN 978-3-662-07430-5 (eingeschränkte Vorschau in der Google-Buchsuche).
- Frank H. Stephenson: Calculations for Molecular Biology and Biotechnology. Academic Press, 2016, ISBN 978-0-128-02598-7, S. 111.
- A. V. Tataurov, Y. You, R. Owczarzy: Predicting ultraviolet spectrum of single stranded and double stranded deoxyribonucleic acids. In: Biophysical chemistry. Band 133, Nummer 1–3, März 2008, S. 66–70, doi:10.1016/j.bpc.2007.12.004, PMID 18201813.
- Patrick Couvreur: Pharmaceutical Aspects of Oligonucleotides. CRC Press, 2003, ISBN 978-0-203-30566-9, S. 60.
- Stefan Surzycki: Basic Techniques in Molecular Biology. Springer Science & Business Media, 2012, ISBN 978-3-642-56968-5, S. 24.
- J. A. Glasel: Validity of nucleic acid purities monitored by 260nm/280nm absorbance ratios. In: BioTechniques. Band 18, Nummer 1, Januar 1995, S. 62–63, PMID 7702855.
- V. F. Kalb, R. W. Bernlohr: A new spectrophotometric assay for protein in cell extracts. In: Analytical biochemistry. Band 82, Nummer 2, Oktober 1977, S. 362–371, PMID 20815.
- R. Dahm: Discovering DNA: Friedrich Miescher and the early years of nucleic acid research. In: Human Genetics. Band 122, Nummer 6, Januar 2008, S. 565–581, doi:10.1007/s00439-007-0433-0, PMID 17901982 (Review).
- E. Volkin, C. E. Carter: The Preparation and Properties of Mammalian Ribonucleic Acids. Un J. Am. Chem. Soc. (1951), Band 73, Heft 4, S. 1516–1519.
- R. A. Cox, H. R. V. Arnstein: The isolation, characterization and acid–base properties of ribonucleic acid from rabbit-reticulocyte ribosomes. In: J. Biochem. (1963), Band 89, S. 574–584, PMID 14101978, PMC 1202465 (freier Volltext).
- E. G. Bligh, W. J. Dyer: A rapid method of total lipid extraction and purification. In: Can J Biochem Physiol. (1959) Aug;37(8):911-7. PMID 13671378.
- W. Grassman, G. Deffner: Verteilungschromatographisches Verhalten von Proteinen und Peptiden in phenolhaltigen Lösungsmitteln. In: Hoppe Seyler’s Z. Physiol. Chem. (1953), Band 293, S. 89–98.
- K. S. Kirby: A New Method for the Isolation of Ribonucleic Acids from Mammalian Tissues. In: Biochem. J. (1956), Band 64, S. 405–408, PMID 13373784, PMC 1199750 (freier Volltext).
- A. Ullrich, J. Shine, J. Chirgwin, R. Pictet, E. Tischer, W. J. Rutter, H. M. Goodman: Rat insulin genes: construction of plasmids containing the coding sequences. In: Science. Band 196, Nummer 4296, Juni 1977, S. 1313–1319, PMID 325648.
- J. M. Chirgwin, A. E. Przybyla, R. J. MacDonald, W. J. Rutter: Isolation of biologically active ribonucleic acid from sources enriched in ribonuclease. In: Biochemistry. Band 18, Nummer 24, November 1979, S. 5294–5299, PMID 518835.
- J. R. Feramisco, J. E. Smart, K. Burridge, D. M. Helfman, G. P. Thomas: Co-existence of vinculin and a vinculin-like protein of higher molecular weight in smooth muscle. In: The Journal of biological chemistry. Band 257, Nummer 18, September 1982, S. 11024–11031, PMID 6809764.
- R. M. McCormick: A solid-phase extraction procedure for DNA purification. In: Analytical biochemistry. Band 181, Nummer 1, August 1989, S. 66–74, doi:10.1016/0003-2697(89)90394-1, PMID 2554759.
- A. Meijer, J. A. van der Vliet, L. M. Schouls, A. de Vries, P. J. Roholl, J. M. Ossewaarde: Detection of microorganisms in vessel wall specimens of the abdominal aorta: development of a PCR assay in the absence of a gold standard. In: Research in microbiology. Band 149, Nummer 8, September 1998, S. 577–583, doi:10.1016/s0923-2508(99)80005-9, PMID 9795995.