Horizontaler Gentransfer
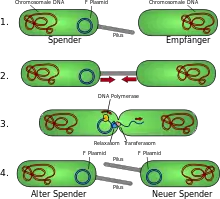
Horizontaler Gentransfer (HGT) oder lateraler Gentransfer (LGT) bezeichnet eine Übertragung von genetischem Material nicht entlang der Abstammungslinie, also nicht von einer Generation zur darauf folgenden, sondern „horizontal“ von einem Organismus in einen bereits existierenden anderen hinein. Im Unterschied dazu erfolgt der vertikale Gentransfer von Vorfahren zu Nachkommen. Der bereits nachgewiesene Gentransfer aus Gen-Resten über Zeitgrenzen hinweg wird als anachronistische Evolution bezeichnet. Natürlichen lateralen Gentransfer beobachtet man vorwiegend bei Prokaryoten, insbesondere bei Bakterien, die durch Konjugation einen Teil ihres genetischen Materials an ein anderes Individuum weiterreichen. Anders als bei der vertikalen Transmission, bei der Erbgut eines Krankheitserregers von einer infizierten Generation auf die nächste übertragen wird (z. B. bei Wolbachia pipientis), ist der horizontale Gentransfer nicht an einen Fortpflanzungsvorgang gebunden. Die Gene befinden sich oftmals in einer mobilen Form (ein Vektor wie Bakteriophagen oder Plasmide). Beispielsweise bei der Konjugation von Bakterien kommt es – unabhängig von Vermehrungsvorgängen – zu einer Übertragung von Erbgut zwischen Individuen derselben Art oder auch verschiedener Arten. Dabei wird ein zuvor repliziertes Plasmid auf eine Empfängerzelle übertragen. Hierdurch erfolgt eine Anreicherung des Empfängerorganismus mit genetischer Information.
Evolution
Horizontaler Gentransfer bildet daher in der Evolutionstheorie eine Möglichkeit zur Erklärung von Sprüngen in der Entwicklung vor allem bei den Mikroorganismen. Der horizontale Gentransfer ermöglicht eine beschleunigte Anpassung an veränderte Umgebungsbedingungen, z. B. durch Weitergabe von Antibiotikaresistenzen oder eines Virulenzfaktors, teilweise auch gruppiert in sog. Pathogenitätsinseln.
Der horizontale Gentransfer erschwert die Bestimmung von Stammbäumen durch molekulare Uhren.[2][3][4] Auf der anderen Seite wird postuliert, dass horizontaler Gentransfer die Aufrechterhaltung einer universellen Lebensbiochemie und folglich die Universalität des genetischen Codes fördert.[5] Mit anderen Worten: Der Horizontale Gentransfer überwacht die Universalität des genetischen Codes und zwingt diesen in eine lingua franca des Lebens auf der Erde.[6] Auf diese Weise kann genetischer Austausch[7] oder die Verbreitung von biologischer Neuheit durch die Biosphäre stattfinden.[8]
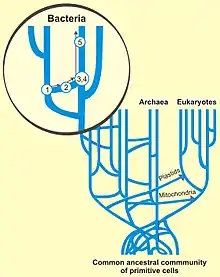
Seit das komplette Genom einer Vielzahl von Bakterien und Archaeen sequenziert wurde, hat sich gezeigt, dass auch der Gentransfer zwischen entfernt verwandten Organismen eine große Rolle gespielt hat, insbesondere, wenn sie überlappende ökologische Nischen bewohnen. Bei mesophilen Bakterien, die am besten bei Temperaturen zwischen etwa 20 und 45 °C gedeihen und anaerob leben, fand man, dass 16 % des Genoms aus horizontalem Gentransfer stammen. Ein Drittel der Gene von Enzymen des chemotrophen Stoffwechsels stammen aus einem Gentransfer, während ribosomale Proteine um den Faktor 150 seltener horizontal ausgetauscht wurden. Der Anteil an durch Genaustausch übertragenen Genen ist bei aeroben Mikroorganismen etwa um den Faktor 2 niedriger. Auch bei ihnen hat der HGT erheblich zu ihrer Anpassungsfähigkeit und der Vielfalt der von Mikroorganismen genutzten Substrate beigetragen.[9]
Eine wichtige Rolle hat auch der Gentransfer von Bakterien zu den Archaeen gespielt. Ungefähr 5 % des Genoms des methanogenen Organismus Methanosarcina ist bakteriellen Ursprungs.[10] Diese Organismen haben durch die transferierten Gene die Fähigkeit erworben, Acetat zu nutzen und in Methan umzuwandeln.[11] Es ist möglich, dass diese durch HGT erworbene Fähigkeit der methanogenen Archaeen zu der Klimakatastrophe vor rund 252 Millionen Jahren (Perm-Trias-Grenze) beigetragen hat, die damals zu dem größten bekannten Massenaussterben der Erdgeschichte geführt hat.[12]
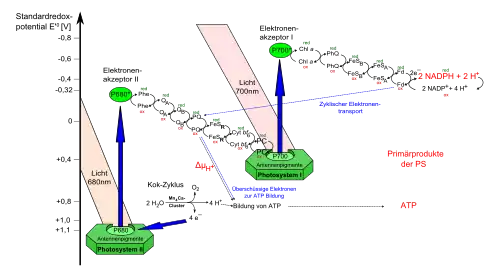
Noch einschneidender in der Erdgeschichte war das Entstehen von molekularem Sauerstoff und seine Anreicherung in der Erdatmosphäre. Für diese oxygene Photosynthese waren Cyanobakterien verantwortlich, die durch Lichtenergie aus Wasser O2 freisetzen. Dazu verwenden sie in ihren Membranen eine chemiosmotisch gekoppelte Elektronentransportkette. Sie besteht aus zwei Systemen, die getrennt von nicht verwandten Bakterien entwickelt worden waren und dann per HGT zu einem Vorfahren der Cyanobakterien gelangten.[13]
Endosymbiotischer Gentransfer
Gemäß der Endosymbiontentheorie sind Plastiden (Chloroplasten u. a.) und mitochondrien-ähnliche Organellen (englisch mitochondrion-related organelles, MROs) Nachkömmlinge von Cyanobakterien respektive Alphaproteobacterien (o. ä.), die im Lauf der Evolution einen immer größeren Teil ihrer Gene durch endosymbiotischen Gentransfer (EGT) auf den Zellkern übertragen haben. Dies ging mit einem Verlust der Selbstständigkeit einher, durch den die ursprünglichen Bakterien zu Organellen wurden.[14][15] Es handelt sich beim EGT somit um einen horizontalen Gentransfer vom Endosymbiont zum Wirt. Dieser kann auch heute noch beobachtet werden.[16][17] Der EGT kann in letzter Konsequenz zum völligen Verlust der DNA des Organells führen.[18]
Gentechnik
Horizontaler Gentransfer bildet in der Gentechnik eine wichtige Methode, um transgene Organismen zu erzeugen. Für Prokaryoten und Eukaryoten sind jeweils unterschiedliche Methoden erfolgreich in Gebrauch. Bei Prokaryoten werden die Konjugation, die Transduktion und die Transformation verwendet. Bei Eukaryoten kann als Äquivalent zur Transformation bei Prokaryoten eine Transfektion durchgeführt werden.
Gentherapie
Das Einbringen von genetischem Material in menschliche Keimzellen, die sogenannte „Keimbahntherapie“, etwa zur Behebung eines Gendefekts, ist in Deutschland wegen der hohen Risiken durch das Embryonenschutzgesetz von 1991 verboten. Das Einschleusen therapeutisch wirksamen genetischen Materials in die Körperzellen eines bereits existierenden Empfängerorganismus (horizontaler Gentransfer) ist erlaubt, findet in einzelnen Bereichen bereits erfolgreich Anwendung und wird als somatische Gentherapie bezeichnet.
Nachweise und Beispiele
Direkt nachgewiesen wurde die Übertragung prokaryotischer DNA auf Eukaryoten bisher bei Agrobacterium tumefaciens (Pflanzenzellen) und Bartonella henselae (menschliche Zellen).[19][20]
Ein Beispiel für einen horizontalen Gentransfer unter höheren Organismen ist der Transfer von Genen für die Carotinoid-Synthese von einem Pilz auf die Erbsenlaus (Acyrthosiphon pisum).[21][22]
Ebenso gilt als Beispiel die Übernahme eines Cellulasegens durch einen Zweig der Nematoda von ihren Endosymbionten.[23][24]
2012 fanden Ricardo Acuña und Kollegen[25] Indizien dafür, dass der Kaffeekirschenkäfer (Hypothenemus hampei) das Gen HhMAN1 für das Verdauungsenzym Mannanase durch horizontalen Gentransfer von einem noch unidentifizierten Bakterium erworben hat. Das Enzym erlaubt es dem Käfer, Galactomannane, die wichtigsten Speicher-Kohlenhydrate der Kaffeebohne, aufzuschließen und zu verdauen.
Bei einer umfassenden Suche nach Bakteriengenen in den inzwischen zahlreichen offen zugänglichen Sequenzen menschlicher Genome wurden insbesondere in bestimmten Krebszellen eingeschleuste Bakteriengene gefunden, aber in wenigen Fällen auch in gesunden Zellen.[26]
Einzelnachweise
- Nature Reviews Microbiology. 3, September 2005, S. 675–678.
- J. H. Badger: Genomic analysis of Hyphomonas neptunium contradicts 16S rRNA gene-based phylogenetic analysis: implications for the taxonomy of the orders 'Rhodobacterales' and Caulobacterales. In: INTERNATIONAL JOURNAL OF SYSTEMATIC AND EVOLUTIONARY MICROBIOLOGY. 55, 2005, S. 1021, doi:10.1099/ijs.0.63510-0. PMID 24318976.
- Nicholas J Matzke, Patrick M Shih, Cheryl A Kerfeld: Bayesian Analysis of Congruence of Core Genes in Prochlorococcus and Synechococcus and Implications on Horizontal Gene Transfer. In: PLoS One. Band 9, Nr. 1, 2014, doi:10.1371/journal.pone.0085103, PMC 3897415 (freier Volltext).
- Volker Knoop, Kai Müller: Gene und Stammbäume: ein Handbuch zur molekularen Phylogenetik. 2. Auflage. Spektrum Akademischer Verlag, Heidelberg 2009, ISBN 978-3-8274-2230-9, S. 331–333, doi:10.1007/978-3-8274-2230-9.
- V. Kubyshkin, C. G. Acevedo-Rocha, N. Budisa: On universal coding events in protein biogenesis. In: Biosystems. 2017. doi:10.1016/j.biosystems.2017.10.004.
- V. Kubyshkin, N. Budisa: Synthetic alienation of microbial organisms by using genetic code engineering: Why and how?. In: Biotechnologie Journal. 12, 2017, S. 1600097. doi:10.1002/biot.201600097.
- C. R. Woese: A New Biology for a New Century. In: Microbiology and Molecular Biology Reviews. 68, 2004, S. 173–186. doi:10.1128/MMBR.68.2.173-186.2004.
- J. P. Gogarten, J. P. Townsend: Horizontal gene transfer, genome innovation and evolution. In: Nature Reviews Microbiology. 3, 2005, S. 679–687. doi:10.1038/nrmicro1204.
- Alejandro Caro-Quintero, Konstantinos T. Konstantinidis: Inter-phylum HGT has shaped the metabolism of many mesophilic and anaerobic bacteria. In: ISME J. Band 9, Nr. 4, 2015, S. 958–967, doi:10.1038/ismej.2014.193.
- Sofya K. Garushyants, Marat D. Kazanov, Mikhail S. Gelfand: Horizontal gene transfer and genome evolution in Methanosarcina. In: BMC Evolutionary Biology. Band 15, Nr. 1, 2015, S. 01–14, doi:10.1186/s12862-015-0393-2.
- Gregory P. Fournier, J. Peter Gogarten: Evolution of acetoclastic methanogenesis in Methanosarcina via horizontal gene transfer from cellulolytic Clostridia. In: Journal of bacteriology. Band 190, Nr. 3, 2008, S. 1124–1127.
- Daniel H. Rothman, Gregory P. Fournier, Katherine L. French, Eric J. Alm, Edward A. Boyle, Changqun Cao, Roger E. Summons: Methanogenic burst in the end-Permian carbon cycle. In: PNAS. 111 (15), 2014, S. 5462–5467; print March 31, 2014, doi:10.1073/pnas.1318106111.
- Georg Fuchs, Hans Günter Schlegel, Thomas Eitinger: Allgemeine Mikrobiologie. 9., vollständig überarbeitete und erweiterte Auflage. Georg Thieme Verlag, Stuttgart 2014, ISBN 978-3-13-444609-8, Kap. 15, S. 472 links unten.
- William Martin, Tamas Rujan, Erik Richly, Andrea Hansen, Sabine Cornelsen, Thomas Lins, Dario Leister, Bettina Stoebe, Masami Hasegawa, David Penny: Evolutionary analysis of Arabidopsis, cyanobacterial, and chloroplast genomes reveals plastid phylogeny and thousands of cyanobacterial genes in the nucleus. In: Proceedings of the National Academy of Sciences. 99, Nr. 19, 2002, S. 12246–12251. bibcode:2002PNAS...9912246M. doi:10.1073/pnas.182432999. PMID 12218172. PMC 129430 (freier Volltext).
- Chun Y. Huang, Michael A. Ayliffe, Jeremy N. Timmis: Direct measurement of the transfer rate of chloroplast DNA into the nucleus. In: Nature. 422, Nr. 6927, 2003, S. 72–6. bibcode:2003Natur.422...72H. doi:10.1038/nature01435. PMID 12594458.
- DNA Jumps Directly from the Cell’s Chloroplasts into its Nucleus, auf: SciTechDaily vom 16. April 2012
- Direkter Transfer von Pflanzen-Genen aus Chloroplasten in den Zellkern, Pressemitteilung der MPG (MPI für molekulare Pflanzenphysiologie) vom 13. April 2012
- Tel Aviv University researchers discover unique non-oxygen breathing animal, auf: EurekAlert! vom 25. Februar 2020
- M. Van Montagu: It is a long way to GM agriculture. In: Annual review of plant biology. Band 62, Juni 2011, S. 1–23, ISSN 1545-2123. doi:10.1146/annurev-arplant-042110-103906. PMID 21314429.
- G. Schröder, R. Schuelein u. a.: Conjugative DNA transfer into human cells by the VirB/VirD4 type IV secretion system of the bacterial pathogen Bartonella henselae. In: PNAS. Band 108, Nummer 35, August 2011, S. 14643–14648. doi:10.1073/pnas.1019074108. PMID 21844337. PMC 3167556 (freier Volltext).
- Lateral Transfer of Genes from Fungi Underlies Carotenoid Production in Aphids. In: Science/AAAS. www.sciencemag.org, abgerufen am 15. Januar 2011.
- A Fungal Past to Insect Color. In: Science/AAAS. www.sciencemag.org, abgerufen am 15. Januar 2011.
- John T. Jones, Cleber Furlanetto, Taisei Kikuchi: Horizontal gene transfer from bacteria and fungi as a driving force in the evolution of plant parasitism in nematodes. (Memento des Originals vom 21. September 2013 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis. In: Nematology. Band 7, Nr. 5, 2005, S. 641–646.
- Werner E. Mayer u. a.: Horizontal gene transfer of microbial cellulases into nematode genomes is associated with functional assimilation and gene turnover. In: BMC Evolutionary Biology. Band 11, Nr. 1, 2011, S. 13.
- Ricardo Acuña, Beatriz E. Padilla, Claudia P. Flórez-Ramos, José D. Rubio, Juan C. Herrera, Pablo Benavides, Sang-Jik Lee, Trevor H. Yeats, Ashley N. Egan, Jeffrey J. Doyle, Jocelyn K. C. Rose: Adaptive horizontal transfer of a bacterial gene to an invasive insect pest of coffee. In: Proceedings of the National Academy of Sciences USA. vol. 109 no. 11, 2012, S. 4197–4202, doi:10.1073/pnas.1121190109.
- David R. Riley, Karsten B. Sieber u. a.: Bacteria-Human Somatic Cell Lateral Gene Transfer Is Enriched in Cancer Samples. In: PLoS Computational Biology. 9, 2013, S. e1003107, doi:10.1371/journal.pcbi.1003107.