Enterobakterien
Die Enterobakterien bzw. die Vertreter der 2016 etablierten Ordnung Enterobacterales sind eine große Gruppe innerhalb der Domäne Bacteria. Nach dem phylogenetischen System gehören sie zur Klasse der Gammaproteobacteria in der Abteilung (Divisio, bei den Prokaryoten auch als Phylum bezeichnet) Proteobacteria und bilden dort eine eigene Ordnung.
| Enterobakterien | ||||||||
|---|---|---|---|---|---|---|---|---|
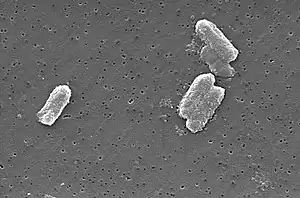
Citrobacter freundii (sekundärelektronenmikroskopisches Bild) | ||||||||
| Systematik | ||||||||
| ||||||||
| Wissenschaftlicher Name | ||||||||
| Enterobacterales | ||||||||
| Adeolu et al. 2016 |
Vor 2016 wurde für diese Bakteriengruppe der Name „Enterobacteriales“ verwendet, der jedoch nach den Regeln des Bakteriologischen Codes (ICBN) nicht gültig war. Weiterhin gab es nur eine Familie – die Enterobacteriaceae – innerhalb der Ordnung. Mit der Etablierung der Ordnung Enterobacterales verbunden ist eine Aufteilung der bisher bekannten Taxa auf mehrere neue Familien.[1] Die neue Systematik führt beispielsweise dazu, dass die Gattungen Escherichia, Morganella und Yersinia drei unterschiedlichen Familien angehören (vgl. Abschnitt Systematik).
Der Name Enterobakterien leitet sich von enteron (altgriechisch ἕντερον ‚Darm‘) ab, weil viele von ihnen typische Darmbewohner sind. Aber auch viele freilebende und ubiquitär vorkommende, nicht darmbewohnende Bakterienarten gehören in diese Ordnung.
Merkmale
Erscheinungsbild
Die Zellen sind stäbchenförmig und gewöhnlich 1 bis 5 µm lang und besitzen einen Durchmesser von etwa 0,5–1,0 µm. Es werden keine Endosporen gebildet. Die meisten können sich mit Flagellen aktiv bewegen, sie sind motil, es kommen jedoch auch Gattungen vor, die sich nicht aktiv bewegen können. Da die Zellwand aus wenigen Mureinschichten und einer zweiten, äußeren Membran aus Phospholipiden und Lipopolysacchariden besteht, sind die Enterobakterien gramnegativ.
Stoffwechsel
Ihr Stoffwechsel ist fakultativ anaerob, daher können sie sowohl über Oxidation unter Anwesenheit von Sauerstoff Stoffe abbauen, als auch unter anaeroben Bedingungen (kein Sauerstoff) Gärung betreiben. Zwei wichtige anaerobe Stoffwechselwege, die zur Unterscheidung der einzelnen Gattungen genutzt werden, sind die 2,3-Butandiol-Gärung und die gemischte Säuregärung (mixed acid fermentation). Bei der gemischten Säuregärung treten als End- und Nebenprodukte vorwiegend Säuren, wie Essigsäure, Milchsäure und Bernsteinsäure (Succinat), aber kein Butandiol auf. Bei der 2,3-Butandiol-Gärung entstehen aus der Gärung von Glucose als End- und Nebenprodukte geringere Mengen von Säuren, aber vor allem in großen Mengen der Alkohol 2,3-Butandiol. Ein weiteres Merkmal der 2,3-Butandiol-Gärung ist das Zwischenprodukt Acetoin und die wesentlich höhere Gasproduktion (CO2). Man findet Butandiolgärung z. B. bei Enterobacter, Klebsiella, Erwinia und Serratia. Gemischte Säuregärung nutzen u. a. Gattungen wie Escherichia, Salmonella und Proteus.
Zur Bestimmung der einzelnen Gattungen wird eine Vielzahl von Diagnosetests genutzt. Zum Beispiel wird mit Hilfe des Voges-Proskauer-Tests das Zwischenprodukt Acetoin der 2,3-Butandiol-Gärung nachgewiesen. Auch der Nachweis des Enzyms β-Galactosidase wird häufig zur Unterscheidung verwendet. Bakterien, die über dieses Enzym verfügen, können das Disaccharid Lactose (Milchzucker) hydrolytisch in die Monosaccharide Glucose und Galactose spalten, um sie im Stoffwechsel zu nutzen.
Phylogenetik
Durch Verwendung phylogenetischer Methoden kann die Stammesgeschichte und die verwandtschaftlichen Beziehungen der Bakterien untereinander geklärt werden. In der 2016 veröffentlichten Beschreibung der Ordnung Enterobacterales werden fünf ‚konservierte charakteristische Indels‘ (engl. conserved signature inserts and deletions, CSI; Näheres dazu im Abschnitt Systematik und Taxonomie) festgelegt, die typisch für die Vertreter der Ordnung sind, aber nicht bei anderen Bakterien vorkommen. Die fünf CSI kommen in den Gensequenzen vor, die für die Proteine L-Arabinose-Isomerase, Elongationsfaktor-P ähnliches Protein YeiP, Peptid-ABC-Transporter Permease, Pyrophosphatase und ein hypothetisches Protein codieren.[1]
Vorkommen

Viele Enterobakterien sind Teil der gesunden Darmflora von Menschen und Tieren; sie kommen jedoch auch überall in der Umwelt vor (Boden, Wasser). Einige sind Krankheitserreger bei Mensch und Tier. Sie kommen vielfach als nosokomiale Erreger vor („Krankenhauskeime“) und befallen Menschen mit schwachem Immunsystem.
Der wahrscheinlich wichtigste Vertreter der Enterobakterien ist Escherichia coli, einer der wichtigsten Modellorganismen der Genetik und Biochemie sowie der Mikrobiologie. Auffällig ist des Weiteren die Gattung Proteus, bei der man das sogenannte „Schwärm-Phänomen“ beobachtet. Wenn sich wachsende Kolonien dieser Bakterien auf einer Agar-Platte ausbreiten, sieht man einen Bakterienrasen mit konzentrischen Ringen.
Systematik und Taxonomie
Verwendete Methoden der phylogenetische Systematik
Bei den Vertretern der „Enterobacteriales“ führten die Ergebnisse phylogenetischer Methoden der letzten Jahre, die insbesondere auf Untersuchung der 16S rRNA beruhten, ein für Prokaryoten typischer Vertreter der ribosomalen RNA, zu keinem zufriedenstellenden Ergebnis. Daher war es das Ziel mehrerer Wissenschaftler (Adeolu et al.) der McMaster University in Ontario, Kanada eine phylogenetische Systematik zu erstellen, die auf Vergleich von genetischen Merkmalen möglichst vieler Vertreter der Gruppe basiert. Zum Zeitpunkt der Untersuchung (2016) waren bereits 14.000 Genome von 54 den Enterobakterien zugehörigen Gattungen in der Genomdatenbank des Nationalen Zentrums für Biotechnologieinformation (NCBI) verfügbar. Für die Analyse wurden 179 repräsentative Genomsequenzen herangezogen und verschiedene molekularer Marker ausgesucht und verglichen, um jeweils einen phylogenetischen Baum zu erstellen, der die evolutionären Beziehungen zwischen den Vertretern der Enterobakterien darstellt.[1]
Zum einen wurden dafür 1548 Hauptproteine herangezogen, die typisch für die Gruppe sind und DNA-Sequenzen verglichen, die für die Proteine codieren. Analog dazu wurden 53 ribosomale Proteine (rProteine) verwendet und weiterhin wurde die DNA-Sequenzanalyse von mehreren Loci (MLSA), die für vier Proteine codieren, durchgeführt. Bei den Proteinen handelt es sich um GyrB (Gyrase B), RpoB, AtpD and InfB. Die so konstruierten drei phylogenetischen Bäume ergeben ein einheitliches Bild mit jeweils sieben Kladen, die als Enterobacter-Escherichia-Klade, Erwinia-Pantoea-Klade, Pectobacterium-Dickeya-Klade, Yersinia-Serratia-Klade, Hafnia-Edwardsiella-Klade, Proteus-Xenorhabdus-Klade und Budvicia-Klade bezeichnet werden. Darauf basierend wird die Ordnung Enterobacterales mit sieben Familien definiert (eine weitere Familie, die Thorselliaceae, wurde durch eine andere Gruppe von Wissenschaftlern erstbeschrieben). Das Ergebnis dieser phylogenetischen Analyse wird durch die Resultate der Analyse der Genomverwandtschaft unterstützt. Diese Methode erfolgt ebenfalls in silico (computerbasiert) als Ersatz für die DNA-DNA-Hybridisierung und ist hilfreich für die Einordnung taxonomisch höherer Rangstufen.[1]
Die Genomanalyse ermöglicht auch das Entdecken bzw. Festlegen von ‚konservierten (bewahrten) molekularen Eigenschaften‘, die sich innerhalb einer Gruppe von nah miteinander verwandten Organismen nicht oder kaum voneinander unterscheiden, während sie sich bei Organismen einer anderen Gruppe durch Evolution deutlich verändert haben. Für die phylogenetische Systematik der Prokaryoten werden dazu ‚konservierte charakteristische Indels‘ (engl. conserved signature inserts and deletions, CSI) verwendet. Gruppenspezifische CSI sind charakteristisch für ein bestimmtes Taxon (beispielsweise eine Ordnung), da sie in allen Vertretern dieser Gruppe und nicht in anderen Gruppen auftreten. Das ursprüngliche Indel eines gruppenspezifischen CSI trat vermutlich bereits vor der Aufspaltung im letzten gemeinsamen Vorfahren auf und wurde von den daraus entstandenen Vertretern der Gruppe übernommen („vererbt“).[2] Ein weiteres wichtiges Ergebnis der Untersuchung von Adeolu et al. ist die Entdeckung von 71 CSI, darunter fünf, die einzigartig für alle Vertreter der Ordnung Enterobacterales sind und daher für ihre Beschreibung verwendet werden. Die übrigen 66 CSI sind spezifisch für die sieben Gruppen und können bei zukünftigen Genomanalysen von neu entdeckten Spezies der Enterobakterien für deren systematische Klassifikation verwendet werden.[1]
| Kladogramm zu den Verwandtschaftsverhältnissen innerhalb der Ordnung Enterobacterales (Stand 2016)[1] | |||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| |||||||||||||||||||||||||||||||||||||||
| Das Kladogramm gibt die Anzahl der dazugehörigen konservierten charakteristischen Indels (CSI) an. Die Familie der Thorselliaceae ist nicht enthalten. |
Das Ergebnis der umfangreichen genomweiten Analysen aus dem Jahr 2016 ist die im Folgenden dargestellte Systematik.
Nomenklatur und Taxonomie der Ordnung
Die Gattung Enterobacter ist nicht die Typusgattung der Familie Enterobacteriacea, dies ist die Gattung Escherichia. Damit wird von der festgelegten Nomenklatur gemäß dem Bakteriologischen Code (International Code of Nomenclature of Bacteria) abgewichen, was 1958 durch Festlegung in der Judicial Opinion 15 der Judicial Commission (in etwa „richterliche oder unparteiische Kommission“) der Internationalen Kommission für die Systematik der Prokaryoten (International Committee on Systematics of Prokaryotes, ICSP) bestätigt wurde.[3] Nach den Regeln des Bakteriologischen Codes müsste eine Ordnung mit der Typusgattung Escherichia folglich „Escherichiales“ genannt werden, eine Ordnung mit der Typusgattung Enterobacter müsste „Enterobacterales“, jedoch nicht „Enterobacteriales“ genannt werden.[4] Um die Akzeptanz der neuen Systematik sicherzustellen und die mögliche Verwirrung bei Benennung der Ordnung als „Escherichiales“ zu vermeiden, wurde der Name Enterobacterales ord. nov. mit der Typusgattung Enterobacter gewählt.[1]
Aktuelle Systematik
Zu der 2016 etablierten Ordnung der Enterobacterales gehören acht Familien mit insgesamt etwa 60 Gattungen.[1][5][6] Die neu festgelegte und damit den Regeln des Bakteriologischen Codes (ICBN) entsprechende Typusgattung der Ordnung ist die Gattung Enterobacter. Die Beschreibung der Ordnung Enterobacterales Adeolu et al. 2016 ord. nov. entspricht der Beschreibung der Familie der Enterobacteriaceae im Bergey's Manual of Systematic Bacteriology von 2005, mit der Erweiterung, dass die Vertreter der Ordnung von allen anderen Bakterien durch fünf CSI unterschieden werden können.[1]
Die Gattung Phytobacter Zhang et al. 2017 emend. Pillonetto et al. 2018 wurde bisher keiner Familie zugeordnet.
Im Folgenden eine Auflistung der zugehörigen Familien mit Nennung den meisten der dazugehörigen Arten (Stand 2019).[6]
- Budviciaceae Adeolu et al. 2016, fam. nov.
- Budvicia Bouvet et al. 1985 emend. Lang et al. 2013, Typusgattung der Familie
- Leminorella Hickman-Brenner et al. 1985
- Pragia Aldová et al. 1988
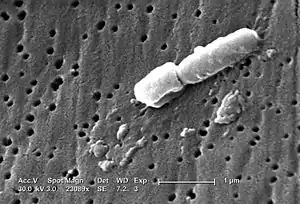
(sekundärelektronenmikroskopische Aufnahme)
- Enterobacteriaceae Rahn 1937 emend. Adeolu et al. 2016
- Biostraticola Verbarg et al. 2008
- Buttiauxella Ferragut et al. 1982
- Cedecea Grimont et al. 1981
- Citrobacter Werkman and Gillen 1932
- Cronobacter Iversen et al. 2008
- Enterobacter Hormaeche and Edwards 1960 emend. Brady et al. 2013
- Escherichia Castellani and Chalmers 1919, Typusgattung der Familie, z. B.: Escherichia coli
- Gibbsiella Brady et al. 2011 emend. Kim et al. 2013
- Klebsiella Trevisan 1885 (Approved Lists 1980) emend. Drancourt et al. 2001, z. B. :Klebsiella pneumoniae
- Kluyvera Farmer et al. 1981
- Leclercia Tamura et al. 1987
- Mangrovibacter Rameshkumar et al. 2010
- Plesiomonas corrig. Habs and Schubert 1962, z. B.: Plesiomonas shigelloides
- Pseudescherichia Alnajar and Gupta 2017
- Raoultella Drancourt et al. 2001
- Saccharobacter Yaping et al. 1990
- Salmonella Lignieres 1900
- Shigella Castellani and Chalmers 1919
- Shimwellia Priest and Barker 2010
- Thorsellia Kämpfer et al. 2006
- Trabulsiella McWhorter et al. 1992
- Yokenella Kosako et al. 1985
- Erwiniaceae Adeolu et al. 2016, fam. nov.
- Hafniaceae Adeolu et al. 2016, fam. nov.
- Edwardsiella Ewing and McWhorter 1965
- Hafnia Møller 1954, Typusgattung der Familie
- Obesumbacterium Shimwell 1963
- Morganellaceae Adeolu et al. 2016, fam. nov.
- Arsenophonus Gherna et al. 1991
- Cosenzaea Giammanco et al. 2011
- Moellerella Hickman-Brenner et al. 1984
- Morganella Fulton 1943, Typusgattung der Familie
- Photorhabdus Boemare et al. 1993
- Proteus Hauser 1885, z. B.: Proteus vulgaris, Proteus mirabilis
- Providencia Ewing 1962
- Xenorhabdus Thomas and Poinar 1979 (Approved Lists 1980) emend. Thomas and Poinar 1983
- Pectobacteriaceae Adeolu et al. 2016, fam. nov.
- Brenneria Hauben et al. 1999 emend. Brady et al. 2012 emend. Brady et al. 2015
- Dickeya Samson et al. 2005
- Lonsdalea Brady et al. 2012
- Pectobacterium Waldee 1945 (Approved Lists 1980) emend. Hauben et al. 1998, Typusgattung der Familie
- Sodalis Dale and Maudlin 1999
- Thorselliaceae Kämpfer et al. 2015, fam. nov.
- Coetzeea Kämpfer et al. 2016
- Yersiniaceae Adeolu et al. 2016, fam. nov.
- Ewingella Grimont et al. 1984
- Rahnella Izard et al. 1981 emend. Brady et al. 2017
- Samsonia Sutra et al. 2001
- Serratia Bizio 1823, z. B.: Serratia marcescens
- Yersinia, van Loghem 1944, Typusgattung der Familie, z. B.: Yersinia pestis, Auslöser der Lungen- und Beulenpest
Einige Synonyme und Umstellungen
- Alle Arten von Levinea Young et al. 1971 wurden zu der Gattung Citrobacter Werkman & Gillen 1932 gestellt
- Verschiedene Erwinia-Arten wurden in die Gattungen Pantoea, Enterobacter, Pectobacterium und Brenneria aufgeteilt
- Liquidobacterium ist ein Synonym für Proteus
Meldepflicht
In Deutschland ist der direkte Nachweis von Enterobacterales namentlich meldepflichtig nach § 7 des Infektionsschutzgesetzes (IfSG), aber nur bei Nachweis einer Carbapenemase-Determinante oder mit verminderter Empfindlichkeit gegenüber Carbapenemen außer bei natürlicher Resistenz. Die Meldepflicht besteht nur bei Infektion oder Kolonisation. (§ 7 Abs. 1 Satz 1 Nr. 52 b) IfSG)
Zudem ist das Auftreten von zwei oder mehr nosokomialen Infektionen nichtnamentlich zu melden, bei denen ein epidemischer Zusammenhang wahrscheinlich ist oder vermutet wird. (§ 6 Absatz 3 IfSG).
Quellen
Literatur
- Volume 6: Proteobacteria: Gamma Subclass. In: Martin Dworkin, Stanley Falkow, Eugene Rosenberg, Karl-Heinz Schleifer, Erko Stackebrandt (Hrsg.): The Prokaryotes. A Handbook on the Biology of Bacteria. 3. Auflage. Springer-Verlag, New York 2006, ISBN 978-0-387-25496-8, doi:10.1007/0-387-30746-X_9.
- Don J. Brenner, J. J. Farmer III: Family I. Enterobacteriaceae. In: George M. Garrity, Don J. Brenner, Noel R. Krieg, James T. Staley (Hrsg.): Bergey's Manual of Systematic Bacteriology: Volume 2: The Proteobacteria, Part B: The Gammaproteobacteria. 2. Auflage. Springer-Verlag, New York 2005, ISBN 978-0-387-95040-2, S. 587–607.
- Michael T. Madigan, John M. Martinko, Jack Parker: Brock – Mikrobiologie, Spektrum Akademischer Verlag, Heidelberg, Berlin 2001, ISBN 3-8274-0566-1
Einzelnachweise
- M. Adeolu, S. Alnajar, S. Naushad, R. S. Gupta: Genome-based phylogeny and taxonomy of the ‘Enterobacteriales’: proposal for Enterobacterales ord. nov. divided into the families Enterobacteriaceae, Erwiniaceae fam. nov., Pectobacteriaceae fam. nov., Yersiniaceae fam. nov., Hafniaceae fam. nov., Morganellaceae fam. nov., and Budviciaceae fam. nov. In: International Journal of Systematic and Evolutionary Microbiology. Nr. 66, Dezember 2016, S. 5575–5599, doi:10.1099/ijsem.0.001485.
- Radhey S. Gupta, Emma Griffiths: Critical Issues in Bacterial Phylogeny. In: Theoretical Population Biology. Nr. 61, Juni 2002, S. 423–434, doi:10.1006/tpbi.2002.1589, PMID 12167362.
- Jean Euzéby, Aidan C. Parte: Genus Enterobacter. In: List of Prokaryotic names with Standing in Nomenclature, Systematik der Bakterien (LPSN). Abgerufen am 20. Dezember 2019.
- S. P. Lapage, P. H. A. Sneath, E. F. Lessel, V. B. D. Skerman, H. P. R. Seeliger, W. A. Clark (Hrsg.): International Code of Nomenclature of Bacteria – Bacteriological Code, 1990 Revision. ASM Press, Washington (DC), USA 1992, ISBN 1-55581-039-X (NCBI Bookshelf).
- P. Kämpfer, S. P. Glaeser, L. K. J. Nilsson, T. Eberhard, S. Håkansson, L. Guy, S. Roos, H.-J. Busse, O. Terenius: Proposal of Thorsellia kenyensis sp. nov. and Thorsellia kandunguensis sp. nov., isolated from larvae of Anopheles arabiensis, as members of the family Thorselliaceae fam. nov. In: International Journal of Systematic and Evolutionary Microbiology. Nr. 65, Februar 2015, S. 444–451, doi:10.1099/ijs.0.070292-0.
- Jean Euzéby, Aidan C. Parte: Classification of domains and phyla - Hierarchical classification of prokaryotes (bacteria). In: List of Prokaryotic names with Standing in Nomenclature, Systematik der Bakterien (LPSN). Abgerufen am 20. Dezember 2019.