Rekombination (Genetik)
Unter Rekombination versteht man in der Biologie die Neuanordnung (Re-) von genetischem Material (DNA, RNA) in den Zellen und im engeren Sinne den Austausch von Allelen. Durch Rekombination kommt es zu neuen Gen- und Merkmalskombinationen. Rekombination und Mutation verursachen die genetische Variabilität innerhalb einer Population. Die genetische Variabilität ist wiederum die Basis für die Anpassung an wechselnde Umweltbedingungen im Mikroevolutionsprozess.
Rekombination durch sexuelle Fortpflanzung
Bei der sexuellen Fortpflanzung von Eukaryoten wie Pflanzen und Tieren gibt es einen Kernphasenwechsel, d. h. einen periodischen Wechsel zwischen einer haploiden und einer diploiden Phase bei der Fortpflanzung von einer Generation zur nächsten. Die sexuelle Rekombination betrifft bei diesen Arten das gesamte Genom und ist damit die tiefgreifendste Form der Rekombination. Hierbei sind zwei Weisen der Rekombination zu unterscheiden:
- Interchromosomale Rekombination, durch Neukombination ganzer Chromosomen im Chromosomensatz.
- Intrachromosomale Rekombination, durch Neukombination von Allelen innerhalb von Chromosomen infolge Crossing-over bei der 1. Reifeteilung.
Auf beide Weisen wird bei sexueller Fortpflanzung die genetische Variation der folgenden Generation beträchtlich erhöht, indem einzelne Abschnitte des Genoms in verschiedenen Kombinationen mit anderen auftreten. Bezogen auf die Population einer Art können dabei möglicherweise vorteilhafte Kombinationen ebenso entstehen wie womöglich abträgliche. Doch verglichen mit der asexuellen Fortpflanzung ist damit auch eine höhere Anpassungsrate möglich, ein beschleunigter Anpassungsvorgang der Population. Das gilt auch, wenn eine Mutation auftritt, denn diese bleibt nicht in einer genetischen Kombination gebunden. Daher verschwindet sie als unvorteilhafte rascher oder verbreitet sich als zuträgliche schneller als bei einer asexuellen Vermehrung, mit einer Dynamik, die sich auf molekularer Ebene widerspiegelt.[1]
Interchromosomale Rekombination
Für die interchromosomale Rekombination lassen sich dabei zwei Phasen unterscheiden:
- Die Verteilung der Chromosomen bei der Meiose auf (haploide) Keimzellen.
- Die Verschmelzung der Keimzellen zur (diploiden) Zygote bei der Befruchtung.
Die Zahl an interchromosomalen Rekombinationsmöglichkeiten ist abhängig von der Anzahl der Chromosomen. So sind beispielsweise für einen aus 2 Paaren homologer Chromosomen (etwa: 1a1b, 2a2b) bestehenden Chromosomensatz mehrere verschiedene Aufteilungen in einfache Chromosomensätze möglich (1a,2a; 1a,2b; 1b,2a; 1b,2b), hier denn 22 (= 4).
Bei einem Satz mit 23 Chromosomenpaaren wie beim Menschen sind 223 (= 8.388.608) mögliche Kombinationen gegeben, haploide Keimzellen interchromosomal verschieden auszubilden. Da bei der Befruchtung zwei Geschlechtszellen miteinander verschmelzen, ergeben sich für die Nachkommenschaft eines Menschenpaares theoretisch 223 · 223 = 246 (≈ 70 Billionen) Möglichkeiten bezüglich der Neukombination von Chromosomen euploider Chromosomensätze. Schon aufgrund dieser interchromosomalen Rekombination sind bei der sexuellen Fortpflanzung des Menschen zwei genetisch identische Nachkommen nahezu unmöglich, wenn es nicht eineiige Mehrlinge sind.[2]
Die interchromosomale Rekombination bei Viren mit einem segmentierten Genom wird dort als Reassortment bezeichnet, beispielsweise bei den Influenzaviren. Sie setzt eine Doppel- oder Mehrfachinfektion voraus.
Intrachromosomale Rekombination
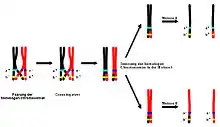
Zusätzlich sind intrachromosomale Rekombinationen möglich, indem zwischen gepaarten Chromatiden Abschnitte gegeneinander ausgetauscht werden. Anfangs der 1. meiotischen Teilung (im Zygotän der Prophase der Reduktionsteilung) lagern sich die bereits verdoppelten homologen Chromosomen paarweise aneinander und bilden so Homologenpaare. Die beiden Chromosomen liegen mit je zwei Schwester-Chromatiden vor und bilden gepaart eine Einheit aus vier Chromatiden, eine sogenannte Tetrade. Ihre gegenseitig genaue Zuordnung vermittelt ein reißverschlussähnlich verbindender synaptonemaler Komplex als temporäres inneres Proteingerüst (im Pachytän dieser Prophase I). In diesem Stadium kann es an Stellen engen Kontakts – unter Ausbildung von Rekombinationsknoten – zur wechselseitigen Anlagerung von Nicht-Schwesterchromatiden kommen und sich ein Crossover ereignen. Solche Bereiche fallen später als kreuzweise Überlagerung, Chiasma genannt, auf (im Diplotän von Prophase I).
Meiosen können achiasmatisch verlaufen, in den meisten Fällen tritt jedoch bei fast jeder Tetrade mindestens ein Chiasma auf, oft mehrere. Bei der (in Anaphase I) folgenden Trennung der gepaarten Chromosomen werden die Chiasmata derart aufgelöst, dass in den Chromatiden nun Abschnitte gegeneinander ausgetauscht sind. Diese Chromatiden tragen also nicht mehr allein von einem Elternteil vererbte Allele, sondern sowohl mütterlicherseits als auch väterlicherseits geerbte Anteile. Damit können sie eine neue Kombination von Genvarianten innerhalb eines Chromosoms darstellen.
Die intrachromosomale Rekombination infolge eines einzigen Crossovers in einer Tetrade kann so zu vier Chromatiden mit je unterschiedlicher Kombination von Allelen führen. Die vordem als Schwester-Chromatiden eines X-förmigen Chromosoms einander gleichen sind dann verschieden. Sie bleiben im weiteren Verlauf der 1. meiotischen Teilung am Centromer verbunden und werden so gemeinsam einem der beiden Tochterkerne zugeteilt. Die Paare homologer Chromosomen werden mittels Fasern des Spindelapparats auseinandergezogen und die gepaarten Nicht-Schwester-Chromatiden voneinander getrennt. Der Aufteilung in Zellkerne folgt die Teilung der Zelle in zwei Tochterzellen. Aus der anschließenden 2. meiotischen Teilung (Äquationsteilung) beider Zellen gehen schließlich vier haploide Zellen hervor, ihr jeweiliger Chromosomensatz besteht aus je einer der vier Chromatiden einer jeden Tetrade und ist daher genetisch verschieden.
Da die Anzahl der Crossover-Ereignisse wie auch deren Orte von Meiose zu Meiose variieren, lässt sich die Zahl der intrachromosomalen Rekombinationsmöglichkeiten nicht genau angeben. Nimmt man für jede der Tetraden nur ein Crossover an, so ergeben sich bei einem Chromosomensatz wie dem menschlichen bereits 423 (≈ 70 Billionen) Möglichkeiten bezüglich der Neukombination von Allelen im einfachen (haploiden) Chromosomensatz. Ohne den Austausch von Abschnitten homologer Chromosomen hingegen würde die intrachromosomale Kombination jeweils unverändert bleiben. In diesem Fall gäbe es dann allein ≈ 8,4 Millionen Möglichkeiten durch interchromosomale Rekombination, individuell verschiedene Eizellen oder Samenzellen zu bilden (siehe oben).[2]
Intrachromosomale (homologe) Rekombination wird ebenfalls bei Viren beobachtet (z. B. Coronaviren). Auch hier ist Doppel- oder Mehrfachinfektion mit genetisch ähnlichen Virusstämmen die Voraussetzung.
Rekombination durch parasexuelle Prozesse
Parasexualität tritt bei Bakterien und einigen Pilzen auf. Dabei findet entweder ein Transfer von Teilen des Genoms statt, oder es fusionieren Zellen, die auf nichtgeschlechtlichem Weg entstanden sind (vegetative Zellen). Ein Transfer von Genomteilen kann durch folgende Prozesse stattfinden:
- Konjugation, einem direkten Transfer genetischen Materials zwischen zwei miteinander verbundenen Zellen.
- Transduktion, einem Transfer mit Hilfe von Viren.
- Transformation, durch Aufnahme und Integration von extrazellulärer DNA in das Genom einer Zelle.
Rekombination bei Viren
Auch bei Viren gibt es molekularbiologische Phänomene, die die evolutionsbiologische Wirkung von Rekombination haben. Entscheidend hierfür ist, ob Segmente des Virus-Genoms nebeneinander vorliegen beziehungsweise aus wie vielen Nukleinsäuremolekülen (DNA und/oder RNA) es besteht. Diese Genom-Segmente entsprechen dabei funktionell den Chromosomen der Eukaryonten und werden daher manchmal auch so bezeichnet.

Manche Viren, darunter Influenzavirus A, der Erreger der echten Grippe, haben ein segmentiertes Genom aus mehreren Nukleinsäure-Molekülen (multipartit). So besteht das Genom von Influenzavirus A aus 8 Segmenten (langkettigen RNA-Molekülen). Wenn nun eine Zelle gleichzeitig von Viren verschiedener Influenza-A-Stämme infiziert wird (Mehrfachinfektion), können sich Viruspartikel bilden, die einige Stränge des einen Stammes und einige Stränge des anderen Stammes enthalten. Insgesamt können so verschiedene Varianten gebildet werden. Dieses Phänomen wird mit dem englischen Ausdruck als Reassortment bezeichnet und stellt bei Viren das Analogon zur interchromosomalen Rekombination dar.
Es gibt aber auch Viren, deren Genom zwar nur aus einem einzigen DNA- oder RNA-Molekül besteht (unsegmentiert oder monopartit), die aber im Fall einer Mehrfachinfektion dennoch gelegentlich rekombinieren können, durch Brüche der Nukleinsäureketten. Ein solcher Vorgang wird sowohl für die Entstehung des Erregerstamms von SARS (SARS-CoV[-1]) angenommen wie auch für den nekrotisch wirkenden Stamm PVYNTN von Kartoffelvirus Y ([en]) vermutet und stellt das virale Gegenstück zur intrachromosomalen Rekombination (Crossing-over) dar.
Somatische Rekombination
Bei Eukaryoten ist Rekombination nicht auf die Meiose und die Keimzellen beschränkt. Auch in somatischen Zellen kann es zu einer DNA-Umgruppierung („DNA-Rearrangement“) kommen. Dieses wirkt sich auf die Genexpression aus. Als Beispiele seien Transposons („springende Gene“) und die somatische Rekombination der Immunglobuline genannt, siehe V(D)J-Rekombination.
Homologe und nicht homologe Rekombination
Homologe Rekombination
Die homologe Rekombination (HR) tritt bei allen Organismen auf. Voraussetzung sind homologe, doppelsträngige DNA-Abschnitte. Homolog heißt, dass es große Ähnlichkeiten in der Nucleotidsequenz gibt. Bei Doppelstrangbrüchen kann durch homologe Rekombination der Schaden ausgebessert werden, indem die Informationen auf dem unbeschädigten Chromatid als Vorlage genutzt wird. HR ist also ein Werkzeug der Zelle, um Genmutationen zu reparieren. Homologe Rekombinationen laufen meist nach folgendem Schema ab:
- Parallele Annäherung („Paarung“) zweier doppelsträngiger DNA-Moleküle, so dass die Bereiche ähnlicher (homologer) Nucleotidsequenzen auf gleicher Höhe liegen.
- In einem komplexen Vorgang kann es nun zu einem Crossing-over kommen. Dabei werden DNA-Abschnitte zwischen den beiden „gepaarten“ DNA-Molekülen ausgetauscht.
- Die Stelle, an der die ausgetauschten DNA-Abschnitte neu verknüpft werden, kann irgendwo innerhalb der homologen Nukleotidsequenzen liegen.
- Der Bruch und die Wiederverbindung der DNA-Moleküle erfolgt durch spezifische Enzyme, die sog. Rekombinasen, so präzise, dass kein Nucleotid verloren geht oder dazukommt.
Im Verlauf der HR tritt die sogenannte Holliday-Struktur auf.
Das Verhältnis von Homologer Rekombination (HR) zu Nichthomologer Rekombination kann in verschiedenen Spezies um mehrere Größenordnungen variieren. So gibt es innerhalb der Pflanzen vor allem beim Laubmoos Physcomitrella patens eine so hohe HR-Rate, dass Gene gezielt ausgeschaltet werden können, um so ihre Funktion zu analysieren[4]. Diese Technik nennt man Gene-Targeting „(englisch gene targeting)“, den methodischen Ansatz nennt man „Reverse Genetik“.
Sequenzspezifische Rekombination
Eine gezielte (also nicht zufällige) Integration von DNA in ein Genom kann auch noch durch die sequenzielle Rekombination erfolgen. Diese nicht homologe Rekombination wird durch ein Enzym bewerkstelligt, wie es z. B. vom Bakteriophagen λ codiert wird, die sogenannte Integrase. Die Integrase bringt zwei nicht homologe Sequenzen zweier DNA-Moleküle zusammen, katalysiert deren Spaltung und verbindet sie miteinander. So kann etwa ein Virengenom an einem vorgesehenen Ort in ein Chromosom eingebaut werden.
Rekombination in der Gentechnik
In der Gentechnik stehen heute Werkzeuge zur Verfügung, mit deren Hilfe rekombinante DNA künstlich hergestellt und in Organismen eingeschleust werden kann. Im Zuge einer Klonierung wird meist DNA mit Restriktionsenzymen an spezifischen Erkennungssequenzen geschnitten und mit Ligasen neu verknüpft. Häufig dienen Plasmide oder Viren als Vektoren, um die rekombinante DNA in den Zielorganismus zu transferieren.
Eine neuartige Alternative zur konventionellen DNA-Klonierung mit Restriktionsenzymen und Ligasen sind auf homologer Rekombination basierende Technologien, wie das Recombineering und das RMCE-Kassettenaustauschverfahren.
Literatur
- Bruce Alberts, Lutz Nover: Lehrbuch der Molekularen Zellbiologie. 3. Auflage, Wiley-VCH, Weinheim 2005, ISBN 3-527-31160-2.
- Neil A. Campbell, Jane B. Reece, Jürgen Markl: Biologie. 6. Auflage, Spektrum Akademischer Verlag, Heidelberg u. a. 2003, ISBN 3-8274-1352-4.
- Elisabeth Günther, Eike Libbert u. a.: Allgemeine Biologie (= UTB für Wissenschaft. Uni-Taschenbücher. Band 1197). 6., durchgesehene Auflage, Fischer, Stuttgart 1988, ISBN 3-437-20387-8.
Siehe auch
- Mit Attached-X-Chromosomen bei Drosophila melanogaster kann die Rekombination direkt untersucht werden.
- Knockout-Moos
- Reassortment (Reassortierung) bei Viren
Weblinks
- Homologous Recombination, Tempy & Trun – Animation über den molekularbiologisch komplexen Vorgang der homologen Rekombination. Blackwell Science Publishers.
Einzelnachweise
- Michael J. McDonald, Daniel P. Rice, Michael M. Desai: Sex speeds adaptation by altering the dynamics of molecular evolution. In: Nature. 2016, doi:10.1038/nature17143.
- Wilfried Janning, Elisabeth Knust: Genetik: Allgemeine Genetik – Molekulare Genetik – Entwicklungsgenetik. 2. Auflage. Georg Thieme, Stuttgart 2008, ISBN 978-3-13-151422-6, S. 37 f.
- C. M. Deeg, E. C.T. Chow, C. A. Suttle: The kinetoplastid-infecting Bodo saltans virus (BsV), a window into the most abundant giant viruses in the sea. In: eLife. 7, 2018, S. e33014. doi:10.7554/eLife.33014.
- Ralf Reski: Physcomitrella and Arabidopsis: the David and Goliath of reverse genetics. In: Trends in Plant Science. 3, 1998, S. 209, doi:10.1016/S1360-1385(98)01257-6.