Lysindecarboxylase
Lysindecarboxylase (LDC) ist ein Enzym, das hauptsächlich in Bakterien und auch in Eukaryoten vorkommt.[1] LDC katalysiert den ersten Schritt der Chinolizidin-Alkaloid-Biosynthese, bei der Alkaloide mit Chinolizidin-Grundstruktur synthetisiert werden, insbesondere Lupinen-Alkaloide.
| Lysindecarboxylase | ||
|---|---|---|
| ||
| Bändermodell der konstitutiven Lysindecarboxylase von E. coli, nach PDB 5FKZ | ||
| Bezeichner | ||
| Externe IDs | ||
| Enzymklassifikation | ||
| EC, Kategorie | 4.1.1.18, Lyase | |
| Reaktionsart | Decarboxylierung | |
| Substrat | L-Lysin | |
| Produkte | 1,5-Diaminopentan + CO2 | |
| Vorkommen | ||
| Übergeordnetes Taxon | Bakterien, Eukaryoten | |
Chinolizidinalkaloid-Biosynthese
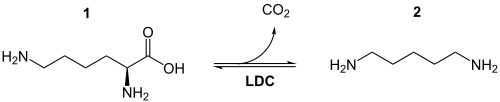
Der erste Schritt der Chinolizidinalkaloid-Biosynthese besteht aus der Abspaltung von Kohlenstoffdioxid (Decarboxylierung) aus L-Lysin (1) mittels der Lysindecarboxylase zu Cadaverin (2), auch als 1,5-Diaminopentan bekannt.[2]
Danach wird Cadaverin mithilfe des Enzyms Diaminoxidase zu 3-Aminopropanal umgewandelt.[3] Erst durch die spontane Cyclisierung des 3-Aminopropanals zu 1-Piperidein kann die Synthese verschiedener Chinolizidinalkaloide erfolgen. Anschließend erfolgt die Modifikation der Alkaloide durch Dehydrierung, Oxygenierung oder Veresterung. Die Bildung von Chinolizidinalkaloidestern markiert das Ende der Biosynthese und die Endprodukte stellen ebenfalls eine Speicherform für verschiedene Organismen dar.[2]
Unter den Chinolizidinalkaloiden unterscheidet man zwischen Lupinenalkaloiden, die aus der Aminosäure L-Lysin hervorgehen und insbesondere in Lupinenarten (z. B. Lupinus angustifolius), aber auch im Besenginster, Stechginster und Goldregen vorkommen,[4] und Nupharalkaloiden (aus Nuphar), die auf dem Terpenweg gebildet werden.[5]
Bekannte Vertreter der Lupinenalkaloide sind zum Beispiel (−)-Lupinin, (+)-Epilupinin, (+)-Multiflorin, (+)-Lupanin, (+)-Matrin, (−)-Spartein und (+)-Cytisin.[6]
Nachweis des bakteriellen Enzyms
Zahlreiche Bakterienarten verfügen über die Lysindecarboxylase. Der Nachweis des Enzyms in Vertretern der gramnegativen Enterobakterien dient zur Differenzierung und ist Bestandteil einer Bunten Reihe zur Bestimmung der Gattung oder Art.[7] Das Testverfahren wurde 1955 eingeführt[8] und ist seit den 1970er Jahren Bestandteil von miniaturisierten Testsystemen (z. B. im API 20 E-System).[9]
Für den Nachweis des bakteriellen Enzyms – oft als LDC abgekürzt – wird das standardisierte, lysinhaltige Nährmedium mit Bakterienmaterial beimpft und unter anoxischen Bedingungen inkubiert.[7] Um den Zutritt von Sauerstoff in das Teströhrchen zu verhindern, wird der inokulierte Ansatz entweder mit Paraffinöl oder mit Mineralöl überschichtet, dies verhindert falsch-positive Ergebnisse.[10] Durch die Bildung des Diamins Cadaverin steigt der pH-Wert im Testmedium, die Auswertung erfolgt anhand des Farbumschlags des im Nährmediums integrierten pH-Indikators.[7] Für die optimale Enzymaktivität ist ein pH-Wert unter 5,5 erforderlich (saurer Bereich), während Nährmedien üblicherweise einen neutralen pH-Wert aufweisen.[11]
Verschiedene Testmedien
Bezüglich der Zusammensetzung des Differenzierungsmediums wie des verwendeten pH-Indikators gibt es Unterschiede:
In dem zuerst entwickelten Nährmedium nach Møller wird neben Lysin auch D-Glucose eingesetzt, sowie Bromkresolpurpur als pH-Indikator, der pH-Wert wird auf 6,0 eingestellt. Die Bakterien verwerten zunächst den geringen Glucose-Anteil in einer Gärung, wobei Säuren entstehen (vergleiche Gemischte Säuregärung), wodurch der pH-Wert unter 5,5 gesenkt wird. Die dann einsetzende Reaktion der LDC alkalisiert das Testmedium und Bromkresolpurpur zeigt dies durch Farbumschlag von Gelb nach Purpur an. Es ist immer ein Vergleichsröhrchen mitzuführen, das kein Lysin enthält, damit die anfängliche Säurebildung überprüft werden kann.[11] Ein Nachteil ist, dass der Ansatz über vier Tage inkubiert werden muss, bevor die Auswertung erfolgen kann.[10] Eine Abwandlung dieses Differenzierungsmediums stellt der Lysin-Eisen-Agar dar, mit dem zusätzlich noch die Bildung von Schwefelwasserstoff durch die Bakterien überprüft werden kann.[11]
Als schnellere Variante gilt eine Methode, ähnlich zu der beim Nachweis der Ornithindecarboxylase verwendeten. Hierbei enthält das Testmedium keine Glucose, neben Lysin werden noch Peptone und Hefeextrakt eingesetzt, sowie Bromthymolblau als pH-Indikator, der pH-Wert wird auf 5,2–5,4 eingestellt. Das flüssige Nährmedium wird mit reichlich Bakterienmaterial inokuliert und für vier Stunden inkubiert. Falls die Bakterien Lysindecarboxylase besitzen, wird das Testmedium durch das Reaktionsprodukt alkalisiert und Bromthymolblau zeigt dies durch Farbumschlag von Gelb nach Blau an. Auch eine grüne Färbung (pH-Wert knapp unter 7,0) wird als positiv gewertet.[10]

Das Substrat des LDC-Tests im API 20 E-Systems enthält ebenfalls keine Glucose, als pH-Indikator dient Phenolrot und der ursprüngliche pH-Wert ist auf 6,2 eingestellt. Hierbei soll eine Inkubationsdauer von 18 bis 24 Stunden eingehalten werden, bevor man die Lysindecarboxylase-Reaktion beurteilt. Die Alkalisierung führt zum Farbumschlag von Gelb nach Rot, auch eine orange Färbung (pH-Wert knapp über 7,0) ist als positiv zu werten. Wird dies beachtet, ergibt sich eine Übereinstimmung von 98 % mit dem Verfahren nach Møller.[9]
Beispiele für LDC-positive und LDC-negative Bakterien
Der Nachweis der bakteriellen Lysindecarboxylase ist für die Unterscheidung der Enterobakterien von Bedeutung. Vertreter der Gattungen Edwardsiella, Hafnia und Salmonella (außer Salmonella enterica subsp. enterica ser. Paratyphi) verfügen über dieses Enzym. Hingegen sind Vertreter der Gattungen Cedecea, Citrobacter, Moellerella, Proteus, Providencia, Rhanella, Shigella, Yersinia (außer Yersinia ruckeri) und die Spezies Cronobacter sakazakii LDC-negativ. Innerhalb der Gattungen Enterobacter, Escherichia, Klebsiella, Morganella und Serratia gibt es LDC-positive und -negative Vertreter, zu deren Unterscheidung der Nachweis der Lysindecarboxylase-Reaktion beiträgt.[12]
Einzelnachweise
- UniProtKB results. In: UniProtKB. Abgerufen am 31. Dezember 2019.
- Somnuk Bunsupa, Mami Yamazaki, Kazuki Saito: Quinolizidine alkaloid biosynthesis: recent advances and future prospects. In: Frontiers in Plant Science. Band 3, 2012, doi:10.3389/fpls.2012.00239.
- Quinolizidine Alkaloids. In: Biocyclopedia. Abgerufen am 2. Januar 2020.
- Lupinenalkaloide. In: Lexikon der Biologie. Spektrum, abgerufen am 2. Januar 2020.
- Chinolizidinalkaloide. In: Lexikon der Biochemie. Spektrum, abgerufen am 2. Januar 2020.
- Ian Bass Seiple: The Lupin Alkaloids. (PDF) In: https://www.scripps.edu/. Abgerufen am 2. Januar 2020.
- Roland Süßmuth, Jürgen Eberspächer, Rainer Haag, Wolfgang Springer: Biochemisch-mikrobiologisches Praktikum. 1. Auflage. Thieme Verlag, Stuttgart/New York 1987, ISBN 3-13-685901-4, S. 78–85.
- Vagn Møller: Simplified tests for some amino acid decarboxylases and for the arginine dihydrolase system. In: Acta pathologica et microbiologica Scandinavica. Band 36, Nr. 2, 1955, S. 158–172, doi:10.1111/j.1699-0463.1955.tb04583.x, PMID 14375937.
- P. B. Smith, K. M. Tomfohrde, D. L. Rhoden, A. Balows: API system: a multitube micromethod for identification of Enterobacteriaceae. In: Applied Microbiology. Band 24, Nr. 3, September 1972, S. 449–452, PMID 4562482, PMC 376540 (freier Volltext).
- D. C. Brooker, M. E. Lund, D. J. Blazevic: Rapid test for lysine decarboxylase activity in Enterobacteriaceae. In: Applied Microbiology. Band 26, Nr. 4, Oktober 1973, S. 622–623, PMID 4751806, PMC 379861 (freier Volltext).
- Elmer W. Koneman: Koneman's Color Atlas and Textbook of Diagnostic Microbiology. Lippincott Williams & Wilkins, 2006, ISBN 0-7817-3014-7, S. 225–226 (eingeschränkte Vorschau in der Google-Buchsuche).
- J. J. Farmer III, B. R. Davis u. a.: Biochemical identification of new species and biogroups of Enterobacteriaceae isolated from clinical specimens. In: Journal of Clinical Microbiology. Band 21, Nr. 1, Januar 1985, S. 46–76, PMID 3881471, PMC 271578 (freier Volltext).