MALDI-TOF
MALDI-TOF ist eine Methode der Massenanalyse von chemischen Verbindungen. Das Verfahren kombiniert die Matrix–Assistierte Laser–Desorption–Ionisierung (MALDI) mit der Flugzeitanalyse (engl. time of flight, TOF) freigesetzter Ionen zur Massenspektrometrie.
Geschichte

Das Akronym MALDI, geprägt 1985 von Michael Karas, Doris Bachmann und Franz Hillenkamp, bedeutet Matrix-assistierte Laser-Desorption-Ionisierung.[1] Sie fanden, dass die Aminosäure Alanin zu ionisieren war, wenn man die Aminosäure Tryptophan dazugab. Denn nur Tryptophan absorbiert die Energie eines Pulslasers bei 266 nm und hilft, das nicht absorbierende Alanin zu ionisieren. Einen entscheidenden Fortschritt erzielte die Gruppe um Koichi Tanaka, als sie winzige Kobalt-Partikel (30 nm) zu Glyzerin mischten und einen preiswerten Stickstofflaser (337 nm) einsetzte. Mit solcher Matrix-Laser-Kombination ließen sich große Biomoleküle wie die Carboxypeptidase-A ionisieren. Im Jahr 2002 erhielt Tanaka ein Viertel des Nobelpreises für Chemie.[2][3]
Anfang der 1990er Jahre kamen erste kommerzielle Instrumente auf den Markt und machten MALDI einer breiten Anwendung und weiterer Forschung zugänglich.[4][5]
Funktionsweise
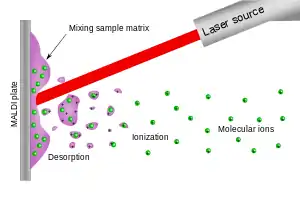
MALDI ist ein dreistufiger Prozess:
- Die Probe wird mit unterstützendem Matrix-Material versehen und auf einem Träger fixiert.
- Ein gepulster Laser-Strahl löst Moleküle als heißes Gas aus der Probe; dieser Vorgang wird Ablation bzw. Desorption genannt.
- Die Abgabe oder Annahme von Protonen wandelt die befreiten Moleküle zu Ionen.[6]
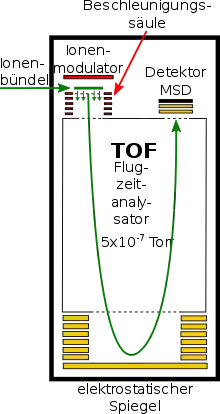
Schließlich werden die Ionen in einem Massenspektrometer beschleunigt und ihre jeweilige TOF registriert.
MALDI-TOF dient zur Analyse großer Moleküle und (Bio-)Polymere. Die Moleküle einer Probe werden zuerst ionisiert (MALDI) und dann mittels Massenspektroskopie entsprechend ihrer Flugzeit (TOF) analysiert.
Bei MALDI ionisiert man den Analyten mittels Laserbeschuss über eine Matrix, in welche die eigentlichen Analyten eingebettet sind. Für eine genauere Erläuterung dieses Schritts siehe Matrix-unterstützte Laser-Desorption/Ionisation (MALDI). Die entstandenen Ionen werden in einem elektrischen Feld beschleunigt. Typische Beschleunigungsspannungen hierfür liegen bei 10–30 kV. Wegen der gepulsten Ionenerzeugung bietet sich für die Massenanalyse ein TOF-Analysator (Time-of-flight Massenspektrometer, teilweise mit Reflektron) an.
Die Flugzeit () der Ionen ist folgendermaßen von ihrer Masse () und ihrer Ladungszahl () abhängig:
Ein Ionendetektor (oft ein Sekundärelektronenvervielfacher wie etwa eine Mikrokanalplatte) wandelt die ankommenden Ionen in ein elektrisches Signal um. Der Vorteil dieser Messmethode ist die schnelle Analyse eines vergleichsweise großen Massenbereichs. Bemerkenswert ist, dass der TOF-Analysator keine prinzipielle Massenbegrenzung zu großen Massen hin besitzt, so dass schon Ionen von m/z bis zu 1.000.000 gemessen wurden. Als ionisierte Spezies treten vor allem die Pseudomolekülionen [M+H]+, [M+Na]+ oder auch [M+K]+ auf. Das Auftreten (bzw. die relativen Intensitäten) der unterschiedlichen Spezies hängt jedoch stark von der Vorgeschichte der Probe sowie von der Substanzklasse an sich ab.
Da Membranproteine oder Lipide ohne Tenside nicht wasserlöslich sind und Tenside bei der MALDI-TOF-MS stören,[7] werden teilweise spaltbare Tenside verwendet. Spaltbare amphiphile Derivate von α-Cyano-4-hydroxyzimtsäure (CHCA), die über Formaldehyd an Decanol gekoppelt wurden, müssen dagegen nicht entfernt werden, da sie gleichzeitig als Matrix in der MALDI-TOF-MS verwendet werden können. Ebenso kann eine Matrix aus α-Cyano-4-hydroxyzimtsäure mit dem Tensid CTAB bei der MALDI-TOF-MS verwendet werden.[8] Es gibt Matrix-Mischungen, die weniger empfindlich gegenüber Tensiden sind.[9]
Anwendung

Von der Matrix abgesehen, ist MALDI-TOF eine markierungsfreie Methode, bei der keine Farbstoffe, Antikörper oder radioaktive Elemente benötigt werden. Deswegen steht sie einem breiten Anwendungsgebiet zur Verfügung, nicht nur der technischen Materialkontrolle, sondern auch biologischen und medizinischen Fragen.[10]
In der Bakteriologie kann die Technologie zur Identifizierung der Spezies verwendet werden. Messungen von isolierten Reinkulturen gleicht man mittels Referenzspektren aus einer Datenbank ab.[11][12] Im Vergleich zu molekularbiologischen Methoden wie der 16S rRNA- oder ITS-Sequenzierung sind die laufenden Kosten deutlich geringer, weshalb das Verfahren auch verstärkt in der Lebensmittelmikrobiologie eingesetzt wird, um die Produktsicherheit und Betriebshygiene zu erhöhen.[13] Für hochpathogene Mikroorganismen werden die Geräte in Laboren mit entsprechender biologischer Schutzstufe aufgestellt und die Kulturen vorher sicher inaktiviert.[14]
Die Behördliche Lebensmittelüberwachung in Deutschland arbeitet in Kooperation mit Handelslaboratorien und medizinischen Versorgungszentren an der Erstellung einer Austauschplattform von Spektrenbibliotheken, um den wissenschaftlichen Austausch zu fördern.[15] Neben Mikroorganismen ist es Lebensmittelchemikern des CVUA Stuttgart gelungen, auch eine Methode zur Differenzierung von Säugetier-Spezies zu entwickeln, um Lebensmittelbetrug in der Gastronomie aufzudecken.[16] Eine Identifizierung von Tierarten ist auch in Milchprodukten wie Mozzarella und Feta möglich.[17] Die Technik lässt sich auf die Authentifizierung von Fisch übertragen.[18]
Bei der Diagnose von Tumoren mit dem Mikroskop sind bösartige von normalen Zellen zu unterscheiden. MALDI-TOF könnte auf Grund des Proteinmusters auf die Aggressivität einer Geschwulst oder auf ihre Empfindlichkeit gegenüber einer Therapie hinweisen. Die Methode unterstützt die Tumor-Typisierung und mag zum objektiven Grading beitragen. Für die morphologische Zuordnung des MALDI-Bildes bedarf es eines gefärbten Bildes von demselben Gewebeschnitt. Die genaue Überlagerung zweier Pixel-Dateien gehört in der digitalen Mikroskopie zur Routine.[19][20][21]
In der Kunststoffanalytik kann die Molmasse sowie der Molmassenverteilung mittels MALDI-TOF MS überprüft werden.[22]
Literatur
- Franz Hillenkamp, M Karas. Mass spectrometry of peptides and proteins by matrix-assisted ultraviolet laser desorption/ionization. In: Methods Enzymol 193/1990: 280–295. doi:10.1016/0076-6879(90)93420-P.
- Franz Hillenkamp, Anthony Tsarbopoulos, Michael L Gross. Focus on desorption ionization and macromolecular mass spectrometry. In: J Am Soc Mass Spectrom 19/2008: 1041–1044. doi:10.1016/j.jasms.2008.06.017.
Weblinks
Einzelnachweise
- Michael Karas, Doris Bachmann, Franz Hillenkamp. Influence of the wavelength in high-irradiance ultraviolet laser desorption mass spectrometry of organic molecules. In: Analytical Chem 57/1985: 2935–2939. doi:10.1021/ac00291a042
- Koichi Tanaka, Y. Ido, S. Akita, Y. Yoshida, T. Yoshida: Detection of high mass molecules by laser desorption time-of-flight mass spectrometry. In: Proceedings of the Second Japan-China Joint Symposium on Mass Spectrometry 1987: 185–188.
- nobelprize.org: Koichi Tanaka - Facts, abgerufen am 27. Februar 2017.
- Michael Karas, U. Bahr. Laser desorption ionization mass spectrometry of large biomolecules. In: Trends Analyt Chem 9/1990: 321–325. doi:10.1016/0165-9936(90)85065-F
- Michael Karas, U. Bahr. Laser desorption ionization mass spectrometry of bioorganic molecules. In: Methods Mol Biol 17/1993: 215–228. doi:10.1385/0-89603-215-9:215
- Michael Karas, Ralf Krüger: Ion formation in MALDI: The cluster ionization mechanism. In: Chem Rev 103/2003: 427–440. doi:10.1021/cr010376a.
- J. L. Norris, N. A. Porter, R. M. Caprioli: Mass spectrometry of intracellular and membrane proteins using cleavable detergents. In: Analytical chemistry. Band 75, Nummer 23, Dezember 2003, S. 6642–6647. doi:10.1021/ac034802z. PMID 14640740.
- R. Gottardo, A. Chiarini, I. Dal Prà, C. Seri, C. Rimondo, G. Serpelloni, U. Armato, F. Tagliaro: Direct screening of herbal blends for new synthetic cannabinoids by MALDI-TOF MS. In: Journal of Mass Spectrometry Band 47, Nummer 1, Januar 2012, S. 141–146. doi:10.1002/jms.2036. PMID 22282100.
- L. Signor, E. Boeri Erba: Matrix-assisted laser desorption/ionization time of flight (MALDI-TOF) mass spectrometric analysis of intact proteins larger than 100 kDa. In: Journal of visualized experiments : JoVE. Nummer 79, 2013. doi:10.3791/50635. PMID 24056304. PMC 3857990 (freier Volltext).
- Fahad J Alharbi, Tarekegn Geberhiwot, Derralynn A Hughes, Douglas G Ward. A novel rapid MALDI-TOF-MS-based method for measuring urinary globotriaosylceramide in Fabry patients. In: J Am Soc Mass Spectrom 27/2016:719–725. doi:10.1007/s13361-015-1318-4.
- M. Pavlovic, K. Grünwald, V. Zeller-Péronnet, M. Maggipinto, I. Huber, R. Konrad, A. Berger, U. Messelhäußer, P. Zimmermann, S. Hörmansdorfer, A. Sing, U. Busch: Identifizierung von Bakterien mit dem MALDI Biotyper am Bayerischen Landesamt für Gesundheit und Lebensmittelsicherheit. In: Das Gesundheitswesen. 73/2011, doi:10.1055/s-0031-1274482
- P Nenoff, M Erhard, J C Simon, J Herrmann, G K Muylowa, W Rataj, Yvonne Gräser. MALDI-TOF mass spectrometry: A rapid method for the identification of dermatophyte species. In: Med Mycol 51/2013: 17–24. doi:10.3109/13693786.2012.685186
- LABO online: MALDI-TOF-MS verhindert teure Fruchtsaftbombagen, 1. Mai 2012
- Peter Lasch, Herbert Nattermann, Marcel Erhard, Maren Stämmler, Roland Grunow: MALDI-TOF Mass Spectrometry Compatible Inactivation Method for Highly Pathogenic Microbial Cells and Spores. In: Analytical Chemistry. Band 80, Nr. 6, 15. März 2008, ISSN 0003-2700, S. 2026–2034, doi:10.1021/ac701822j.
- J. Rau, R. Sting (CVUA Stuttgart); T. Eisenberg (Landesbetrieb Hessisches Landeslabor Gießen): MALDI-UP – Ein offener Katalog für Datenbank-Einträge von Anwendern für Anwender auf MALDI-TOF-MS-user-platform.ua-bw.de
- Jörg Rau: Tierartendifferenzierung von Fleisch mittels MALDI-TOF-MS, CVUA Stuttgart
- Jörg Rau, Nadine Korte, Martin Dyk, Olivera Wenninger, Pat Schreiter, Ekkehard Hiller: Rapid animal species identification of feta and mozzarella cheese using MALDI-TOF mass-spectrometry. In: Food Control. Band 117, 2020, ISSN 0956-7135, S. 107349, doi:10.1016/j.foodcont.2020.107349 (sciencedirect.com).
- Antje Stahl, Uwe Schröder: Development of a MALDI–TOF MS-Based Protein Fingerprint Database of Common Food Fish Allowing Fast and Reliable Identification of Fraud and Substitution. In: Journal of Agricultural and Food Chemistry. Band 65, Nr. 34, 2017, ISSN 0021-8561, S. 7519–7527, doi:10.1021/acs.jafc.7b02826.
- Jörg Kriegsmann, Mark Kriegsmann, Rita Casadonte: MALDI TOF imaging mass spectrometry in clinical pathology: A valuable tool for cancer diagnostics (Review). In: Int J Oncol 46/2015: 893–906. doi:10.3892/ijo.2014.2788.
- H. Li, Z. Tang, H. Zhu, H. Ge, S. Cui, W. Jiang: Proteomic study of benign and malignant pleural effusion. In: Journal of cancer research and clinical oncology. Band 142, Nummer 6, Juni 2016, S. 1191–1200, doi:10.1007/s00432-016-2130-7, PMID 26945985.
- K. Jia, W. Li, F. Wang, H. Qu, Y. Qiao, L. Zhou, Y. Sun, Q. Ma, X. Zhao: Novel circulating peptide biomarkers for esophageal squamous cell carcinoma revealed by a magnetic bead-based MALDI-TOFMS assay. In: Oncotarget. Band 7, Nummer 17, April 2016, S. 23569–23580, doi:10.18632/oncotarget.8123, PMID 26993605, PMC 5029648 (freier Volltext).
- DIN EN ISO 10927:2018-10, Kunststoffe - Bestimmung der Molmasse und Molmassenverteilung von polymeren Spezies durch matrixunterstützte Laser-Desorptions/Ionisations-Flugzeit-Massenspektrometrie (MALDI-TOF-MS) Deutsche Fassung. Beuth Verlag, doi:10.31030/2843045.