Gen
Als Gen wird meist ein Abschnitt auf der DNA bezeichnet, der Grundinformationen für die Entwicklung von Eigenschaften eines Individuums und zur Herstellung einer biologisch aktiven RNA enthält. Bei diesem Prozess der Transkription wird vom codogenen DNA-Strangabschnitt eine komplementäre Kopie in Form einer RNA hergestellt.
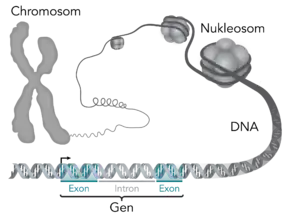
Gezeigt ist ein eukaryotisches Gen, das (hier verkürzt) Introns und Exons enthält, und im Hintergrund der zum Chromosom kondensierte DNA-Doppelstrang.
Es gibt verschiedene Arten der RNA. Bei der Translation, einem Teilvorgang der Proteinbiosynthese, wird die Aminosäuresequenz des betreffenden Proteins von der mRNA abgelesen. Die Proteine übernehmen im Körper jeweils spezifische Funktionen, mit denen sich die Merkmale ausprägen können. Der Aktivitätszustand eines Gens bzw. dessen Ausprägung, seine Expression, kann in einzelnen Zellen verschieden reguliert werden.
Als Erbanlage oder Erbfaktor werden allgemein die nur elektronenmikroskopisch sichtbaren Gene auf spezifischen Plätzen in den Chromosomen bezeichnet, da sie die Träger von Erbinformation sind, die durch Reproduktion an Nachkommen weitergegeben wird. Die Erforschung des Aufbaus, der Funktion und Vererbung von Genen ist Gegenstand der Genetik. Die gesamte Erbinformation einer Zelle wird Genom genannt.
Forschungsgeschichte
1856 begann Johann Gregor Mendel, in Kreuzungsversuchen mit Erbsen die Vererbung von sichtbaren Merkmalen zu untersuchen. Er schlug als erster die Existenz von bestimmten „materiellen Elementen“ vor, die als Erbfaktoren von Eltern auf die Nachkommen übertragen werden. Er fand, dass Merkmale voneinander unabhängig vererbt werden können, und dass es dominante und rezessive Faktoren gibt. Er entwickelte die Hypothese, dass es homo- und heterozygote Individuen gibt und legte damit die Grundlage für die Unterscheidung zwischen Genotyp und Phänotyp.[1]
1900 gilt als das Jahr der „Wiederentdeckung“ der mendelschen Regeln, als die Botaniker Hugo de Vries, Erich Tschermak und Carl Correns Mendels Entdeckung aufgriffen, dass es quantifizierbare Regeln gibt, nach denen die Faktoren, die für die Ausprägung von Merkmalen verantwortlich sind, an die Nachkommen weitergegeben werden. Correns prägte dabei den Begriff der Anlage bzw. Erbanlage. William Bateson erinnerte 1902 in Mendel’s Principles of Heredity daran, dass es zwei Varianten der Erbfaktoren in jeder Zelle gibt. Er nannte das zweite Element Allelomorph nach dem griechischen Wort für „Andere“ und prägte damit den Begriff des Allels. Archibald Garrod, ein britischer Arzt, hatte sich mit Stoffwechselerkrankungen beschäftigt und festgestellt, dass diese in Familien vererbt wurden. Garrod erkannte, dass die Gesetze also auch bei Menschen gültig waren, und vermutete, die Erbanlagen seien die Basis für die Chemische Individualität von Menschen. Dass die von Mendel „Elemente“ genannten Faktoren auf den Chromosomen zu finden sind, wurde 1902 von Walter Sutton vermutet.
August Weismann stellte in seinen Vorträgen zur Deszendenztheorie 1904 die Entdeckung vor, dass es einen Unterschied zwischen Körperzellen und Keimzellen gibt, und dass nur Letztere in der Lage sind, neue Organismen hervorzubringen. Keimzellen sollten eine „Vererbungssubstanz“ enthalten, die sich aus einzelnen Elementen zusammensetze, die er Determinanten nannte. Diese Determinanten sollten für die sichtbare Ausprägung beispielsweise der Gliedmaßen verantwortlich sein.
Die Bezeichnung „Gen“ wurde erstmals 1909 von dem Dänen Wilhelm Johannsen gebraucht.[2] Er benannte die Objekte, mit denen sich die Vererbungslehre beschäftigt, nach dem griechischen Substantiv γένος genos für „Nachkommenschaft“. Für ihn waren sie jedoch nur eine Rechnungseinheit. Bereits drei Jahre zuvor hatte William Bateson die Wissenschaft von der Vererbung als Genetik bezeichnet, nach dem griechischen Adjektiv γεννητικός gennetikos für „hervorbringend“. Zu diesem Zeitpunkt war die materielle Natur der Gene immer noch vollkommen unklar.
In den ersten Jahren des 20. Jahrhunderts nahmen sich die Genetiker nach verschiedenen Pflanzen auch Insekten und später Vögel vor, um die Vererbungsgesetze zu testen. In Kombination mit den 1842 entdeckten und 1888 benannten Chromosomen entstand so die Chromosomentheorie der Vererbung. Es war durch verbesserte Färbetechniken beobachtet worden, dass sich vor der Zellteilung die Chromosomen verdoppeln und sich dann auf die Tochterzellen verteilen. Daher waren sie als Träger der Erbanlagen in Frage gekommen. Während dieser Zeit herrschte eine Kontroverse zwischen den Vertretern der Hypothese von Johannsen und Mendel, dass Gene etwas Materielles sind, und deren Kritikern, die eine Verbindung von Genen und Chromosomen als „Physikalismus“ und „Mendelismus“ abtaten und Gene weiterhin als abstrakte Einheiten betrachteten.
Thomas Hunt Morgan war ebenfalls überzeugt, dass die Einheiten, die für die verschiedenen Merkmale verantwortlich waren, nicht physikalischer Natur sein konnten. Er versuchte, den Mendelismus zu widerlegen und begann 1910 mit Kreuzungsversuchen an Schwarzbäuchigen Taufliegen. Seine Arbeiten erbrachten jedoch das Gegenteil: Den endgültigen Beweis, dass Gene auf Chromosomen liegen und damit materieller Natur sind. Zusammen mit seinen Mitarbeitern, darunter Calvin Bridges, Alfred Sturtevant und Hermann Muller, fand er viele natürliche Mutationen und untersuchte in unzähligen Kreuzungen die Wahrscheinlichkeit, dass zwei Merkmale gemeinsam vererbt werden. Sie konnten so zeigen, dass Gene an bestimmten Stellen auf den Chromosomen liegen und hintereinander aufgereiht sind. Da unter dem Mikroskop auch das Crossing over beobachtet werden konnte, war bekannt, dass Chromosomen Abschnitte austauschen können. Je näher zwei Gene auf dem Chromosom beieinander liegen, desto größer die Wahrscheinlichkeit, dass sie gemeinsam vererbt und nicht durch ein Crossing-over-Ereignis getrennt werden. Dadurch konnten Angaben über die Entfernung zweier Gene gemacht werden, die nach Morgan in centiMorgan angegeben werden. Gemeinsam erstellte die Forschergruppe in jahrelanger Arbeit die erste Genkarte.
Hermann Muller begann einige Zeit später, mit Röntgenstrahlen zu experimentieren, und konnte zeigen, dass die Bestrahlung von Fliegen deren Mutationsrate stark erhöht. Diese Erkenntnis aus dem Jahr 1927 war eine Sensation, da dadurch zum ersten Mal tatsächlich gezeigt wurde, dass Gene physikalische Objekte sind, die sich von außerhalb beeinflussen lassen.
1928 wies Frederick Griffith in dem als „Griffiths Experiment“ bekannt gewordenen Versuch zum ersten Mal nach, dass Gene von Organismen auf andere übertragen werden können. Der von ihm nachgewiesene Vorgang war die genetische Transformation. 1941 zeigten George Wells Beadle und Edward Lawrie Tatum, dass Mutationen in Genen für Defekte in Stoffwechselwegen verantwortlich sind, was zeigte, dass spezifische Gene spezifische Proteine codieren. Diese Erkenntnisse führten zur „Ein-Gen-ein-Enzym-Hypothese“, die später zur „Ein-Gen-ein-Polypeptid-Hypothese“ präzisiert wurde. Oswald Avery, Colin MacLeod und Maclyn McCarty zeigten 1944, dass die DNA die genetische Information enthält. 1953 wurde die Struktur der DNA von James D. Watson und Francis Crick, basierend auf den Arbeiten von Rosalind Franklin und Erwin Chargaff, entschlüsselt und das Modell der DNA-Doppelhelix entworfen. 1969 gelang Jonathan Beckwith als erstem die chemische Isolierung eines einzelnen Gens.
Definition des Begriffs
Die Definition, was genau ein Gen ist, hat sich verändert und wurde neuen Erkenntnissen angepasst. 2006 suchten 25 Wissenschaftler des Sequence Ontologie Consortiums der Universität Berkeley nach einer treffend formulierten aktuellen Definition. Sie einigten sich nach zwei Tagen auf eine gemeinsame Version. Ein Gen ist demnach “a locatable region of genomic sequence, corresponding to a unit of inheritance, which is associated with regulatory regions, transcribed regions and/or other functional sequence regions” (deutsch: „eine lokalisierbare Region genomischer DNA-Sequenz, die einer Erbeinheit entspricht und mit regulatorischen, transkribierten und/oder funktionellen Sequenzregionen assoziiert ist“).[3]
Durch das Projekt ENCODE (ENCyclopedia Of DNA Elements), bei dem die Transkriptionsaktivität des Genoms gemappt wurde, wurden neue komplexe Regulationsmuster entdeckt. Dabei wurde auch festgestellt, dass die Transkription nichtcodierender RNA in weit größerem Umfang als bislang angenommen stattfindet. Dieser Befund wird im folgenden Definitionsvorschlag berücksichtigt: “A gene is a union of genomic sequences encoding a coherent set of potentially overlapping functional products” (deutsch: „Ein Gen ist eine Vereinigung genomischer Sequenzen, die einen zusammenhängenden Satz von eventuell überlappenden funktionellen Produkten codieren“).[4]
Aufbau
Auf molekularer Ebene besteht ein Gen aus zwei unterschiedlichen Bereichen:
- Einem DNA-Abschnitt, von dem durch Transkription eine einzelsträngige RNA-Kopie hergestellt wird.
- Allen zusätzlichen DNA-Abschnitten, die an der Regulation dieses Kopiervorgangs beteiligt sind.
Es gibt verschiedene Besonderheiten im Aufbau von Genen verschiedener Lebewesen. In der Zeichnung wird der Aufbau eines typischen eukaryotischen Gens dargestellt, das ein Protein codiert.

Schematischer Aufbau eines eukaryotischen Gens
Vor der Transkriptionseinheit oder auch innerhalb der Exons (hellblau und dunkelblau) und Introns(rosa und rot) liegen regulatorische Elemente, wie zum Beispiel Enhancer oder Promotor. An diese binden, abhängig von der Sequenz, verschiedene Proteine, wie beispielsweise die Transkriptionsfaktoren und die RNA-Polymerase. Die prä-mRNA (unreife mRNA), die im Zellkern bei der Transkription zunächst entsteht, wird in dem Reifungsprozess zur reifen mRNA modifiziert. Die mRNA enthält neben dem direkt proteincodierenden Offenen Leserahmen noch untranslatierte, also nichtcodierende Bereiche, den 5' untranslatierten Bereich (5' UTR) und den 3' untranslatierten Bereich (3' UTR). Diese Bereiche dienen zur Regulation der Translationsinitiation und zur Regulation der Aktivität der Ribonukleasen, die die RNA wieder abbauen.
Die Gene der Prokaryoten unterscheiden sich im Aufbau von eukaryotischen Genen dadurch, dass sie keine Introns besitzen. Zudem können mehrere unterschiedliche RNA-bildende Genabschnitte sehr nah hintereinander geschaltet sein (man spricht dann von polycistronischen Genen) und in ihrer Aktivität von einem gemeinsamen regulatorischen Element geregelt werden. Diese Gencluster werden gemeinsam transkribiert, aber in verschiedene Proteine translatiert. Diese Einheit aus Regulationselement und polycistronischen Genen nennt man Operon. Operons sind typisch für Prokaryoten.
Gene codieren nicht nur die mRNA, aus der dann die Proteine translatiert werden, sondern auch die rRNA und die tRNA sowie weitere Ribonukleinsäuren, die andere Aufgaben in der Zelle haben, beispielsweise bei der Proteinbiosynthese oder der Genregulation. Ein Gen, das ein Protein codiert, enthält eine Beschreibung der Aminosäure-Sequenz dieses Proteins. Diese Beschreibung liegt in einer chemischen Sprache vor, nämlich im genetischen Code in Form der Nukleotid-Sequenz der DNA. Die einzelnen „Kettenglieder“ (Nukleotide) der DNA stellen – in Dreiergruppen (Tripletts, Codon) zusammengefasst – die „Buchstaben“ des genetischen Codes dar. Der codierende Bereich, also alle Nukleotide, die direkt an der Beschreibung der Aminosäuresequenz beteiligt sind, wird als offener Leserahmen bezeichnet. Ein Nukleotid besteht aus einem Teil Phosphat, einem Teil Desoxyribose (Zucker) und einer Base. Eine Base ist entweder Adenin, Thymin, Guanin oder Cytosin.
Gene können mutieren, sich also spontan oder durch Einwirkung von außen (beispielsweise durch Radioaktivität) verändern. Diese Veränderungen können an verschiedenen Stellen im Gen erfolgen. Demzufolge kann ein Gen nach einer Reihe von Mutationen in verschiedenen Zustandsformen vorliegen, die man Allele nennt. Eine DNA-Sequenz kann auch mehrere überlappende Gene enthalten. Durch Genduplikation verdoppelte Gene können sequenzidentisch sein, dennoch aber unterschiedlich reguliert werden und damit zu unterschiedlichen Aminosäuresequenzen führen, ohne dass sie Allele sind.
Verhältnis Introns zu Exons
Generell schwankt das Verhältnis zwischen Introns und Exons von Gen zu Gen sehr stark. So gibt es einige Gene ohne Introns, während andere zu über 95 % aus Introns bestehen.[5] Beim Dystrophin-Gen – mit 2,5 Millionen Basenpaaren das größte menschliche Gen[6] – besteht das daraus codierte Protein aus 3685 Aminosäuren.[7] Der Anteil der codierenden Basenpaare beträgt somit 0,44 %.
In der nachfolgenden Tabelle sind einige Proteine und das jeweils codierende Gen aufgeführt.
| Protein | Anzahl der Aminosäuren |
Gen | Anzahl der Basenpaare |
Anzahl codierender Basenpaare |
Anteil codierender Sequenz |
Referenz |
|---|---|---|---|---|---|---|
| Dystrophin | 3685 | DMD | 2.500.000 | 11.055 | 0,44 % | [6][7] |
| FOXP2 | 715 | FOXP2 | 603.000 | 2145 | 0,36 % | [8] |
| Neurofibromin | 2838 | NF1 | 280.000 | 8514 | 3,0 % | [9] |
| BRCA2 | 3418 | BRCA2 | 84.000 | 10.254 | 12,2 % | [10] |
| BRCA1 | 1863 | BRCA1 | 81.000 | 5589 | 6,9 % | [10] |
| Survivin | 142 | BIRC5 | 15.000 | 426 | 2,9 % | [11][12] |
Genaktivität und Regulation
Gene sind dann „aktiv“, wenn ihre Information in RNA umgeschrieben wird, das heißt, die Transkription stattfindet. Je nach Funktion des Gens entsteht also mRNA, tRNA oder rRNA. In der Folge kann also, muss aber nicht zwingend, bei mRNA aus dieser Aktivität auch ein Protein translatiert werden. Eine Übersicht über die Vorgänge bieten die Artikel Genexpression und Proteinbiosynthese.
Die Aktivität einzelner Gene wird über eine Vielzahl von Mechanismen gesteuert und kontrolliert. Ein Weg ist die Steuerung über die Rate ihrer Transkription in hnRNA. Ein anderer Weg ist der Abbau der mRNA, bevor sie beispielsweise über siRNA translatiert wird. Kurzfristig erfolgt die Genregulation durch Bindung und Ablösung von Proteinen, sogenannten Transkriptionsfaktoren, an spezifische Bereiche der DNA, die sogenannten „regulatorischen Elemente“. Langfristig wird dies über Methylierung oder das „Verpacken“ von DNA-Abschnitten in Histonkomplexe erreicht. Auch die regulatorischen Elemente der DNA unterliegen der Variation. Der Einfluss von Änderungen in der Genregulation einschließlich der Steuerung des alternativen Splicings dürfte vergleichbar mit dem Einfluss von Mutationen proteincodierender Sequenzen sein. Mit klassischen genetischen Methoden – durch Analyse von Erbgängen und Phänotypen – sind diese Effekte in der Vererbung normalerweise nicht voneinander zu trennen. Lediglich die Molekularbiologie kann hier Hinweise geben. Eine Übersicht über die Regulationsvorgänge von Genen wird im Artikel Genregulation dargestellt.
Organisation von Genen
Bei allen Lebewesen codiert nur ein Teil der DNA für definierte RNAs. Die übrigen Teile der DNA werden als nichtcodierende DNA bezeichnet. Sie hat Funktionen in der Genregulation, beispielsweise für die Regulation des alternativen Splicings, und hat Einfluss auf die Architektur der Chromosomen.
Der Ort auf einem Chromosom, an dem sich das Gen befindet, wird als Genort bezeichnet. Gene sind darüber hinaus nicht gleichmäßig auf den Chromosomen verteilt, sondern kommen zum Teil in sogenannten Clustern vor. Gencluster können dabei aus zufällig in räumlicher Nähe zueinander liegenden Genen bestehen, oder es handelt sich um Gruppen von Genen, die für Proteine codieren, die in einem funktionellen Zusammenhang stehen. Gene, deren Proteine ähnliche Funktion haben, können aber auch auf verschiedenen Chromosomen liegen.
Es gibt Abschnitte auf der DNA, die für mehrere verschiedene Proteine codieren. Der Grund dafür sind überlappende offene Leserahmen.
Genetische Variation und genetische Variabilität
Als genetische Variation wird das Auftreten von genetischen Varianten (Allele, Gene oder Genotypen) bei individuellen Lebewesen bezeichnet. Sie entsteht durch Mutationen, aber auch durch Vorgänge bei der Meiose („Crossing over“), durch die Erbanlagen der Großeltern unterschiedlich auf die Geschlechtszellen verteilt werden. Für die Entstehung neuer Gene können ebenfalls Mutationen oder De-novo-Entstehung ursächlich sein.[13]
Genetische Variabilität ist dagegen die Fähigkeit einer gesamten Population, Individuen mit unterschiedlichem Erbgut hervorzubringen. Hierbei spielen nicht nur genetische Vorgänge, sondern auch Mechanismen der Partnerwahl eine Rolle. Die genetische Variabilität spielt eine entscheidende Rolle für die Fähigkeit einer Population, unter veränderten Umweltbedingungen zu überleben, und stellt einen wichtigen Faktor der Evolution dar.
Besondere Gene
RNA-Gene in Viren
Obwohl bei allen zellbasierten Lebensformen Gene als DNA-Abschnitte vorliegen, gibt es einige Viren, deren genetische Information in Form von RNA vorliegt. RNA-Viren befallen eine Zelle, die dann sofort mit der Produktion von Proteinen direkt nach Anleitung der RNA beginnt; eine Transkription von DNA nach RNA entfällt. Retroviren hingegen übersetzen ihre RNA bei der Infektion in DNA, und zwar unter Mitwirkung des Enzyms Reverse Transkriptase.
Pseudogene
Als Gen im engeren Sinne bezeichnet man in der Regel eine Nukleotidsequenz, die die Information für ein Protein enthält, das unmittelbar funktionsfähig ist. Pseudogene stellen dagegen Genkopien dar, die kein funktionelles Protein in voller Länge codieren. Oftmals sind diese durch Genduplikationen entstanden und/oder durch Mutationen, die sich in der Folge ohne Selektion auch im Pseudogen akkumulieren (anhäufen), und ihre ursprüngliche Funktion verloren haben. Einige scheinen dennoch eine Rolle bei der Regulierung der Genaktivität zu spielen. Das menschliche Genom enthält etwa 20.000 Pseudogene. Das Humangenomprojekt wurde mit dem Ziel gegründet, das Genom des Menschen vollständig zu entschlüsseln.
Springende Gene
Sie werden auch als Transposons bezeichnet und sind mobile Erbgutabschnitte, die sich innerhalb der DNA einer Zelle frei bewegen können. Aus ihrem angestammten Ort im Erbgut schneiden sie sich selbst aus und fügen sich an einer beliebig anderen Stelle wieder ein. Biologen um Fred Gage vom Salk Institute for Biological Studies in La Jolla (USA) haben nachgewiesen, dass diese springenden Gene nicht nur wie bislang angenommen in den Zellen der Keimbahn vorkommen, sondern auch in Nerven-Vorläuferzellen aktiv sind. Forschungsergebnisse von Eric Lander et al. (2007) zeigen, dass Transposons eine wichtige Funktion haben, indem sie als kreativer Faktor im Genom wichtige genetische Innovationen rasch im Erbgut verbreiten können.[14]
Orphangene
Orphan-Gene sind Gene ohne nachweisbare Homologie in anderen Linien. Sie werden auch ORFans genannt, insbesondere in der mikrobiellen Literatur (mit ORF als Akronym für englisch open reading frame ‚offener Leserahmen‘). Orphan-Gene sind eine Teilmenge von taxonomisch eingeschränkten Genen, die auf einer bestimmten taxonomischen Ebene (z. B. pflanzenspezifisch) einzigartig sind. Sie gelten in der Regel als einzigartig für ein sehr schmales Taxon, sogar für eine Art (Spezies). Orphan-Gene unterscheiden sich dadurch, dass sie linienspezifisch sind und keine bekannte Geschichte der gemeinsamen Verdoppelung und Neuordnung außerhalb ihrer spezifischen Spezies oder Gruppe haben. In Menschen gibt es beispielsweise 634 Gene, die dem Schimpansen fehlen. Umgekehrt fehlen dem Menschen 780 Schimpansen-Gene.[15]
Typische Genomgrößen und Genanzahl
| Organismus / Biologisches System | Anzahl der Gene | Basenpaare insgesamt |
|---|---|---|
| Gemeiner Wasserfloh[16] | 30.907 | 2·108 |
| Acker-Schmalwand (Arabidopsis thaliana, Modellpflanze) | >25.000 | 108–1011 |
| Mensch[17] | ~22.500 | 3·109 |
| Drosophila melanogaster (Fliege) | 12.000 | 1,6·108 |
| Backhefe (Saccharomyces cerevisiae) | 6.000 | 1,3·107 |
| Bakterium | 180–7.000 | 105−107 |
| Escherichia coli | ~5.000 | 4,65·106 |
| Carsonella ruddii | 182 | 160.000 |
| DNA-Virus | 10–300 | 5.000–200.000 |
| RNA-Virus | 1–25 | 1.000–23.000 |
| Viroid | 0 | 246–401 |
Literatur
- Ernst Peter Fischer: Geschichte des Gens. Fischer, Frankfurt 2003, ISBN 3-596-15363-8
- Benjamin Lewin: Molekularbiologie der Gene. Spektrum Akademischer Verlag, Heidelberg 2002, ISBN 3-8274-1349-4 (deutsch)
- Benjamin Lewin: Genes 8. Pearson Prentice Hall, London 2004, ISBN 0-13-143981-2 (englisch)
- Inge Kronberg: Welche Gene machen den Menschen zum Menschen? In: Biologie in unserer Zeit. 34.2004,4, S. 206–207, ISSN 0045-205X
- Siddhartha Mukherjee: Das Gen. Fischer, Frankfurt 2017, ISBN 978-3-10-002271-4
Weblinks
- vcell.de: Der lange Weg zum Gen: Meilensteine
- plato.stanford.edu: Gene Eintrag in Edward N. Zalta (Hrsg.): Stanford Encyclopedia of Philosophy.
- Peter Godfrey-Smith, Kim Sterelny: Biological Information. In: Edward N. Zalta (Hrsg.): Stanford Encyclopedia of Philosophy.. Pressemitteilung Universität Freiburg, 3. September 2008
- Joachim Bauer: swr.de: Aus der Werkstatt der Evolution – Neue Erkenntnisse über die Gene. Vortrag in der SWR 2-Sendereihe Aula, 23. November 2008 (Real Audio ca. 30 Min. – Manuskript ca. sechs Seiten)
- genecards.org: Human Gene Database (englisch)
- Michael Stang: deutschlandfunk.de: Gehackte Gene. Deutschlandfunk, Wissenschaft im Brennpunkt, 3. Oktober 2014. Zum Datenschutz der „genetischen Privatsphäre“
Einzelnachweise
- Gregor Mendel: Versuche über Pflanzen-Hybriden. In: Verhandlungen des naturforschenden Vereines in Brünn. Band IV, 1865, S. 3–47 (mpg.de [abgerufen am 20. Dezember 2021]).
- Wilhelm Ludwig Johannsen: Elemente der exakten Erblichkeitslehre mit Grundzügen der biologischen Variationsstatistik. 1913 (Onlinefassung).
- Helen Pearson: What is a gene? In: Nature. Band 441, Mai 2006, S. 398–401, PMID 16724031.
- M. Gerstein, C. Bruce, J. Rozowsky, D. Zheng, J. Du, J. Korbel, O. Emanuelsson, Z. Zhang, S. Weissman, M. Snyder: What is a gene, post-ENCODE? History and updated definition. In: Genome Research. Band 17, Nr. 6, Juni 2007, S. 669–681, PMID 17567988.
- E. H. McConekey: How the Human Genome works. Jones & Bartlett, 2004, ISBN 0-7637-2384-3, S. 5 (englisch).
- N. Shiga u. a.: Disruption of the Splicing Enhancer Sequence within Exon 27 of the Dystrophin Gene by a Nonsense Mutation Induces Partial Skipping of the Exon and Is Responsible for Becker Muscular Dystrophy. In: J. Clin. Invest. Band 100, 1997, S. 2204–2210, PMID 9410897 (englisch).
- M. Matsuo: Duchenne muscular dystrophy. In: Southeast Asian J Trop Med Public Health. Band 26, 1995, S. 166–171, PMID 8629099 (englisch).
- A. F. Wright, N. Hastie: Genes and Common Diseases. Genetics in Modern Medicine. Cambridge University Press, 2007, ISBN 0-521-83339-6 (englisch).
- I. Bottillo u. a.: Functional analysis of splicing mutations in exon 7 of NF1 gene. In: BMC Medical Genetics. Band 8, 2007, PMID 17295913 (englisch, biomedcentral.com).
- B. Górski u. a.: Usefulness of polymorphic markers in exclusion of BRCA1/BRCA2 mutations in families with aggregation of breast/ovarian cancers. In: J. Appl. Genet. Band 44, 2003, S. 419–423, PMID 12923317 (englisch, poznan.pl [PDF; 43 kB]).
- UniProt O15392
- M. Kappler: Molekulare Charakterisierung des IAP Survivin in Weichteilsarkomen. Bedeutung für Prognose und Etablierung neuer Therapiestrategien. Universität Halle-Wittenberg, 2005 (uni-halle.de [PDF; 1,4 MB] Dissertation).
- A. R. Carvunis, T. Rolland u. a.: Proto-genes and de novo gene birth. In: Nature. Band 487, Nummer 7407, Juli 2012, S. 370–374. doi:10.1038/nature11184, PMID 22722833, PMC 3401362 (freier Volltext).
- Eric Lander et al: Genome of the marsupial Monodelphis domestica reveals innovation in non-coding sequences, Nature 447, 167-177 (10. Mai 2007).
- Jorge Ruiz-Orera, Jessica Hernandez-Rodriguez, Cristina Chiva, Eduard Sabidó, Ivanela Kondova: Origins of De Novo Genes in Human and Chimpanzee. In: PLOS Genetics. Band 11, Nr. 12, 31. Dezember 2015, ISSN 1553-7404, S. e1005721, doi:10.1371/journal.pgen.1005721, PMID 26720152, PMC 4697840 (freier Volltext) – (plos.org [abgerufen am 23. September 2019]).
- John K. Colbourne et al.: The Ecoresponsive Genome of Daphnia pulex. In: Science. Vol. 331, Nr. 6017, 4. Februar 2011, S. 555–561, doi:10.1126/science.1197761.
- Mihaela Pertea and Steven L Salzberg (2010): Between a chicken and a grape: estimating the number of human genes. Genome Biology 11:206