Plasmid
Plasmide sind kleine, in der Regel ringförmige, autonom replizierende, doppelsträngige DNA-Moleküle, die in Bakterien und in Archaeen vorkommen können, aber nicht zum Bakterienchromosom (Kernäquivalent) zählen, also extrachromosomal vorliegen (Abb. 1). Sie werden daher auch als extrachromosomale Elemente (ECEs) bezeichnet. Nur selten treten Plasmide auch in Eukaryoten auf (z. B. als 2-Mikrometer-Ring in Backhefe). Ihre Größe beträgt meist zwischen 2 kBp und 200 kBp,[1] in Ausnahmefällen zwischen weniger als 1 kBp (Miniplasmid) und über 1000 kBp (Megaplasmid),[2] wobei der Übergang zwischen Megaplasmid und Minichromosom fließend ist.[3] Analoge extrachromosomale DNA in Archaeen sind Borgs.
.svg.png.webp)

Aufbau
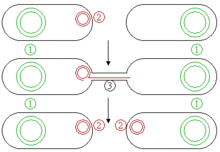
Plasmide können viele verschiedene Gene enthalten. Vermitteln diese Gene z. B. eine Antibiotika-Resistenz, kann daraus für das Wirtsbakterium ein Selektionsvorteil resultieren. Jedes Plasmid enthält mindestens eine Sequenz, die als Replikationsursprung (engl. Origin of Replication, kurz ORI) dient. Ist der Replikationsursprung kompatibel zu dem Bakterienstamm, so kann das Plasmid unabhängig von der chromosomalen DNA repliziert werden (Abb. 2). Plasmide sind somit autonom replizierend (Replikons) und werden vererbt. Je nach Art des Replikationsursprungs liegen in einer Bakterienzelle wenige (low-copy, aufgrund von Iterons, Antisense-RNA oder ctRNA) oder sehr viele Kopien (high-copy) vor.
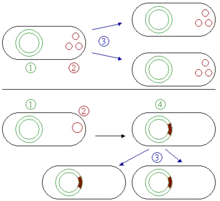
Episome sind bei Bakterien Plasmide, die sich in die chromosomale DNA des Wirtsorganismus integrieren können, wie das F-Plasmid (Abb. 3). Dort können sie für lange Zeit verbleiben, werden dabei mit jeder Zellteilung des Wirts mit repliziert und können sogar zu einem integralen Bestandteil seiner DNA werden. Sie können jedoch auch wie andere Plasmide eigenständig existieren.
Bei den Eukaryoten gelegentlich vorkommende virale Episome wie die cccDNA des Hepatitis-B-Virus oder die Episome der Herpesviren und Adenoviren integrieren nicht in das Genom des Wirtes, sondern liegen daneben im Zellkern vor. Eine Ausnahme bilden die Episome von Pockenviren, welche im Zytosol auftreten.[4] Virale Episome sind Mechanismen zur Immunevasion bei Infektionen mit persistenten Pathogenen.
Klassifikation
Es gibt zwei grundlegende Gruppen von Plasmiden, konjugierende und nicht-konjugierende. Konjugierende Plasmide enthalten ein so genanntes tra-Gen, das die Konjugation, den parasexuellen Austausch von Plasmiden zwischen zwei Bakterien, auslösen kann (Abb. 4). Nicht-konjugierende Plasmide haben diese Fähigkeit nicht, sie können aber zusammen mit konjugierenden Plasmiden während der Konjugation übertragen werden.
Verschiedene Plasmidtypen können nebeneinander in ein und derselben Zelle existieren, in Escherichia coli z. B. bis zu sieben. Sind zwei Plasmide zueinander inkompatibel, wird eines von ihnen zerstört. Abhängig von der Fähigkeit, nebeneinander in derselben Zelle zu existieren, können sie also in Inkompatiblitätsgruppen eingeteilt werden.
Die Klassifikation von Plasmiden kann durch ihre Funktion erfolgen. Es werden fünf Hauptklassen unterschieden:
- Fruchtbarkeits-(F-)Plasmide (siehe F-Plasmid), die nur tra-Gene enthalten. Ihre einzige Funktion ist die Einleitung der Konjugation.
- Resistenz-(R-)Plasmide, die Resistenzgene gegen Antibiotika oder Gifte enthalten.
- Col-Plasmide, die Gene enthalten, die für Colicine (Proteine, die für andere Bakterien toxisch sind) kodieren.
- Degradations-Plasmide, die den Abbau von ungewöhnlichen Substanzen, wie z. B. von Toluol oder Salicylsäure ermöglichen.
- Virulenz-Plasmide, die ein Bakterium zu einem Krankheitserreger machen.
Eine besondere Art von Plasmiden stellen die sog. Ti-Plasmide (Tumor inducing) dar, die eine Transfer-DNA übertragen. Sie sind oft ein Bestandteil von bestimmten Bakterien (Agrobacterium tumefaciens oder Agrobacterium rhizogenes) und werden von diesen in Pflanzen übertragen. Dort verursachen sie die einzige bekannte Krebserkrankung in Pflanzen.
Ein weiteres besonderes Plasmid ist pR1SE von Halobacterium lacusprofundi R1S1, gefunden 2017 in der Antarktis. Dieses ist in der Lage, Vesikel (Schutzblasen) auszubilden. Es wird diskutiert, ob pR1SE eine Übergangs- oder Zwischenform zu Viren darstellen könnte.[5][6]
Plasmide vom Typ IncP1 können von Bakterien auf Organismen aller drei Domänen übertragen werden (englisch trans-kingdom conjugation, TKC – sic! – zwischen Organismen verschiedener Domänen).[7]
Verwendung
Plasmide sind wichtige Werkzeuge der Molekularbiologie, Genetik, Biochemie und anderer biologischer und medizinischer Bereiche. Sie werden in der Gentechnik als Vektoren bezeichnet und dazu benutzt, um Gene zu vervielfältigen oder zu exprimieren. Die gezielte Anpassung eines Vektors wird als Vektordesign bezeichnet.
Viele der für diese Zwecke eingesetzten Plasmide sind kommerziell erhältlich, sie werden aber auch zwischen Laboren geteilt und sind häufig bei nicht-kommerziellen Anbietern, wie Addgene, erhältlich. Sie leiten sich von den natürlich vorkommenden Plasmiden ab und besitzen noch deren strukturelle Bestandteile wie den Replikationsursprung (Origin of Replication, ORI). Außerdem wurden sie so verändert, dass sie leicht für Klonierungen verwendet werden können (sie enthalten dafür eine so genannte MCS (Multiple Cloning Site), welche Erkennungssequenzen für eine Vielzahl von Restriktionsenzymen enthält): Das zu vervielfältigende Gen wird in Plasmide eingefügt, die über ein Gen mit einer Antibiotika-Resistenz verfügen. Dann werden diese Plasmide in Bakterien eingebracht, die auf einem mit dem entsprechenden Antibiotikum behandelten Nährmedium wachsen. Es werden also nur die Bakterien überleben, die das Plasmid mit der Information für die Antibiotika-Resistenz aufgenommen haben. Bakterien, die das Plasmid nicht aufgenommen haben, sterben durch das Antibiotikum ab. So wirkt das Antibiotikum als Selektionsmarker, der nur die Bakterien mit dem Resistenzplasmid überleben lässt. Meist wird durch das Einfügen des Gens in das Plasmid ein anderes Gen (Reportergen) unterbrochen, welches dann nicht mehr exprimiert werden kann. Diese fehlende Eigenschaft kann zum Screening genutzt werden, weil nur solche Bakterien, welche ein Plasmid mit dem gewünschten Gen aufgenommen haben, diese Eigenschaft nicht mehr besitzen. Bringt man Gene in Plasmide ein, welche dafür sorgen, dass die eingebrachten Gene in großer Menge exprimiert werden (Expressionsplasmide), kann man die entsprechenden Genprodukte in großen Mengen gewinnen. Diese Vorgehensweise erlaubt heute die Herstellung von rekombinantem Insulin (also dem Humaninsulin identischen Insulin), welches früher aufwendig aus Bauchspeicheldrüsen von Schweinen isoliert werden musste.
Soll ein DNA-Abschnitt in einen Organismus (Bakterium, Pflanze, Tier, Mensch etc.) eingebracht werden, können ebenfalls Plasmide als Überträger (Vektoren) verwendet werden. Bei Tier und Mensch ist dieses Verfahren nur bei Zellen in Zellkultur wirklich etabliert, da dort die Zellen als Monolage gut zugänglich sind. Je nach Typ der Zielzelle(n) benötigt man für den Transfer entweder gefällte DNA (z. B. zur Modifikation von Muskelzellen mittels Plasmidaufnahme durch Endocytose) oder in Liposomen verpackte DNA. Es existieren zahlreiche weitere Methoden wie Elektroporation, Genkanone, DNA-Minicircles, Virusvektoren u. v. a., welche die Transfektionseffizienz (Eukaryoten) bzw. Transformationseffizienz (Prokaryoten) für die Zielzellen weiter erhöhen können.
Visualisierung
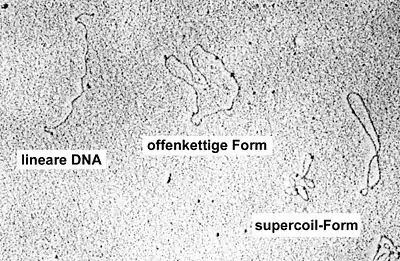
Plasmide kann man durch geeignete Verfahren im Elektronenmikroskop sichtbar machen, etwa durch Färbung mit Uran-Atomen (BAC-Spreitung). Die Ringstruktur des Plasmids in Form einer um sich selbst gewundenen DNA hat dabei topologische Konsequenzen. Die Plasmide können in drei verschiedenen Gestaltformen vorliegen. Die Supercoil-Form ist die natürliche Konformation des Plasmids. Da die DNA-Doppelhelix um sich selbst gewunden ist und sich in einem geschlossenen Plasmid nicht entwinden kann, entsteht eine Torsionsspannung, wodurch sich das Plasmid im Raum um sich selbst krümmt. Der gleiche Effekt ist bei Telefonschnüren zu beobachten, die sich um ihre eigene Achse festwinden. Bei der offenkettigen Form ist einer der beiden DNA-Stränge an einer Stelle gebrochen (einem sogenannten nick), wodurch sich der offene Strang frei um den fixierten drehen kann; dadurch entspannt sich die Torsionsspannung, das Plasmid liegt offen vor. Bei der linearen DNA sind beide Stränge gebrochen, die Kreisstruktur ist aufgehoben.
Unterschied zum Chromosom
Die Unterscheidung von Plasmiden und Chromosomen ist nicht ganz einfach. Eine Definition besagt, dass Plasmide kleiner als Chromosomen sind. Allerdings ist z. B. das Plasmid pSymB von Sinorhizobium meliloti doppelt so groß wie das primäre Genom von Mycoplasma pneumoniae.[8][9] Eine andere Definition ist, dass Plasmide keine essentiellen Gene tragen. Allerdings hängt die Frage, was ein „essentielles Gen“ ist, vom Lebensumfeld ab. Eine weitere Definition besagt, dass Plasmide in der Zelle in mehr Kopien vorliegen als Chromosomen. Allerdings liegen Plasmide wie das F-Plasmid in E. coli und viele andere in gleicher Kopienzahl wie das Chromosom vor. Ein entscheidendes Charakteristikum eines Chromosoms ist, dass seine Replikation mit dem Zellzyklus koordiniert ist. Das heißt, der Start der Replikation erfolgt bei gleichmäßigem Wachstum immer im gleichen Alter der Bakterienzelle. Nach dieser Definition wäre das zweite Chromosom von V. cholerae tatsächlich ein Chromosom.[10] Für Replikons an der Grenze zwischen Chromosomen und Plasmiden wurde der Begriff Chromid vorgeschlagen.[11]
Literatur
- Jeremy M. Berg, John L. Tymoczko, Lubert Stryer: Biochemie. 6. Auflage. Spektrum Akademischer Verlag, Heidelberg 2007, ISBN 978-3-8274-1800-5.
- Donald Voet, Judith G. Voet: Biochemistry. 3. Auflage. John Wiley & Sons, New York 2004, ISBN 0-471-19350-X.
- Bruce Alberts, Alexander Johnson, Peter Walter, Julian Lewis, Martin Raff, Keith Roberts: Molecular Biology of the Cell. 5. Auflage. Taylor & Francis, 2007, ISBN 978-0-8153-4106-2.
Einzelnachweise
- F. Lottspeich, H. Zorbas (Hrsg.): Bioanalytik. Spektrum Akademischer Verlag, Heidelberg/ Berlin 1998, S. 578.
- Brock Mikrobiologie. 11. Auflage. 2009, ISBN 978-3-8273-7358-8, S. 305.
- Finbarr Hayes: Chapter 1 - The Function and Organization of Plasmids. In: Nicola Casali, Andrew Presto. E. Coli Plasmid Vectors: Methods and Applications. (= Methods in Molecular Biology. 235). Humana Press, 2003, ISBN 1-58829-151-0, S. 1–5.
- D. M. Knipe, Peter M. Howley (Hrsg.): Fields Virology. 5. Auflage. Philadelphia 2007, ISBN 978-0-7817-6060-7.
- Susanne Erdmann u. a.: A plasmid from an Antarctic haloarchaeon uses specialized membrane vesicles to disseminate and infect plasmid-free cells. In: Nature Microbiology. 17. Juli 2017, doi:10.1038/s41564-017-0009-2 (englisch)
- Robert Gast: Brachten Mikroben aus der Antarktis die ersten Viren hervor? auf: Spektrum.de, 22. August 2017.
- Fatin Iffah Rasyiqah Mohamad Zoolkefli, Kazuki Moriguchi, Yunjae Cho, Kazuya Kiyokawa, Shinji Yamamoto, Katsunori Suzuki: Isolation and Analysis of Donor Chromosomal Genes Whose Deficiency Is Responsible for Accelerating Bacterial and Trans-Kingdom Conjugations by IncP1 T4SS Machinery, in: Front. Microbiol., Band 21, 20. Mai 2021, S. 971, doi:10.3389/fmicb.2021.620535, ISSN 1664-302X. Dazu:
- Mutant Genes Can Promote Genetic Transfer Between Totally Different Types of Organisms, auf: SciTechDaily vom 10. Juli 2021
- S. Weidner, B. Baumgarth u. a.: Genome Sequence of Sinorhizobium meliloti Rm41. In: Genome Announcements. 1, 2013, S. e00013–e00012, doi:10.1128/genomeA.00013-12.
- T. Kenri, A. Horino u. a.: Complete Genome Sequence of Mycoplasma pneumoniae Type 2a Strain 309, Isolated in Japan. In: Journal of Bacteriology. 194, 2012, S. 1253, doi:10.1128/JB.06553-11.
- E. S. Egan, A. Løbner-Olesen, M. K. Waldor: Synchronous replication initiation of the two Vibrio cholerae chromosomes. In: Curr Biol. 14(13), 13. Jul 2004, S. R501–R502.
- Peter W. Harrison, Ryan P. J. Lower u. a.: Introducing the bacterial ‘chromid’: not a chromosome, not a plasmid. In: Trends in Microbiology. 18, 2010, S. 141, doi:10.1016/j.tim.2009.12.010.