Kauffmann-White-Schema
Das Kauffmann-White-Schema (aktuell korrekter, aber unüblicher Name: White-Kauffmann-Le Minor-Schema) ist ein in der Bakteriologie verwendetes Klassifizierungssystem für Vertreter der Enterobakterien-Gattung Salmonella. Es erlaubt die serologische Einordnung von Varietäten und Serotypen (auch „Serovare“ genannt). Im vollständigen Kauffmann-White-Schema sind über 2500 verschiedene Serovare von Salmonella klassifiziert. Salmonellen, die eng mit Arten aus der Gattung Escherichia verwandt sind, sind Pathogene und können sowohl Tiere als auch über die Nahrungskette Menschen infizieren. Infektionskrankheiten mit diesem Übertragungsweg werden auch als Zoonosen bezeichnet.
Geschichte
Im Jahr 1880 entdeckten Karl Joseph Eberth und Robert Koch den Erreger des menschlichen Typhus (damals Eberthella typhosa, heute Salmonella typhi genannt), 1885 identifizierte Daniel Elmer Salmon, nach dem die Gattung Salmonella benannt wurde, den Erreger der „Schweinecholera“ (S. choleraesuis). Bei Tieren wurden weitere Salmonella-Arten (etwa S. typhimurium (Mäusetyphus) und S. abortusovis (Abort des Schafs)) gefunden. Mit den klassischen Methoden der Mikrobiologie – beispielsweise biochemische Charakteristika wie Unterschiede in der Verwertung verschiedener Zucker (siehe auch Bunte Reihe) – ließen sich diese Erreger aufgrund ihrer engen Verwandtschaft nicht voneinander unterscheiden. Nur anhand serologischer Merkmale war eine Klassifizierung dieser Erregergruppe möglich.
1926 veröffentlichte der britische Bakteriologe Philip Bruce White ein Schema zur Klassifikation von Salmonellen auf serologischer Basis,[1] das vom deutsch-dänischen Bakteriologen Fritz Kauffmann von 1933 bis 1978[2] ausgebaut und erweitert wurde. Diese Erweiterung findet auch heute noch statt, im Abstand von einigen Jahren publiziert der französische Bakteriologe Michel Popoff eine Aktualisierung des Schemas. Die letzte Aktualisierung wurde 2007 veröffentlicht.[3] Zwischen zehn und 20 neue Serotypen werden jährlich in diesen Aktualisierungen neu erfasst und beschrieben. In Zusammenarbeit mit der Weltgesundheitsorganisation WHO gibt Popoff in größeren Zeitabständen aktualisierte Kauffmann-White-Schemata in Buchform heraus.[4]
Nomenklatur der Salmonellen
Siehe auch: Hauptartikel Salmonella
Die Nomenklatur der Salmonella-Arten ist sehr komplex. Zuerst wurden Salmonellen nach klinischen Gesichtspunkten (Name der Krankheit und des Wirts) benannt. Als erkannt wurde, dass die Wirtsspezifität mancher Arten nicht existiert – S. typhimurium und S. choleraesuis sind auch für den Menschen pathogen – benannte man neue Serovare als eigenständige Salmonella-Arten nach dem Ort, an dem der erste Stamm der neuen Art isoliert wurde. Im Jahr 2005 entschied der Beschlussfassungsausschuss (Judicial Commission) des International Committee on Systematics of Prokaryotes (ICSP), dass die Gattung Salmonella aus zwei Arten besteht, S. enterica und S. bongori, wobei S. enterica in zahlreiche Unterarten untergliedert wurde.[5][6]
Diese formale, von mikrobiologischen Systematikern erstellte Nomenklatur steht nicht in Einklang mit der traditionellen Systematik der Spezies Salmonella und mit der Kauffmann’schen Artbenennung aufgrund der Serovare. Mit diesem Benennungsprinzip sind jedoch die Fachärzte für Mikrobiologie und Infektiologen seit Jahrzehnten vertraut, so dass diese eigentlich falsche Nomenklatur noch heute weit verbreitet ist und auch in den Beispielen im folgenden Artikel verwendet wird.
Grundlagen des Kauffmann-White-Schemas
Das Prinzip des Kauffmann-White-Schemas
Bei der Erstellung eines Kauffmann-White-Schemas mit beispielsweise drei verschiedenen Salmonella-Stämmen stehen drei gegen diese Stämme gerichtete Antiseren als Nachweisreagenzien zur Verfügung. In einer ersten Versuchsreihe werden die Reaktionen der Antiseren gegen den jeweiligen Ursprungsstamm und gegen die beiden anderen Stämme quantifiziert. In einer zweiten Versuchsreihe werden die Antiseren vor der Quantifizierung mit den nicht homologen Stämmen vorbehandelt. Diese Vorbehandlung wird auch Präadsorption genannt. Die Versuchsergebnisse werden tabellarisch zusammengefasst (4+: starke; 2+: mittlere; 0: keine Reaktion):
| Antisera | Stamm 1 | Stamm 2 | Stamm 3 |
|---|---|---|---|
| Anti-1, nicht adsorbiert | 4+ | 2+ | 4+ |
| Anti-2, nicht adsorbiert | 2+ | 4+ | 2+ |
| Anti-3, nicht adsorbiert | 4+ | 2+ | 4+ |
| Anti-1, adsorbiert an Stamm 2 | 2+ | 0 | 2+ |
| Anti-1, adsorbiert an Stamm 3 | 2+ | 0 | 2+ |
| Anti-2, adsorbiert an Stamm 1 | 0 | 2+ | 0 |
| Anti-2, adsorbiert an Stamm 3 | 0 | 2+ | 0 |
| Anti-3, adsorbiert an Stamm 1 | 2+ | 0 | 2+ |
| Anti-3, adsorbiert an Stamm 2 | 0 | 2+ | 0 |
Es bestehen Unterschiede, aber auch Gemeinsamkeiten zwischen den drei Stämmen, Stamm 1 und 3 zeigen identische Reaktionsmuster, sind also serologisch identisch. Stämme 1 und 3 haben mit Bezug auf Stamm 2 sowohl Gemeinsamkeiten, als auch Unterschiede, man könnte also den Stämmen 1 und 3 in einem künstlichen System die Determinanten (oder Faktoren) A und B zuteilen, Stamm 2 die Determinanten B und C. Man könnte diese Faktoren aber auch in einem Alternativschema mit den griechischen Buchstaben α, β, und γ bezeichnen. Sollte ein neu entdeckter Salmonella-Stamm mit einem anderen, komplett neuartigen Antigenmuster in dieses System aufgenommen werden, wird das erweiterte Schema so aussehen:
| Antisera | Stamm 1 | Stamm 2 | Stamm 3 | Stamm 4 |
|---|---|---|---|---|
| Anti-1, nicht adsorbiert | 4+ | 2+ | 4+ | 0 |
| Anti-2, nicht adsorbiert | 2+ | 4+ | 2+ | 0 |
| Anti-3, nicht adsorbiert | 4+ | 2+ | 4+ | 0 |
| Anti-4, nicht adsorbiert | 0 | 0 | 0 | 4+ |
| Anti-1, adsorbiert an Stamm 2 | 2+ | 0 | 2+ | 0 |
| Anti-1, adsorbiert an Stamm 3 | 2+ | 0 | 2+ | 0 |
| Anti-1, adsorbiert an Stamm 4 | 0 | 0 | 0 | 2+ |
| Anti-2, adsorbiert an Stamm 1 | 0 | 2+ | 0 | 0 |
| Anti-2, adsorbiert an Stamm 3 | 0 | 2+ | 0 | 0 |
| Anti-2, adsorbiert an Stamm 4 | 0 | 0 | 0 | 2+ |
| Anti-3, adsorbiert an Stamm 1 | 2+ | 0 | 2+ | 0 |
| Anti-3, adsorbiert an Stamm 2 | 0 | 2+ | 0 | 0 |
| Anti-4, adsorbiert an Stamm 4 | 0 | 0 | 0 | 2+ |
| Anti-4, adsorbiert an Stamm 1 | 0 | 0 | 0 | 2+ |
| Anti-4, adsorbiert an Stamm 2 | 0 | 0 | 0 | 2+ |
| Anti-4. adsorbiert an Stamm 3 | 0 | 0 | 0 | 2+ |
Da die Antigene des Stammes 4 mit keinem Antigen der Stämme 1 bis 3 Kreuzreaktionen zeigen, wird diesem Stamm in diesem hypothetischen Schema die Determinante D (oder im Alternativschema δ) zugewiesen. Weitere neu isolierte Stämme mit differierenden Antigenmustern können in dieses Schema integriert werden.
Das Kauffmann-White-Schema basiert letztendlich auf der empirischen Auswertung von Antigen-Antikörper-Kreuzreaktionen präadsorbierter Antiseren mit den Oberflächenantigenen eines Salmonella-Stamms.
Bezeichnung und Natur der Antigene
Salmonellen besitzen drei verschiedene Oberflächenantigene, die H, O und Vi-Antigene genannt werden. Als diese Bezeichnungen eingeführt wurden, wusste man nichts über die Funktion und genaue Lokalisation dieser Antigene.
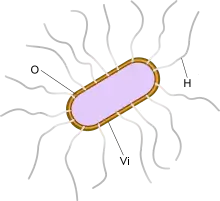
Die Bezeichnung H wurde erstmals zur Beschreibung des Schwarmverhaltens von Proteus mirabilis benutzt. Im halbfesten Agar bewegen sich P. mirabilis und auch Salmonellen dank ihrer peritrichen (d. h. über die ganze Zelloberfläche verteilten) Flagellen schnell fort, das Erscheinungsbild einer schwärmenden Kolonie ähnelt einem Hauch, den der menschliche Atem in Form von Wassertröpfchen beim Anhauchen einer Glasplatte hinterlässt. H-Antikörper sind gegen die Geißelproteine gerichtet. Die meisten Salmonella-Serovare besitzen zwei verschiedene Arten von Geißelantigenen. Diese werden auch als Phasen bezeichnet. In einer Einzelkolonie kommt hauptsächlich eine Phase vor. Mit einer Frequenz von 10−3 bis 10−5 findet in diesem Klon ein Wechsel von der einen zur anderen statt. Das heißt:
- Eine von Hundert bzw. eine von Zehntausend Zellen in einer Kolonie wechselt zur anderen Phase.
- Der Phänotyp der Einzelzelle ist monophasisch, der Genotyp und die Population sind dagegen biphasisch. Nur wenige Serovare (z. B. Salmonella Enteritidis, Salmonella Typhi) sind ausschließlich monophasisch, besitzen also nur ein Geißelantigen. Die H-Antigene der ersten Phase tragen als Bezeichnung Buchstaben, die der zweiten Phase arabische Ziffern, selten auch Buchstaben.
Das O stand ursprünglich nur für ohne Hauch, das bedeutet, diese Bakterien schwärmen nicht auf einer Agarplatte aus. Die O-Antigene wurden zuerst bei unbegeißelten Bakterienstämmen gefunden. O-Antikörper sind gegen die Lipopolysaccharide der Zelloberfläche gerichtet. Man unterscheidet zwischen Haupt-O-Antigenen, die die Gruppenzugehörigkeit der Serovare bestimmen und minor-O-Antigenen. Die minor-O-Antigene kommen meist bei vielen Serovaren vor (Beispielsweise haben alle Salmonella-Stämme aus O-Gruppen A, B, and D das Antigen O:12) und sind also für Klassifizierungszwecke von geringer Bedeutung.
Die Bezeichnung Vi steht für ein zusätzliches Oberflächenantigen, das zunächst primär für Virulenz verantwortlich gemacht wurde; es stellt jedoch einen Spezialfall eines Kapsel-Antigens dar. Die O-Antigen enthaltende Lipopolysaccharidschicht ist mit der dünnen Schicht des Vi-Antigens, eines Polysaccharids überzogen. Vi-Antigene kommen nur bei Salmonella Typhi, Salmonella Paratyphi C und Salmonella Dublin vor und verhindern eine Reaktion der Bakterien mit O-Antikörpern.
Praktische Durchführung der Antigen-Antikörper-Reaktionen
Vor der Durchführung der Antigen-Antikörper-Reaktionen werden Vorversuche durchgeführt, um zu überprüfen, ob unspezifische Agglutinationen stattfinden. Dazu wird auf einem Objektträger etwas Bakterienmaterial in physiologischer Kochsalzlösung verrieben und die Suspension wird vor einem schwarzen Hintergrund (schwarzes Tonpapier, schwarze Kachel) beobachtet. Sofern die Suspension während etwa 5 Minuten milchig trüb bleibt, findet keine Spontanagglutination statt. Um Sicherheit zu schaffen, dass tatsächlich mit einem Salmonella-Stamm gearbeitet wird, kann mit einem omnivalenten Antiserum (einem Antiserum, das alle Antigene erkennt) gegen O-Antigene eine Positivkontrolle durchgeführt werden. Dazu wird etwas Bakterienmaterial mit dem Antiserum verrieben. Nach etwa zwei Minuten sollten auf schwarzem Hintergrund deutlich sichtbare Agglutinate auftreten. Dieses Verfahren wird Objektträgeragglutination genannt.
In der folgenden Bestimmung wird zuerst mit Hilfe gruppenspezifischer O-Antigene in der Objektträgeragglutination die Gruppenzugehörigkeit ermittelt. Danach wird mit Hilfe der H-Antiseren mit der Objektträgeragglutination die vorliegende Geißelphase ermittelt. Hierbei zeigt sich ein Unterschied: Die Antigen-Antikörper-Aggregate sind beim O-Antigen fest und körnig, während sie beim H-Antigen flockig und wenig beständig sind. Ist die erste Phase des H-Antigens ermittelt, erfolgt die Bestimmung der zweiten Phase. Dazu wird eine Schwärmplatte hergestellt, die das entsprechende H-Antiserum enthält. Das Schwärmen der Bakterien, die der ersten Phase angehören, wird dadurch unterdrückt. Nur solche, die in die zweite Phase umgeschaltet haben, können sich unter diesen Bedingungen bewegen. Nach Bebrütung über Nacht kann mit Bakterienmaterial aus der Schwärmzone die zweite Phase durch Agglutination mit geeigneten H-Antiseren bestimmt werden.
Aus der Kombination O-Antigene, H-Antigene der ersten und zweiten Phase kann das Serovar des vorliegenden Salmonella-Stamms aus dem Kauffmann-White-Schema bestimmt werden. Siehe auch[7]
Ein einfaches Kauffmann-White-Schema
In der folgenden Tabelle werden einige wichtige Salmonella-Serovare nach dem Kauffmann-White-Schema klassifiziert, wobei die klassische Nomenklatur benutzt wird:
| „O“-Gruppe | Serovar | „O“ Antigene | „H“ Antigene Phase 1 | „H“ Antigene Phase 2 |
|---|---|---|---|---|
| Gruppe A | Salmonella Paratyphi A | 1, 2, 12 | A | keine Phase 2 |
| Gruppe B | Salmonella Paratyphi B | 1, 4, 5, 12 | B | 1, 2 |
| Gruppe B | Salmonella Typhimurium | 1, 4, 5, 12 | I | 1, 2 |
| Gruppe B | Salmonella Abortusovis | 4, 12 | C | 1,6 |
| Gruppe C | Salmonella Choleraesuis | 6, 7 | C | 1,5 |
| Gruppe C | Salmonella Thompson | 6, 7 | K | 1, 5 |
| Gruppe C | Salmonella Newport | 6, 8 | R | 1, 5 |
| Gruppe C | Salmonella Muenchen | 6, 8 | D | 1, 2 |
| Gruppe D | Salmonella Typhi | 9, 12 | D | keine Phase 2 |
| Gruppe D | Salmonella Enteritidis | 1, 9, 12 | g, m | keine Phase 2 |
| Gruppe D | Salmonella Dublin | 1, 9, 12 | 91 p | keine Phase 2 |
| Gruppe E | Salmonella Anatum | 3, 10 | e, h | 1, 6 |
Auswahl der Antiseren
Der Aufwand, ein vollständiges Lager an Antiseren für die Durchführung eines kompletten Kauffmann-White-Schemas herzustellen und zu verwalten, ist groß. Dies wird vornehmlich in nationalen Referenzlaboren vorgenommen. Die meisten Labore halten nur eine bestimmte Anzahl an Antiseren vorrätig, wobei sich die Auswahl der Antiseren an der wahrscheinlich zu bearbeitenden Salmonellen-Serovare orientiert. Um Resultate zu erzielen, die zwischen den Laboren vergleichbar sind, hat die WHO Standards für die Herstellung der Antiseren vorgeschrieben.[8]
Eine oft anzutreffende Auswahl sieht folgendermaßen aus:
| O-Antisera | H-Antisera |
|---|---|
| Polyvalent-O, Gruppen A–G | Polyvalent-H, spezifisch und unspezifisch |
| 2-O, Gruppe A | Polyvalent-H, unspezifische Faktoren 1,2,5,6,7 |
| 4-O, Gruppe B | a-H (Salmonella Paratyphi A) |
| 6, 7-O, Gruppe C1 | b-H (Salmonella Paratyphi B) |
| 8-O, Gruppe C2 | c-H (Salmonella Paratyphi C) |
| 9-O, Gruppe D | d-H (Salmonella Typhi) |
| 3, 10, 15, 19-O Gruppe E | e,h-H (Salmonella Newport) |
| 11-O, Gruppe F | f,g-H (Salmonella Derby) |
| 13, 22-O, Gruppe G | g,m-H (Salmonella Enteritidis) |
| i-H (Salmonella Typhimurium) | |
| k-H (Salmonella Thompson) | |
| l,v-H (Salmonella London) | |
| m,t-H (Salmonella Oranienburg) | |
| r-H (Salmonella Bovismorbificans) |
Labore, die sich mit Typhuserregern beschäftigen, halten auch Antiseren gegen Vi-Antigene bereit.
Ein Satz „Schnelldiagnostiksera“ (rapid diagnostic sera oder RDS) wird auch angewandt: Er wird für die Bestimmung häufig vorkommender H-Antigene mit Ausnahme von i-H eingesetzt. Sofern die Agglutination mit einem polyvalenten H-spezifischen Antiserum (einem Antiserum, das mehrere H-Antigene erkennt) und einem unspezifischen Antiserum positiv verläuft, werden die drei RDS Antisera eingesetzt, um das H-Antigen zu identifizieren. Aus dem Muster Agglutination-Nichtagglutination kann das jeweilige H-Antigen bestimmt werden.
| Antigen | RDS1 | RDS2 | RDS3 |
|---|---|---|---|
| b | Agglutination | Agglutination | keine Agglutination |
| d | Agglutination | keine Agglutination | Agglutination |
| E | Agglutination | Agglutination | Agglutination |
| G | keine Agglutination | keine Agglutination | Agglutination |
| k | keine Agglutination | Agglutination | Agglutination |
| L | keine Agglutination | Agglutination | keine Agglutination |
| r | Agglutination | keine Agglutination | keine Agglutination |
- E = polyvalent für die Antigene eh, enx, etc.
- G = polyvalent für die Antigene gm, gp, etc.
- L = polyvalent für die Antigene lv, lw, etc.
Neuere Entwicklungen
Das konventionelle Verfahren zur Bestimmung der Zusammensetzung der Oberflächenantigene ist zeit- und materialaufwändig. Die gleichzeitige Detektion von O- und H-Antigenen ist wünschenswert. Dazu wurden nach dem Kauffmann-White-Schema ausgewählte Antikörper in einem Protein-Microarray auf einen Objektträger aufgebracht und mit Fluoreszenzfarbstoffen markierten Salmonella-Zellen behandelt.[9] Diese Miniaturisierung ist jedoch noch nicht Routine im Diagnostiklabor.
Literatur
- Patrick A.D. Grimont & François-Xavier Weill: Antigenic Formular of the Salmonella Serovars (englisch) 9. Auflage. 2007, herausgegeben vom WHO Collaborating Centre for Reference and Research on Salmonella und dem Institut Pasteur
Weblinks
- Webseite des Nationalen Referenzzentrums für Salmonellen und andere bakterielle Enteritiserreger, RKI Bereich Wernigerode
Einzelnachweise
- P.B. White: Further Studies of the Salmonella Group. Great Britain Medical Research Council 103, (Her Majesty’s Stationary Office), 3-160, 1926.
- F. Kauffmann: Das Fundament, Munksgaard, Kopenhagen, 1978.
- M. Y. Popoff, J. Bockemühl, L. L. Gheesling: Supplement 2002 (no. 46) to the Kauffmann-White scheme. In: Research in microbiology. Band 155, Nummer 7, September 2004, S. 568–570, ISSN 0923-2508. doi:10.1016/j.resmic.2004.04.005. PMID 15313257.
- M. Y. Popoff: Antigenic formulas of the Salmonella serovars. 8th ed. World Health Organization Collaborating Center for Reference and Research on Salmonella, Institut Pasteur, Paris, 2001
- Judicial Commission of the International Committee on Systematics of Prokaryotes. The type species of the genus Salmonella Lignieres 1900 is Salmonella enterica (ex Kauffmann and Edwards 1952) Le Minor and Popoff 1987, with the type strain LT2T, and conservation of the epithet enterica in Salmonella enterica over all earlier epithets that may be applied to this species. Opinion 80. In: International journal of systematic and evolutionary microbiology. Band 55, Pt 1 Januar 2005, S. 519–520, ISSN 1466-5026. doi:10.1099/ijs.0.63579-0. PMID 15653929.
- B. J. Tindall, P. A. Grimont, G. M. Garrity, J. P. Euzéby: Nomenclature and taxonomy of the genus Salmonella. In: International journal of systematic and evolutionary microbiology. Band 55, Pt 1 Januar 2005, S. 521–524, ISSN 1466-5026. doi:10.1099/ijs.0.63580-0. PMID 15653930.
- Ruhr-Universität Bochum: Skript zur praktischen Durchführung der Antigen-Antikörperreaktionen (Memento vom 8. Februar 2008 im Internet Archive)
- M. Y. Popoff: Guidelines for the preparation of Salmonella antisera. 5th ed. World Health Organization Collaborating Centre for Reference and Research on Salmonella, Institut Pasteur, Paris 2002
- Development of a novel protein microarray method for serotyping Salmonella enterica strains. In: Journal of clinical microbiology. Band 43, Nummer 7, Juli 2005, S. 3427–3430, ISSN 0095-1137. doi:10.1128/JCM.43.7.3427-3430.2005. PMID 16000469. PMC 1169117 (freier Volltext).