Bunte Reihe (Labor)
Bei der sogenannten Bunten Reihe handelt es sich um eine labortechnische Methode zur Bestimmung (Identifizierung) von Bakterien und Hefen anhand verschiedener Merkmale. Dabei wird auf bestimmte Enzymaktivitäten, den Abbau von Substraten, die Bildung von Stoffwechselprodukten und andere Fähigkeiten (beispielsweise aktive Bewegung) geprüft. Die Bunte Reihe besteht aus Reagenzröhrchen, gegebenenfalls auch Petrischalen, die mit verschiedenen Nährmedien beschickt sind. Die Nährmedien enthalten Reagenzien und Indikatoren zum Nachweis von bestimmten Eigenschaften, die eine Bestimmung von Bakterien auf Ebene der Gattung oder sogar der Art ermöglichen. Beimpft werden die Röhrchen mit einer Reinkultur des zu bestimmenden Bakteriums. Die Ergebnisse (positiv oder negativ) werden mit einer Tabelle verglichen und so kann man mit einer bestimmten Wahrscheinlichkeit die unbekannte Reinkultur einer Spezies zuordnen.[1]
Kleine Bunte Reihe
Die älteste und kompakteste Version ist die traditionelle kleine Bunte Reihe, die vor allem zur Bestimmung von Enterobakterien dient. Sie enthält: Kligler-Agar, Harnstoff-Agar, MIO-Röhrchen und Simmons Citrat-Agar. Die Nährmedien werden je nach Art des Mediums verschieden beimpft. In den meisten Fällen sind damit die häufigsten Enterobakterien schnell zu bestimmen, jedoch gibt es Ausnahmen und Unregelmäßigkeiten. Genauer ist die Untersuchung mittels eines Systems, bei dem 20 bis 30 biochemische Untersuchungen innerhalb von 24 Stunden durchgeführt und visuell oder elektronisch ausgewertet werden können (zum Beispiel Analytical Profile Index API-20E).
Zur Reinheitskontrolle wird auf Columbia-Blutagar ausgestrichen um eine Kontamination z. B. durch grampositive Erreger auszuschließen. Die Kolonien müssen wie die zur Beimpfung der Bunten Reihe verwendete aussehen und es dürfen keine anderen Kolonien auf dem Agar wachsen.
Kligler-Agar
Hierbei handelt es sich um ein Nährmedium zur Identifizierung gramnegativer Darmbakterien nach Kligler (1917, 1918), beispielsweise Salmonella, Shigella, Escherichia, Enterobacter und Proteus. Es dient dem Nachweis des Lactose-Abbaus durch das Enzym β-Galactosidase, der Verwertbarkeit der vorhandenen Kohlenhydrate in einer Gärung, erkennbar an der Säurebildung beim Abbau und ggf. an Gasbildung (CO2), sowie der Bildung von Schwefelwasserstoff (H2S).[2] Statt des klassischen Kligler-Agars mit zwei enthaltenen Zuckern wird häufig auch der TSI-Agar (triple sugar iron agar, Dreizucker-Eisen-Agar) verwendet, der vergleichbare Ergebnisse liefert.[3] Die H2S-Bildung kann auch mit Hilfe des SIM-Agars (Schwefelwasserstoff-Bildung, Indol-Bildung, Motilität) nachgewiesen werden.[1] Die Schwefelwasserstoff-Bildung ist typisch für einige Arten in den Gattungen Citrobacter, Proteus (bzw. Cosenzaea) und Salmonella.[4]
Wirkungsweise
Das Nährmedium wird als Schrägagarröhrchen verwendet, bei der Inokulation wird zunächst senkrecht in den Agar gestochen und danach auf der Schrägfläche in einer mäanderförmigen Linie ausgestrichen. Durch den Glucoseabbau entsteht Säure und bei pH < 7 wird der pH-Indikator Phenolrot gelb. Durch die Entstehung alkalischer Stoffwechselprodukte aus dem Peptonabbau sowie durch Oxidation der Säuren an der Schrägfläche in Gegenwart von Luftsauerstoff wird der Nährboden im oberen Bereich re-alkalisiert (der pH-Wert steigt über 7) und dieser Teil des Mediums wird rot. Falls neben Glucose auch Lactose abgebaut wird (β-Galactosidase-Aktivität positiv), entsteht dadurch zusätzliche Säure, dann bleibt der gesamte Nährboden durch die größere Säuremenge gelb. Geschieht dies ohne Verwendung von Sauerstoff in einer Gärung, so bildet sich CO2, das als kleine bis große Gasblasen und Risse im Nährboden sichtbar wird. Aus der schwefelhaltigen anorganischen Verbindung Natriumthiosulfat gebildetes H2S reagiert mit dem enthaltenen Eisensalz zu Eisensulfid, das als schwarzer Niederschlag ausfällt.[1][4]
Typische Zusammensetzung
Der Nährboden besteht meistens aus (Angaben in Gramm pro Liter):[2]
- Pepton aus Casein 15,0
- Pepton aus Fleisch 5,0
- Fleischextrakt 3,0
- Hefeextrakt 3,0
- Natriumchlorid 5,0
- Lactose (Substrat) 10,0
- Saccharose (Substrat) 10,0
- D-Glucose (Substrat) 1,0
- Ammoniumeisen(III)-citrat (Reagenz) 0,5
- Natriumthiosulfat (Substrat) 0,5
- Phenolrot (pH-Indikator) 0,024
- Agar-Agar 12,0
Harnstoff-Agar
Mit Hilfe des Harnstoff-Agars nach Christensen (1946) gelingt die Unterscheidung von Bakterien, die über das Enzym Urease verfügen, von denen, die Urease-negativ sind. Urease ist ein Enzym, das Harnstoff hydrolysieren kann.[5] Vertreter der Gattungen Klebsiella, Proteus und Yersinia verfügen über die Urease,[4] während beispielsweise Escherichia, Providencia, Salmonella, Serratia und Shigella Urease-negativ sind. Bei den Gattungen Citrobacter und Enterobacter gibt es sowohl Urease-positive wie -negative Arten.[5]
Wirkungsweise
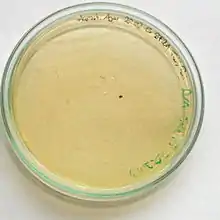

Der Harnstoff-Agar kann in Reagenzröhrchen als Schrägagarröhrchen oder in Petrischalen verwendet werden, dabei wird eine relativ große Menge der Reinkultur ausgestrichen.[5] Das fertige Nährmedium enthält Harnstoff, wird dieser durch ein Bakterium, das das Enzym Urease besitzt, verwertet, so entsteht Kohlenstoffdioxid und Ammoniak. Durch den Ammoniak wird das Medium alkalisch (pH > 7) und der enthaltene pH-Indikator Phenolrot zeigt dies durch Farbumschlag von Gelb nach Rotviolett an. Verfügt das Bakterium nicht über das Enzym Urease, erfolgt keine Harnstoffspaltung und das Medium bleibt gelblich, bedingt durch den enthaltenen pH-Indikator. Auch Urease-negative Bakterien können auf dem Medium wachsen, da es Glucose als Energie- und Kohlenstoffquelle enthält, aber ihr Wachstum führt nicht zu einer Farbveränderung des Mediums.[1]
Typische Zusammensetzung
Der Nährboden besteht meistens aus (Angaben in Gramm pro Liter):[5]
- Pepton aus Fleisch 1,0
- D-Glucose 1,0
- Natriumchlorid 5,0
- Kaliumdihydrogenphosphat 2,0
- Harnstoff (Substrat) 20,0
- Phenolrot (pH-Indikator) 0,012
- Agar-Agar 12,0
MIO-Röhrchen

Mit Hilfe des MIO-Agars lassen sich Bakterien aus der Ordnung der Enterobacterales (Enterobakterien) weiter voneinander unterscheiden. Die Abkürzung MIO resultiert aus drei Merkmalen, die man mit diesem Medium nachweisen kann: (1) aktive Beweglichkeit (Motilität, im Englischen motility). Aktive Bewegung wird bei Vertretern der Enterobacterales durch Flagellen verursacht. (2) Bildung von Indol aus Tryptophan durch das Enzym Tryptophanase. (3) Bildung von Putrescin aus Ornithin durch das Enzym Ornithindecarboxylase (ODC). Beispielsweise ist Escherichia positiv für Motilität und Indobildung, aber negativ für ODC, Enterobacter hingegen ist positiv für Motilität und ODC, aber negativ für Indolbildung.[4]
Wirkungsweise
Der MIO-Agar wird in Reagenzröhrchen als Hochschichtröhrchen verwendet und durch Einstich mittels einer Impfnadel mit der Reinkultur beimpft. Das fertige Nährmedium ist ein so genannter Weichagar, der Agar-Agar in geringerer Konzentration als üblich enthält, nämlich nur 1 – 4 g/L.[6] In einem Agargel mit so geringer Agar-Konzentration können sich begeißelte Bakterien aktiv schwimmend ausbreiten. Dies führt zu einer Trübung des Mediums durch die Bakterien auch außerhalb des Stichkanals, während bei Bakterien ohne aktive Bewegung nur Wachstum und damit Trübung im Impfkanal zu beobachten ist.[1]
Weiterhin ist im Medium die Aminosäure LTryptophan als Substrat enthalten, manche Bakterienarten verfügen über das Enzym Tryptophanase und können somit Tryptophan zu Indol, Pyruvat und Ammoniak abbauen. Das dabei gebildete Indol wird im Indol-Test mit Kovacs-Reagenz nachgewiesen. Dieser Test erfolgt jedoch erst nach der Inkubation und Auswertung der anderen Merkmale, indem man das Reagenz hinzugibt. Eine kirschrote Färbung der Lösung zeigt Indolbildung an (Indol-positive Bakterien), bleibt die Lösung dagegen farblos oder verfärbt sich gelblich, ist das Bakterium Indol-negativ.[1]
Das Medium enthält außerdem noch die Aminosäure L-Ornithin als Substrat und den pH-Indikator Bromkresolpurpur. Wird Ornithin durch ein Bakterium, das das Enzym Ornithindecarboxylase (ODC) besitzt, verwertet, so entsteht durch Decarboxylierung Putrescin (Butan-1,4-diamin). Durch dieses Produkt wird das Medium alkalisch (pH > 7) und der enthaltene pH-Indikator Bromkresolpurpur zeigt dies durch Farbumschlag von Gelb nach Purpur bis Violett an, der ODC-Test ist positiv. Verfügt das Bakterium nicht über das Enzym Ornithindecarboxylase, wird Ornithin nicht abgebaut und das Medium erscheint gelb, bedingt durch den enthaltenen pH-Indikator und die aus Glucose gebildeten Säuren.[4]
Simmons Citrat-Agar
Mit Hilfe des Citrat-Agars nach Simmons (1926) können Bakterien identifiziert werden, die mit Citrat als einziger Energiequelle wachsen können, indem sie durch dessen Abbau Energie gewinnen. Dies trifft auf einige Vertreter der Enterobacteriaceae zu, sowie auf einige Pilze.[7] In der Familie Enterobacteriaceae verhalten sich die meisten Vertreter der Gattungen Citrobacter, Enterobacter und Klebsiella Citrat-positiv, gleiches gilt für Serratia (Familie Yersiniaceae), während Escherichia und Shigella Citrat-negativ sind. Arten bzw. Serotypen von Salmonella verhalten sich unterschiedlich.[4][7]
Wirkungsweise
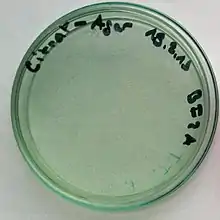
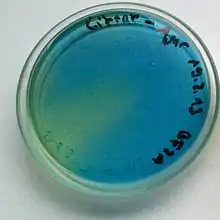
Der Citrat-Agar kann in Reagenzröhrchen als Schrägagarröhrchen oder in Petrischalen verwendet werden und wird mit einer Reinkultur beimpft. Das fertige Nährmedium enthält Natriumcitrat als einzige Energiequelle, den pH-Indikator Bromthymolblau und als Stickstoffquelle lediglich die anorganische Verbindung Ammoniumdihydrogenphosphat. Wenn ein Mikroorganismus Citrat verwerten kann (er verfügt dann über das Enzym Citrat-Lyase), wird dabei Citrat zu Acetyl-CoA und Oxalacetat umgewandelt. Oxalacetat wird weiter zu Pyruvat und Kohlenstoffdioxid (CO2) abgebaut. Aus dem CO2 entsteht mit Wasser auch Hydrogencarbonat (HCO3−) und weiterhin wird Ammoniak (NH3) aus der anorganischen Stickstoffquelle freigesetzt. Diese Reaktionen führen zu einer Alkalisierung (pH > 7) und der im Nährmedium enthaltene pH-Indikator Bromthymolblau zeigt dies durch Farbumschlag von grün nach blau an.[8] Kann das Bakterium kein Citrat verwerten, erfolgt keine pH-Erhöhung und das Medium bleibt grün, bedingt durch den enthaltenen pH-Indikator. In diesem Fall erfolgt aber auch kein Wachstum, da außer Citrat keine anderen Energiequellen enthalten sind.[4]
Typische Zusammensetzung
Der Nährboden besteht meistens aus (Angaben in Gramm pro Liter):[7]
- Ammoniumdihydrogenphosphat 1,0
- Dikaliumhydrogenphosphat 1,0
- Natriumchlorid 5,0
- Magnesiumsulfat 0,2
- Natriumcitrat (Substrat) 2,0
- Bromthymolblau (pH-Indikator) 0,012
- Agar-Agar 13,0
Erweiterte Bunte Reihe
Da man mit der kleinen Bunten Reihe nicht immer eine ausreichende Identifizierung der unbekannten Reinkultur erhält, können nach dem gleichen Prinzip weitere Tests durchgeführt werden. Zur Unterscheidung der Enterobakterien werden insbesondere die IMViC-Reaktionen durchgeführt, dabei handelt es sich um den bereits erwähnten Indol-Test, die Methylrot-Probe auf Säurebildung, den Voges-Proskauer-Test auf Acetoin-Bildung und den ebenfalls schon beschriebenen Nachweis der Citrat-Verwertung.[9]
Außerdem wird für die weitere Differenzierung der Vertreter der Enterobakterien auch der SIM-Agar verwendet, mit dem drei Merkmale gleichzeitig untersucht werden können: Die Bildung von Schwefelwasserstoff (H2S), die Indol-Bildung und die aktive Bewegung (Motilität) von Bakterien.[1]
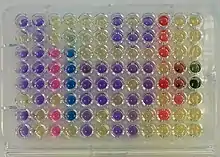
Darüber hinaus gibt es noch zahlreiche weitere Tests, die häufig als Kombination von vielen miniaturisierten Reaktionsgefäßen in einem System angeordnet sind, um so eine schnelle Untersuchung durchführen zu können, wie zum Beispiel das Testsystem Analytical Profile Index API-20E. Einige der dort oder in vergleichbaren Systemen verwendeten Testreaktionen werden auch noch im herkömmlichen Maßstab, also in Reagenzröhrchen oder Mikroreaktionsgefäßen angesetzt und untersucht und sollen anhand einiger Beispiele beschrieben werden.
Neben den Mikroreaktionsgefäßen lassen sich Keime auch mit sehr geringem Substrateinsatz (100 µl) und damit Kosten in Mikrotiterplatten identifizieren. Auch Kombireaktionen wie beim Kligler-Agar sind hier möglich.
Test auf Nitratreduktion

Mit diesem Test wird untersucht, ob ein Bakterium Nitrat zu Nitrit oder weiter zu elementarem Stickstoff (Distickstoff N2) reduzieren kann. Die Reduktion von Nitrat zu Distickstoff wird als Denitrifikation bezeichnet und ist ein Schritt im Stickstoffkreislauf. Bei den Bakterien der Familie der Enterobacteriaceae wird Nitrat lediglich bis zur Stufe des Nitrits reduziert.[4]
Wirkungsweise
Für den Nachweis der Nitratreduktion wird ein flüssiges Komplettmedium (Nährbouillon) beimpft, das neben den das Wachstum ermöglichenden Bestandteilen wie Glucose und Pepton auch 1 g/L Kaliumnitrat (KNO3) enthält. Nach der Inkubation werden Sulfanilsäurelösung und 1-Naphthylaminlösung hinzugegeben. Mit diesen Reagenzien wird gebildetes Nitrit durch Rotfärbung nachgewiesen. Verläuft dieser Test positiv, hat das Bakterium Nitrat zu Nitrit reduziert.
Falls sich keine Verfärbung beobachten lässt, wird noch eine geringe Menge Zinkstaub hinzugefügt. Wenn das Bakterium Nitrat nicht verwerten konnte, ist dieses noch im Medium vorhanden und wird nun zu Nitrit reduziert, was durch die Rotfärbung erkennbar ist. In diesem Fall ist das Bakterium als negativ in Bezug auf die Nitratreduktion zu beurteilen. Falls auch nach Zugabe des Zinks keine Farbveränderung zu beobachten ist, wurde das Nitrat zu elementarem Stickstoff reduziert, der gegebenenfalls als Gas in einem Gärröhrchen aufgefangen werden kann. Das Bakterium ist somit als positiv in Bezug auf die Nitratreduktion zu beurteilen.[1]
Methylrot-Probe

Mit diesem Test ist eine weitere Differenzierung der Vertreter der Enterobakterien möglich, es wird untersucht, ob ein Bakterium im fermentativen Abbau von Glucose große Mengen an Säure produziert. Die gramnegativen Darmbakterien lassen sich hinsichtlich ihrer Gärungsprodukte in zwei Gruppen einteilen. Das ist zum einen der Escherichia-coli-Typ, der vorwiegend verschiedene organische Säuren aus Glucose bildet, in der Gemischten Säuregärung und zum anderen der Enterobacter-Typ, der aus Glucose in der 2,3-Butandiolgärung vor allem Butandiol (und Acetoin als Zwischenprodukt) sowie Gas (CO2 und H2) bildet, aber nur wenig Säure. Die Säurebildung wird mit der Methylrot-Probe (auch MR-Test genannt) nachgewiesen, während die Bildung von Acetoin als Vorstufe des 2,3-Butandiols im Voges-Proskauer-Test (auch als VP-Test abgekürzt) nachweisbar ist.[4][9]
Wirkungsweise
Für diesen Nachweis wird ein flüssiges Medium (Nährbouillon) beimpft, das nur die für das Wachstum notwendigen Bestandteile Glucose und Pepton enthält, sowie Kaliumdihydrogenphosphat als Puffersubstanz. Nach der Inkubation wird das Medium aufgeteilt, mit einem Teil wird der Voges-Proskauer-Test durchgeführt, mit dem anderen die Methylrot-Probe. Dazu wird eine ethanolische Lösung des pH-Indikators Methylrot hinzugegeben. Große Mengen an gebildeter Säure senken den pH-Wert deutlich ab, der Indikator zeigt dies durch einen Farbumschlag nach Rot an, der pH-Wert liegt dann unter pH 4,5. Der Test fällt somit positiv aus (MR-positiv) und das Enterobakterium gehört zur Gruppe der Gemischten Säuregärer. Falls das Bakterium keine oder wenig Säure gebildet hat, zeigt der Indikator eine gelbe Farbe (pH > 4,5), das Testergebnis ist MR-negativ und das Darmbakterium gehört zur Gruppe der Butandiol-Gärer, was mit dem Voges-Proskauer-Test bestätigt werden kann.[1] Eine orange Verfärbung des Indikators (pH-Wert im Bereich 5 bis 6) ist als negatives Ergebnis zu deuten, muss aber mit einem positiven VP-Test bestätigt werden.
Zusammenfassung weiterer Tests
Weitere innerhalb einer Bunten Reihe angewendete Untersuchungen sind Tests auf Verwertung verschiedener Kohlenhydrate unter Säurebildung im OF-Test (Oxidations-Fermentations-Test, auch als Hugh-Leifson-Test bezeichnet) sowie auf Bildung von Exoenzymen. Dazu gehört der Test auf Gelatineverflüssigung, bei dem proteolytische Enzyme das Protein Gelatine hydrolytisch abbauen können; der Test auf Stärke-Hydrolyse, bei dem Exo-Amylasen der Mikroorganismen nachgewiesen werden; der Test auf Cellulose-Hydrolyse, mit dem Exo-Cellulasen erkannt werden oder den DNase-Test (auch DNAse-Test genannt), der das Exoenzym Desoxyribonuklease nachweist, mit dem Mikroorganismen DNA abbauen können.[1]
Weitere Tests basieren auf dem Nachweis von Endoenzymen, also Enzymen, die innerhalb der Zelle gebildet werden und dort Stoffwechselvorgänge katalysieren. Neben der schon bei der Beschreibung des MIO-Röhrchen erwähnten Ornithindecarboxylase (ODC) gibt es den Nachweise der Lysindecarboxylase (LDC), hierbei sind die Bakterien in der Lage, die Aminosäure L-Lysin zu decarboxylieren, wobei Kadaverin (1,5-Diaminopentan) entsteht. Beim Test auf Arginindihydrolase (ADH) finden mehrere Reaktionen durch bakterielle Enzyme statt, durch die die Aminosäure L-Arginin zu Ornithin, Ammoniak und Kohlenstoffdioxid abgebaut wird.[1] Der Nachweis von ADH, LDC und ODC hilft bei der Bestimmung der Enterobakterien.[4]
Für die Bestimmung der Vertreter der Familie der Morganellaceae ist darüber hinaus noch der Nachweis des Enzyms Phenylalanin-Desaminase (PAD) von großer Bedeutung. Nur die Vertreter der Gattungen Morganella, Providencia und Proteus verfügen über dieses Enzym. Bei ihnen ist ebenfalls der Nachweis der Tryptophan-Desaminase (TDA) üblich. Die Enzyme katalysieren die Desaminierung der Aminosäuren L-Phenylalanin bzw. L-Tryptophan, die Abbauprodukte werden durch Zusatz von Eisen(III)-chlorid-Lösung detektiert, es zeigt sich eine dunkelgrüne (PAD) bzw. rot-braune (TDA) Färbung.[10]
Auch die medizinisch wichtigsten Vertreter der Candida (Pilze) sind neben der Assimilation in der Lage die üblich verwendeten Kohlehydrate zu fermentieren. Diese können ebenfalls mit einer kurzen Bunten-Reihe identifiziert werden. So kann Candida albicans von apathogenen Sacchamyceten und anderen Arten wie Cryptococcus neoformans (Fermentation -, Urease +) unterschieden werden.[11]
Wahrscheinlichkeitsberechnung
Berechnung der Wahrscheinlichkeit einer unbekannten Spezies.
Datenbasis
Die Tabelle für die Auswertung (Datenbasis) enthält alle Spezies, welche mit dieser Datenbasis identifiziert werden können mit den Prozentwerten der positiven Reaktion für jede Spezies und dem der jeweiligen Substrate.[12] Die Prozentwerte für die Einzelsubstrate erhält man durch Testung von Reinkulturen exakt bestimmter Spezies oder mit Referenzstämmen z. B. von der DSMZ.
Beispiel einer einfachen Datenbasis
| Spezies | Citrat | H2S | Indol | Laktose | Urease |
|---|---|---|---|---|---|
| Citrobacter freundii |
90 | 95 | 30 | 40 | 35 |
| Escherichia coli |
2 | 4 | 95 | 90 | 1 |
| Enterobacter cloacae |
99 | 1 | 2 | 94 | 55 |
| Proteus mirabilis | 60 | 97 | 3 | 2 | 90 |
Wahrscheinlichkeit einer unbekannten Spezies
Beispiel:
| Spezies | Citrat | H2S | Indol | Laktose | Urease |
|---|---|---|---|---|---|
| unbekannt | + | - | + | + | - |
Für die Berechnung der Wahrscheinlichkeit einer unbekannten Spezies, werden zunächst die absoluten Wahrscheinlichkeiten (W) aller in der Datenbasis vorkommenden Spezies berechnet. Hierfür wird bei positiven Reaktionen mit der Wahrscheinlichkeit P für positive Reaktionen multipliziert, bei negativen Reaktionen der unbekannten Spezies mit 1-P multipliziert.
| Spezies | Citrat + | H2S - | Indol + | Laktose + | Urease - | W | %ID |
|---|---|---|---|---|---|---|---|
| Citrobacter freundii | 0.9 | * 0.05 | * 0.3 | * 0.4 | * 0.65 | = 0.00351 | 0.00351/0.028054566 = 12,51 % |
| Escherichia coli | 0.02 | * 0.96 | * 0.95 | * 0.9 | * 0.99 | = 0.01625184 | 0.01625184/0.028054566 =57,93 % |
| Enterobacter cloacae | 0.99 | * 0.99 | * 0.02 | * 0.94 | * 0.45 | = 0.008291646 | 0.008291646/0.028054566 = 29,56 % |
| Proteus mirabilis | 0.6 | * 0.03 | * 0.03 | * 0.02 | * 0.1 | = 0.00000108 | 0.00000108/0.028054566 = 0,0 % |
| Summe | 0.028054566 | 100 % |
Für die Berechnung der relativen Wahrscheinlichkeit (%ID) einer Spezies, wird die absolute Wahrscheinlichkeit einer Spezies durch die Summe aller in der Datenbasis vorkommenden Spezies geteilt. Die unbekannte Spezies ist hier also mit der Wahrscheinlichkeit 57,9 % E. coli, die Qualität der Identifizierung schlecht. Ursache für eine niedrige %ID sind z. B. es wurde keine Reinkultur verwendet oder die unbekannte Spezies ist nicht in der Datenbank vorhanden.
Weitere Kennzeichen der Qualität einer Identifizierung sind die Anzahl der widersprechenden Reaktionen und der Abstand der identifizierten Spezies vom nächsten Taxon.[13] In diesem Beispiel ist Citrat+ widersprechend für Escherichia coli. Wiederholt man die Berechnung mit Citrat-, erhält man eine Identifizierung von 0.79634016/0.796814634 = 99,9 %.
Einzelnachweise
- Roland Süßmuth, Jürgen Eberspächer, Rainer Haag, Wolfgang Springer: Biochemisch-mikrobiologisches Praktikum. 1. Auflage. Thieme Verlag, Stuttgart/New York 1987, ISBN 3-13-685901-4.
- Technische Informationen Kligler-Agar zur Identifizierung gramnegativer Darmbakterien (Eisen-Zweizucker-Agar nach Kligler). In: Website Merck Millipore. Abgerufen am 1. Januar 2020.
- Technische Informationen Triple Sugar Iron Agar (Eisen-Dreizucker-Agar). In: Website Merck Millipore. Abgerufen am 1. Januar 2020.
- Michael T. Madigan, John M. Martinko, Jack Parker: Brock Mikrobiologie. Deutsche Übersetzung herausgegeben von Werner Goebel, 1. Auflage. Spektrum Akademischer Verlag GmbH, Heidelberg/Berlin 2000, ISBN 978-3-8274-0566-1.
- Technische Informationen Harnstoff-Agar (Basis) nach Christensen (nach ISO 6579, ISO 10273, ISO 19250, ISO 21567). In: Website Merck Millipore. Abgerufen am 1. Januar 2020.
- Eckhard Bast: Mikrobiologische Methoden: Eine Einführung in grundlegende Arbeitstechniken. 2. Auflage. Spektrum Akademischer Verlag GmbH, Heidelberg/Berlin 2001, ISBN 978-3-8274-1072-6.
- Technische Informationen Simmons Citrat-Agar. In: Website Merck Millipore. Abgerufen am 1. Januar 2020.
- Betty A. Forbes, Daniel F. Sahm, Alice S. Weissfeld: BAILEY & SCOTT'S Diagnostic Microbiology, 10. Auflage, Don Ladig 1998, , ISBN 0-8151-2535-6.
- Hans G. Schlegel, Christiane Zaborosch: Allgemeine Mikrobiologie. 7. Auflage. Thieme Verlag, Stuttgart/New York 1992, ISBN 3-13-444607-3.
- B. W. Senior: Media and tests to simplify the recognition and identification of members of the Proteeae. In: Journal of Medical Microbiology. Band 46, Nr. 1, Januar 1997, S. 39–44, doi:10.1099/00222615-46-1-39, PMID 9003744.
- Annette Rüschendorf: Medizinische Mykologie. Bestimmung und Differenzierung von Sprosspilzen, Schimmelpilzen, Dermatophyten und dimorphen Pilzen. 2014, 3. Auflage, ISBN 978-3-86541-629-2
- Patrick R. Murray: Manual of clinical microbiology. ASM Press, 1995, 6th ed., ISBN 1-55581-086-1.
- Bacteria Identification (offline) für Android
