Virus-Taxonomie
Als Virus-Taxonomie bezeichnet man die international verbindliche Benennung von Viren, Virusfamilien und -gattungen. Die Entscheidung über verschiedene Taxa wird von einem internationalen Gremium, dem International Committee on Taxonomy of Viruses (ICTV), beraten und getroffen.
Die Virus-Taxonomie ist ein an die Taxonomien von Pflanzen und Tieren angelehnter Versuch, die Vielfalt viraler und subviraler Entitäten (auch Prionen und Retrotransposons) systematisch und einheitlich zu ordnen. Sie darf nicht mit der weiter gefassten Virusklassifikation verwechselt werden, bei der verschiedene Eigenschaften von Viren zur Einteilung herangezogen werden können (zum Beispiel nur Genomstruktur, Baltimore-Klassifikation, Wirtsspecies, Übertragungsart etc.).
Historie
Vorgänger der heutigen ICTV-Taxonomie ist die von André Lwoff, Robert W. Horne und Paul Tournier im Jahre 1962 entsprechend der von Carl von Linné begründeten binären Klassifikation der Lebewesen begründete System (nach diesen Autoren LHT-System genannt). In ihr werden analog zur Taxonomie anderer Lebewesen, die folgenden Taxa unterteilt:
- Virosphäre (Phylum: Vira)
Die entscheidenden Charakteristika für diese Klassifikation waren:
- die Natur des viralen Genoms (DNA oder RNA)
- die Symmetrie des Kapsids
- Vorhandensein einer Lipidumhüllung
- Größe von Virion und Kapsid
Auf Grundlage des Wissens um die Molekularbiologie der Viren hatte sich ab 1971 eine weitere Klassifikation etabliert, welche auf einen Vorschlag des Nobelpreisträgers David Baltimore zurückgeht.[1] Wichtige Kriterien dieser Baltimore-Klassifikation sind:
- die Genomstruktur:
- die Form (Symmetrie) des Kapsids
- das Vorhandensein einer Hülle
- die Anordnung der Gene innerhalb des Genoms
- die Replikationsstrategie (abhängig von Genomstruktur)
- die Virusgröße
Virustaxonomie nach ICTV
Das International Committee on Taxonomy of Viruses (ICTV) begann Anfang der 1970er Jahre mit der Ausarbeitung und Umsetzung von Regeln für die Benennung und Klassifizierung von Viren, eine Arbeit, die bis in die Gegenwart andauert. Das ICTV ist das einzige Gremium, das von der International Union of Microbiological Societies mit der Aufgabe betraut wurde, eine universelle Virustaxonomie zu entwickeln, zu verfeinern und aufrechtzuerhalten.[2] Das System hat viele Gemeinsamkeiten mit dem Klassifikationssystem der zellulären Organismen, wie z. B. die Taxonstruktur. Es gibt jedoch einige Unterschiede, wie etwa die fest vorgeschriebenen Namensendungen für taxonomische Namen aller Ränge ab Gattung aufwärts, sowie die durchgängige Verwendung von Kursivschrift für alle taxonomischen Namen[3] – anders als für zelluläre Organismen (nach International Code of Nomenclature für Algen, Pilze und Pflanzen und International Code of Zoological Nomenclature).[4]
Gegenüber den Baltimore-Kriterien zielt dabei der Fokus zunehmend auf Übereinstimmungen in der Nukleotidsequenz (Homologie). Dies erlaubt es im Prinzip auch Metagenomanalysen mit einzubeziehen, so dass man für die Aufstellung einer Taxonomie nicht mehr notwendig auf die Isolation eines Virusstamms angewiesen ist. Möglich machen dies die Fortschritte in Vereinfachung, Zugänglichkeit, Dauer und Kosten der Gensequenzierung. Auch wenn die Baltimore-Kriterien an zweiter Stelle immer noch eine wichtige Rolle spielen, geraten sie immer weiter in den Hintergrund. Spezies, über die das ICTV keine ausreichenden Genom-Informationen bekommt, sind daher taxonomisch nicht einzuordnen und werden vom ICTV ggf. auch wieder ausgelistet. Zudem hat das ICTV immer mehr Taxa höheren Ranges aufgestellt, die aufgrund von Gen-Homologien Vertreter verschiedener Baltimore-Gruppen in sich vereinen (z. B. Pleolipoviridae, Ortervirales und zwei der vier Realm – en. realms), und die daher als Gruppe nicht im Baltimore-System unterzubringen sind. Die Entdeckung der (mit Stand Mitte März 2021) noch vom ICTV bestätigten Familie „Cruciviridae“, deren Vertreter mit einem chimär DNA/RNA-Genom nach Baltimore nicht klassifizierbar sind, tut ein Übriges.
Für die Taxonomie unberücksichtigt, aber dennoch für die Zusammenfassung unterschiedlicher Viren mit gemeinsamen medizinischen oder epidemiologischen Merkmalen wichtige Kriterien sind:
- gemeinsame Organismen (Wirte), die sie infizieren
- ähnliche Krankheitsbilder bzw. Infektion des gleichen Organs (z. B. Hepatitis-Viren)
- gemeinsame Übertragungswege, z. B. Übertragung durch Vektoren (Gliederfüßer: Arboviren)
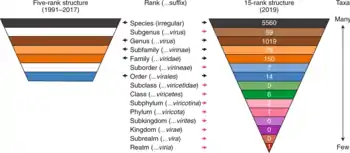
Die taxonomische Struktur war bis 2017 im Prinzip wie bei der herkömmlichen Virusklassifikation ab Stufe Ordnung und darunter (siehe oben) und wurde 2018 durch weitere Stufen wie folgt ergänzt (mit vom LHC-System abweichenden Namensendungen). Oberste Rangstufe ist jetzt Realm[5] (en. realm, anstelle der Domäne bei zellulären Organismen):[6][7]
- Realm (en. realm) (…viria)
Es gibt in diesen Richtlinien (noch) keine Definition von Tribus, Unterarten (Subspezies), Stämmen (analog zu Bakterienstamm, en. strain) oder Isolaten. Die Namen einzelner Viren (im Sinn von Isolaten oder Stämmen) werden – genauso wie reine Akronyme normal (nicht kursiv) geschrieben.[3]
Anzahl der Taxa
Unter den Viren gibt es zurzeit (Oktober 2020, ratifiziert März 2021) folgende Anzahl von Vertretern der jeweiligen taxonomischen Rangstufen:[8]
| Rang | Anzahl Mitglieder |
|---|---|
| Art | 9110 |
| Untergattung | 70 |
| Gattung | 2224 |
| Unterfamilie | 136 |
| Familie | 189 |
| Unterordnung | 8 |
| Ordnung | 59 |
| Unterklasse | 0 |
| Klasse | 39 |
| Subphylum | 2 |
| Phylum | 17 |
| Unterreich | 0 |
| Reich | 10 |
| Subrealm | 0 |
| Realm | 6 |
Virologen haben bei Indischen Riesenflughunden mittels PCR-Techniken nach bekannten und unbekannten Vertretern von neun verschiedenen Virenfamilien respektive -gattungen gesucht, und aufgrund dieser Resultate abgeschätzt, dass die gesamte Säugetierwelt mindestens 320.000 Virenarten beherbergen muss.[9] In der offiziellen Veröffentlichung des ICTV (Stand März 2020, Master Species List 2018b.v2)[10] finden sich aufgrund des starken Zuwachses an Wissen viele sonst übliche Bezeichnungen nicht, da über deren taxonomische Zuordnung bisher noch keine einheitliche Meinung gefunden werden konnte.
Top-Level Taxa
Realm (en. realms)
- Adnaviria (filamentäre archaeale Viren, Baltimore 1, Klasse Tokiviricetes mit den Ordnungen Ligamenvirales und Primavirales — Familie Tristromaviridae).[11]
- Duplodnaviria (Baltimore 1)
- Monodnaviria (Baltimore 1 und 2)
- Riboviria (Baltimore 3 bis 7)
- Ribozyviria (Gattung Deltavirus und ähnliche, Baltimore 5)
- Varidnaviria (Baltimore 1, ursprünglich vorgeschlagen als „Divdnaviria“, „Vertical jelly roll major capsid protein DNA viruses“, „DJR-MCP-Viren“)[12]
Klasse(n) ohne Zuweisung zu einem höheren Rang
- Naldaviricetes (Arthropoda-spezifische Viren mit großen DNA – Familien Baculoviridae, Hytrosaviridae, Nimaviridae, Nudiviridae)
Familien ohne Zuweisung zu einem höheren Rang
- Alphasatellitidae
- Ampullaviridae
- Anelloviridae
- Avsunviroidae
- Bicaudaviridae
- Clavaviridae
- Finnlakeviridae
- Fuselloviridae
- Globuloviridae
- Guttaviridae
- Halspiviridae
- Ovaliviridae
- Plasmaviridae
- Polydnaviridae
- Portogloboviridae
- Pospiviroidae
- Spiraviridae
- Thaspiviridae
- Tolecusatellitidae
- Tristromaviridae[11]
Gattungen ohne Zuweisung zu einem höheren Rang
DNA-Viren
Viren mit DNA-Genom bilden keine taxonomische Einheit.
Doppelsträngige DNA-Viren (dsDNA: double stranded DNA)
Viren mit Doppelstrang-DNA-Genom werden nach Baltimore in die nicht-taxonomische Gruppe 1 klassifiziert:[13]
- zum Realm Varidnaviria
- Phylum Nucleocytoviricota (NCLDV)
- Ordnung Rowavirales (mit Adenoviren)
- Ordnung Halopanivirales (mit Sphaerolipoviridae)
- Ordnung Vinavirales
- Ordnung Priklausovirales (mit Virophagen der Lavidaviridae)
- Ordnung Kalamavirales
- Ordnung Belfryvirales (mit Turriviridae)[14] (mit Gattung Alphaturrivirus,[15] dazu Spezies Sulfolobus turreted icosahedral virus 1 und 2, STIV1 und STIV2)
- zum Realm Monodnaviria
- Klasse Papovaviricetes (Papillomviren und Polyomaviren)
- zum Realm Duplodnaviria
- Ordnung Caudovirales (Bakteriophagen etc. mit Kopf-Schwanz-Struktur)
- Ordnung Herpesvirales
- in keinen höheren Rang klassifiziert:
- Familie Ampullaviridae
- Familie Baculoviridae
- Familie Bicaudaviridae
- Familie Clavaviridae[16] (mit Gattung Clavavirus[17])
- Familie Fuselloviridae
- Familie Globuloviridae[18] (mit Gattung Globulovirus[19])
- Familie Guttaviridae
- Familie Hytrosaviridae
- Familie Nimaviridae
- Familie Nudiviridae
- Familie Ovaliviridae
- Familie Plasmaviridae
- Familie Polydnaviridae (vermutlich polyphyletisch[20])
- Gattung Bracovirus
- Gattung Ichnovirus (siehe NCLDV, vermutlich eine gemeinsame Klade mit Ascoviridae und Iridoviridae zu Nucleocytoviricota)
- Familie Thaspiviridae
- Familie Tristromaviridae[21] (mit Gattung Alphatristromavirus)
- Familie Halspiviridae
- ohne zugewiesene Familie:
- Gattung Dinodnavirus (siehe NCLDV, möglicherweise nahe zu Asfarviridae und der vorgeschlagenen Familie Orpheoviridae)[22]
- Gattung Rhizidiovirus[23] (mit einziger Spezies Rhizidiomyces virus)
Einzelsträngige DNA-Viren (ssDNA: single stranded DNA)
Viren mit Einzelstrang-DNA-Genom werden nach Baltimore unabhängig von ihrer Polarität in die nicht-taxonomische Gruppe 2 klassifiziert:[24]
- zu Monodnaviria
- Familie Bacilladnaviridae (ssDNA(+/-), ggf. stückweise auch dsDNA)[25]
- Gattung Diatodnavirus (ssDNA(+/-))
- Gattung Kieseladnavirus (ssDNA(+/-))
- Gattung Protobacilladnavirus (ssDNA(+/-), stückweise auch dsDNA)
- Familie Bidnaviridae[26] (bipartit, mit Gattung Bidensovirus)
- Familie Circoviridae (ssDNA(+/-): zwei Haupt-ORFs, mit entgegengesetzter Polarität)
- Familie Geminiviridae (ssDNA(+/-): die einzelnen Gene haben unterschiedliche Polarität)
- Familie Genomoviridae
- Gattung Gemycircularvirus mit Typusspezies Sclerotinia-Gemycircularvirus 1 (dazu Sclerotinia sclerotiorum hypovirulence-associated DNA virus 1,[27] SsHADV-1), ein Mykovirus[28]
- Gattung Gemyduguivirus
- Gattung Gemygorvirus
- Gattung Gemykibivirus
- Gattung Gemykolovirus
- Gattung Gemykrogvirus
- Gattung Gemykroznavirus
- Gattung Gemytondvirus
- Gattung Gemyvongvirus
- Familie Inoviridae (+)ssDNA
- Familie Microviridae (+)ssDNA
- Familie Nanoviridae (+)ssDNA
- Gattung Babuvirus
- Gattung Nanovirus
- Familie Parvoviridae
- Familie Smacoviridae[29]
- Gattung Bovismacovirus
- Gattung Cosmacovirus
- Gattung Dragsmacovirus
- Gattung Drosmacovirus
- Gattung Huchismacovirus
- Gattung Porprismacovirus
- ohne zugewiesenen höheren Rang:
- Familie Anelloviridae (alle ssDNA(-), d. h. mit negativer Polarität, en: negative single-stranded DNA)
- Familie Spiraviridae ssDNA(+)
Spezielle einzel- und/oder doppelsträngige DNA-Viren
Die Familie Pleolipoviridae war (mit der Ordnung Ortervirales) eines der ersten vom ICTV anerkannten Taxa (Verwandtschaftsgruppe, ermittelt nach der Genom-Sequenz), dessen Mitglieder in verschiedenen Baltimore-Gruppen (nämlich 1 und 2) fallen.
- zum Realm Monodnaviria
Revers transkribierende DNA- und RNA-Viren (ssRNA-RT und dsDNA-RT)
Dazu gehören Viren mit positiver Einzelstrang-RNA, die per Reverse Transkriptase (RT) in DNA zurückgeschrieben und ins Zellgenom eingebaut wird (Retroviren ssRNA-RT, Baltimore-Gruppe 6), sowie Viren mit Doppelstrang-DNA, die umgekehrt zur Replikation einen RNA-Zwischenschritt benutzen und daher ebenfalls revers transkribieren (Pararetroviren dsDNA-RT, Baltimore-Gruppe 7). Die meisten dieser Vertreter gehören unabhängig davon der Ordnung Ortervirales an.[33]
- zum Realm Riboviria
- zur Ordnung Ortervirales (bis auf Caulimoviridae revers transkribierende RNA-Viren ssRNA-RT)
- Familie Belpaoviridae (ssRNA-RT)
- Familie Metaviridae (ssRNA-RT)
- Familie Pseudoviridae (ssRNA-RT)
- Familie Retroviridae (ssRNA-RT, mit Gattung Lentivirus und darin den Spezies HIV-1 und -2, SIV, BIV, FIV)
- Familie Caulimoviridae (dsDNA-RT: Revers transkribierende DNA-Viren)
- Ordnung Blubervirales (dsDNA-RT: Revers transkribierende DNA-Viren, mit Hepadnaviridae)
RNA-Viren
Viren mit RNA-Genom wurden 2019 in ihrer Gesamtheit zunächst in eine Verwandtschaftsgruppe (Taxon) gestellt (Realm Riboviria).[34] dieser Realm umfasst die (nicht-taxonomischen) Baltimore-Gruppen 3, 4 und 5. Im Jahr 2020 wurden jedoch einige Gruppen wieder ausgegliedert bzw. es wurden neue Taxa von RNA-Viren außerhalb dieses Realm anerkannt, so dass aktuell auch die RNA-Viren keine taxomonische Gruppe mehr bilden.[10]
Doppelsträngige RNA-Viren (dsRNA, double stranded RNA)
Viren mit Doppelstrang-RNA-Genom werden nach Baltimore in die nicht-taxonomische Gruppe 3 klassifiziert:[35]
- Familie Birnaviridae
- Genus Aquabirnavirus
- Genus Avibirnavirus
- Genus Blosnavirus
- Genus Entomobirnavirus
- Familie Chrysoviridae
- Genus Alphachrysovirus,
- Genus Betachrysovirus,[36] mit Species Colletotrichum fructicola chrysovirus 1
- Familie Cystoviridae
- Genus Cystovirus
- Familie Megabirnaviridae
- Genus Megabirnavirus
- Familie Partitiviridae
- Genus Alphapartitivirus
- Genus Betapartitivirus
- Genus Gammapartitivirus
- Genus Deltapartitivirus
- Genus Cryspovirus
- Familie Picobirnaviridae
- Genus Picobirnavirus
- Familie Quadriviridae
- Genus Quadrivirus
- Familie Reoviridae
- Unterfamilie Sedoreovirinae
- Genus Cardoreovirus
- Genus Mimoreovirus
- Genus Orbivirus, mit Species Afrikanisches Pferdepestvirus, Blauzungenvirus, Epizootische-Hämorrhagie-Virus
- Genus Phytoreovirus
- Genus Rotavirus
- Genus Seadornavirus
- Unterfamilie Spinareovirinae
- Genus Aquareovirus
- Genus Coltivirus
- Genus Cypovirus
- Genus Dinovernavirus
- Genus Fijivirus
- Genus Idnoreovirus
- Genus Mycoreovirus
- Genus Orthoreovirus
- Genus Oryzavirus
- Familie Quadriviridae
- Genus Quadrivirus
- Familie Totiviridae
- Genus Giardiavirus
- Genus Leishmaniavirus
- Genus Totivirus, mit Species Ustilago maydis virus H1
- Genus Trichomonasvirus, mit Species Trichomonas vaginalis virus 1
- Genus Victorivirus
- Familie nicht bestimmt
- Genus Botybirnavirus, mit Species Botrytis porri botybirnavirus 1
Einzelstrang-RNA-Viren mit negativer Polarität (ss(−)RNA: negative single-stranded RNA)
Viren mit Einzelstrang-RNA-Genom negativer Polarität werden nach Baltimore in die nicht-taxonomische Gruppe 4 klassifiziert:[40]
- Phylum (Stamm): Negarnaviricota, Subphylum (Unterstamm): Haploviricotina, Klasse: Chunqiuviricetes
- Phylum (Stamm): Negarnaviricota, Subphylum (Unterstamm): Haploviricotina, Klasse: Milneviricetes
- Ordnung Serpentovirales
- Familie Aspiviridae (veraltet Ophioviridae)
- Genus Ophiovirus
- Phylum (Stamm): Negarnaviricota, Subphylum (Unterstamm): Haploviricotina, Klasse: Monjiviricetes
- Ordnung Jingchuvirales
- Familie Chuviridae
- Genus Mivirus, mit Species Crustacean Mivirus
- Ordnung Mononegavirales (nicht segmentierte negativsträngige RNA-Viren)[41]
- Familie Artoviridae
- Genus Peropuvirus (früher zu Nyamiviridae), mit Species Pillworm peropuvirus
- Familie Bornaviridae
- Genus Carbovirus, mit Species Southwest carbovirus (Southwest carpet python virus, SWCPV) und Typusspecies Queensland Carbovirus (Jungle carpet python virus, JCPV)
- Genus Cultervirus, mit Typusspecies Sharpbelly cultervirus
- Genus Orthobornavirus – en. Borna Disease Virus, das Virus der Borna-Krankheit, mit Species Mammalian 1 orthobornavirus (Typus) u. a.
- Familie Filoviridae
- Genus Cuevavirus, mit Typusspecies Lloviu cuevavirus
- Genus Ebolavirus, mit Species Tai Forest ebolavirus und Typusspecies Zaire ebolavirus
- Genus Marburgvirus, mit Typusspecies Marburg marburgvirus
- Familie Lispiviridae
- Genus Arlivirus (inklusive der früheren Gattungen Wastrivirus und Chengivirus/Chengtivirus, mit Species Wuchang arlivirus (Chengtivirus, Tick virus 6, TcTV-6), Tacheng arlivirus (Tacheng chengtivirus 6, TcTV-6) sowie Typusspecies Lishi arlivirus (Lishi spider virus 2, LsSV-2)[42]
- Familie Mymonaviridae
- Genus Sclerotimonavirus mit Typusspecies Sclerotinia sclerotimonavirus (Sclerotinia sclerotiorum negative-stranded RNA virus 1, SsNSRV-1)
- Familie Nyamiviridae
- Genus Berhavirus, mit Species Echinoderm berhavirus
- Genus Crustavirus (ehemals Crabvirus), mit Species Wenzhou crustavirus (WzCV-1)
- Genus Nyavirus, mit Species Sierra nevada nyavirus
- Genus Orinovirus
- Genus Socyvirus
- Genus Tapwovirus
- Familie Paramyxoviridae
- Unterfamilie Avulavirinae
- Genus Metaavulavirus
- Genus Orthoavulavirus (ehem. Avulavirus), mit Species Humanes Parainfluenzavirus, sowie Newcastle-Disease-Virus
- Genus Paraavulavirus
- Unterfamilie Metaparamyxovirinae
- Genus Synodonvirus
- Unterfamilie Orthoparamyxovirinae
- Genus Aquaparamyxovirus
- Genus Ferlavirus
- Genus Henipavirus, mit Species Nipah-Virus, sowie Hendravirus
- Genus Jeilongvirus
- Genus Morbillivirus, mit Species Masernvirus
- Genus Narmovirus
- Genus Respirovirus
- Genus Salemvirus
- Unterfamilie Rubulavirinae
- Genus Orthorubulavirus, (ehem. Rubulavirus), mit Species Mumpsvirus
- Genus Pararubulavirus
- Familie Pneumoviridae
- Genus Metapneumovirus, mit Species Humanes Metapneumovirus – en. Human metapneumovirus (HMPV), sowie Avianes Metapneumovirus – en. Avian metapneumovirus (AMPV)
- Genus Orthopneumovirus, mit Species Humanes Respiratorisches Synzytial-Virus – en. Human orthopneumovirus (HRSV), Bovines Respiratorisches Syncytialvirus – en. Bovine orthopneumovirus(BRSV), sowie Murines Pneumonievirus – en. Murine orthopneumovirus
- Familie Rhabdoviridae
- Genus Almendravirus
- Genus Alphanemrhavirus
- Genus Caligrhavirus
- Genus Curiovirus
- Genus Cytorhabdovirus
- Genus Dichorhavirus
- Genus Ephemerovirus
- Genus Hapavirus
- Genus Ledantevirus
- Genus Lyssavirus, mit Species Tollwutvirus
- Genus Novirhabdovirus
- Genus Nucleorhabdovirus
- Genus Perhabdovirus
- Genus Sigmavirus
- Genus Sprivivirus
- Genus Sripuvirus
- Genus Tibrovirus
- Genus Tupavirus
- Genus Varicosavirus
- Genus Vesiculovirus
- Familie Sunviridae
- Genus Sunshinevirus (SunCV)
- Familie Xinmoviridae
- Genus Anphevirus (XcMV)
- keinem Genus zugeordnet
- Species ‚Taastrup virus‘ (TV)[43]
- Phylum (Stamm): Negarnaviricota, Subphylum (Unterstamm): Haploviricotina, Klasse: Yunchangviricetes
- Ordnung Goujianvirales
- Familie Yueviridae
- Genus Yuyuevirus
- Phylum (Stamm): Negarnaviricota, Subphylum (Unterstamm): Haploviricotina, Klasse: Ellioviricetes
- Ordnung Bunyavirales
- Familie Arenaviridae
- Genus Hartmanivirus, mit Species Haartman-Institut-Schlangen-Virus
- Genus Mammarenavirus (früher Arenavirus), mit Species Lassa-Virus
- Genus Reptarenavirus, mit Species Universität-Gießen-Virus
- Familie Cruliviridae
- Genus Lincruvirus
- Familie Fimoviridae
- Genus Emaravirus, mit Species European mountain ash ringspot-associated virus[44]
- Familie Hantaviridae
- Genus Loanvirus
- Genus Mobatvirus
- Genus Orthohantavirus, mit Species Sin-Nombre-Virus
- Genus Thottimvirus
- Familie Mypoviridae
- Genus Hubavirus
- Familie Nairoviridae
- Genus Orthonairovirus
- Genus Shaspivirus
- Genus Striwavirus
- Familie Peribunyaviridae
- Genus Herbevirus
- Genus Orthobunyavirus, mit Species Akabane-Virus, sowie Tete-Virus
- Genus Shangavirus
- Genus Tospovirus (früher als Orthotospovirus zu Tospoviridae), mit Species Tomatenbronzefleckenvirus – en. Tomato spotted wilt tospovirus
- Familie Phasmaviridae
- Genus Orthophasmavirus
- Genus Feravirus (früher zu Feraviridae)
- Genus Inshuvirus
- Genus Jonvirus (früher als Orthojonvirus zu Jonviridae)
- Genus Sawastrivirus (früher Wastrivirus), mit Typusspecies Sanxia sawastrivirus (Sanxia wastrivirus Water strider virus 4, SxWSV-4)[42]
- Genus Wuhivirus
- Familie Phenuiviridae
- Genus Bandavirus (früher Banyangvirus), mit Species Dabie bandavirus – syn. SFTS-Virus
- Genus Beidivirus
- Genus Goukovirus
- Genus Horwuvirus
- Genus Hudivirus
- Genus Mobuvirus
- Genus Phasivirus
- Genus Phlebovirus
- Genus Pidchovirus
- Genus Tenuivirus
- Genus Wubeivirus
- Familie Wupedeviridae
- Genus Wumivirus
- Phylum (Stamm): Negarnaviricota, Subphylum (Unterstamm): Polyploviricotina, Klasse: Insthoviricetes
- Ordnung Articulavirales
- Familie Amnoonviridae
- Genus Tilapinevirus
- Familie Orthomyxoviridae
- Genus Alphainfluenzavirus, mit einziger Species: Influenzavirus A
- Genus Betainfluenzavirus, mit einziger Species: Influenzavirus B
- Genus Gammainfluenzavirus, mit einziger Species: Influenzavirus C
- Genus Deltainfluenzavirus, mit einziger Species: Influenzavirus D
- Genus Isavirus
- Genus Quaranjavirus
- Genus Thogotovirus
- Phylum (Stamm): nicht bestimmt, Subphylum (Unterstamm): nicht bestimmt, Klasse: nicht bestimmt
- Ordnung nicht bestimmt
- Familie nicht bestimmt
- Genus Deltavirus, mit einziger Species Hepatitis-D-Virus (HDV)
Einzelstrang-RNA-Viren mit positiver Polarität (ss(+)RNA: positive single stranded RNA)
Viren mit Einzelstrang-RNA-Genom positiver Polarität werden nach Baltimore in die nicht-taxonomische Gruppe 5 klassifiziert:[45]
- Ordnung Nidovirales
- Unterordnung Abnidovirineae
- Familie Abyssoviridae
- Unterfamilie Tiamatvirinae
- Genus Alphaabyssovirus – Subgenus: Aplyccavirus
- Familie Arteriviridae
- Unterfamilie Crocarterivirinae
- Genus Muarterivirus
- Unterfamilie Equarterivirinae
- Genus Alphaarterivirus (früher Equartevirus)
- Unterfamilie Heroarterivirinae
- Genus Lambdaarterivirus
- Unterfamilie Simarterivirinae
- Genus Deltaarterivirus – Subgenera: Hedartevirus, Pedartevirus
- Genus Epsilonarterivirus – Subgenus: Sheartevirus
- Genus Etaarterivirus
- Genus Iotaarterivirus – Subgenera: Debiartevirus, Kigiartevirus
- Genus Thetaarterivirus – Subgenera: Kaftartevirus, Mitartevirus
- Genus Zetaarterivirus
- Unterfamilie Variarterivirinae
- Genus Betaarterivirus – Subgenera: Ampobartevirus, Chibartevirus, Eurpobartevirus
- Genus Gammaarterivirus
- Unterfamilie Zealarterivirinae
- Genus Kappaarterivirus
- Aufgrund der Reorganisation dieser Familie gibt es die bisherigen Gattungen Dipartevirus, Nesartevirus, Porartevirus und Simartevirus nicht mehr.
- Unterordnung Cornidovirineae
- Familie Coronaviridae
- Unterfamilie Letovirinae
- Genus Alphaletovirus – Subgenus: Milecovirus
- Unterfamilie Orthocoronavirinae
- Genus Alphacoronavirus – Subgenera: Colacovirus, Decacovirus, Duvinacovirus, Luchacovirus, Minacovirus, Minunacovirus, Myotacovirus, Nyctacovirus, Pedacovirus, Rhinacovirus, Setracovirus, Tegacovirus
- Genus Betacoronavirus – Subgenera: Embecovirus, Hibecovirus, Merbecovirus (mit MERS-CoV), Nobecovirus, Sarbecovirus (mit SARS-CoV und SARS-CoV-2)
- Genus Gammacoronavirus – Subgenera: Cegacovirus, Igacovirus
- Genus Deltacoronavirus – Subgenera: Andecovirus, Buldecovirus, Herdecovirus, Moordecovirus
- Unterordnung Mesnidovirineae
- Familie Medioniviridae
- Unterfamilie Medionivirinae
- Genus Turrinivirus – Subgenus: Beturrivirus
- Unterfamilie Tunicanivirinae
- Genus Bolenivirus – Subgenus: Balbicanovirus
- Familie Mesoniviridae
- Unterfamilie Hexponivirinae
- Genus Alphamesonivirus – Subgenera: Casualivirus, Enselivirus, Hanalivirus, Kadilivirus, Karsalivirus, Menolivirus, Namcalivirus, Ofalivirus
- Unterordnung Monidovirineae
- Familie Mononiviridae
- Unterfamilie Mononivirinae
- Genus Alphamononivirus – Subgenus: Dumedivirus
- Unterordnung Ronidovirineae
- Familie Euroniviridae
- Unterfamilie Ceronivirinae
- Genus Charybnivirus – Subgenera: Cradenivirus, Wenilivirus
- Unterfamilie Crustonivirinae
- Genus Paguronivirus – Subgenus: Behecravirus
- Familie Roniviridae
- Unterfamilie Okanivirinae
- Genus Okavirus – Subgenus: Tipravirus
- Unterordnung Tornidovirineae
- Familie Tobaniviridae
- Unterfamilie Piscanivirinae
- Genus Bafinivirus (früher zu Coronaviridae:Torovirinae) – Subgenera: Blicbavirus, Pimfabavirus
- Genus Oncotshavirus – Subgenus: Salnivirus
- Unterfamilie Remotovirinae
- Genus Bostovirus – Subgenus: Bosnitovirus
- Unterfamilie Serpentovirinae
- Genus Infratovirus – Subgenus: Xintolivirus
- Genus Pregotovirus – Subgenus: Roypretovirus
- Genus Sectovirus – Subgenus: Sanematovirus
- Genus Tiruvirus – Subgenus: Tilitovirus
- Unterfamilie Torovirinae (früher Fam. Coronaviridae)
- Genus Torovirus – Subgenus: Renitovirus
- Ordnung Picornavirales
- Familie Dicistroviridae
- Genus Aparavirus, mit Species Akutes Bienenparalyse-Virus – en. Acute bee paralysis virus (ABPV)
- Genus Cripavirus
- Genus Triatovirus
- Familie Iflaviridae
- Genus Iflavirus, mit Species Langsames Bienenparalyse Virus – en. Slow bee paralysis virus (SBPV), sowie Flügeldeformationsvirus – en. Deformed wing virus (DWV)
- Familie Marnaviridae
- Genus Bacillarnavirus
- Genus Kusarnavirus
- Genus Labyrnavirus
- Genus Locarnavirus
- Genus Marnavirus
- Genus Salisharnavirus
- Genus Sogarnavirus
- Familie Picornaviridae
- Genus Aalivirus
- Genus Ampivirus
- Genus Aphthovirus
- Genus Aquamavirus
- Genus Avihepatovirus
- Genus Avisivirus
- Genus Bopivirus
- Genus Cardiovirus
- Genus Cosavirus
- Genus Crohivirus
- Genus Dicipivirus
- Genus Enterovirus, mit Species Coxsackie-Virus, Poliovirus – en. Enterovirus C, sowie den Rhinoviren – en. Rhinovirus A, B, C,…
- Genus Erbovirus
- Genus Gallivirus
- Genus Harkavirus
- Genus Hepatovirus, mit Species Hepatitis-A-Virus (HAV)
- Genus Hunnivirus
- Genus Kobuvirus
- Genus Kunsagivirus
- Genus Limnipivirus
- Genus Megrivirus
- Genus Mischivirus
- Genus Mosavirus
- Genus Orivirus
- Genus Oscivirus
- Genus Parechovirus
- Genus Pasivirus
- Genus Passerivirus
- Genus Potamipivirus
- Genus Rabovirus
- Genus Rosavirus
- Genus Sakobuvirus
- Genus Salivirus
- Genus Sapelovirus
- Genus Senecavirus
- Genus Shanbavirus
- Genus Sicinivirus
- Genus Teschovirus
- Genus Torchivirus
- Genus Tremovirus
- Familie Polycipiviridae
- Genus Chipolycivirus
- Genus Hupolycivirus
- Genus Sopolycivirus
- Familie Secoviridae
- Unterfamilie Comovirinae
- Genus Comovirus
- Genus Fabavirus
- Genus Nepovirus, mit Species Chicory yellow mottle virus, Grapevine bulgarian latent virus, Grapevine fanleaf virus, Myrobalan latent ringspot virus, Tomatenschwarzringvirus – en. Tomato black ring virus,; Arabis-Mosaic-Virus – en. Arabis mosaic virus, sowie Beet ringspot virus
- Unterfamilie nicht bestiimt
- Genus Cheravirus
- Genus Sadwavirus
- Genus Sequivirus
- Genus Torradovirus
- Genus Waikavirus
- Ordnung Tymovirales
- Familie Alphaflexiviridae
- Genus Allexivirus
- Genus Botrexvirus
- Genus Lolavirus, mit Species Lolium-Latenz-Virus – en. Lolium latenz virus (LoLV)
- Genus Mandarivirus
- Genus Platypuvirus
- Genus Potexvirus
- Genus Sclerodarnavirus
- Familie Betaflexiviridae
- Unterfamilie Quinvirinae
- Genus Carlavirus
- Genus Foveavirus
- Genus Robigovirus
- Unterfamilie Trivirinae
- Genus Capillovirus
- Genus Chordovirus
- Genus Citrivirus
- Genus Divavirus
- Genus Prunevirus
- Genus Tepovirus
- Genus Trichovirus
- Genus Vitivirus
- Familie Gammaflexiviridae
- Genus Mycoflexivirus
- Familie Deltaflexiviridae
- Genus Deltaflexivirus
- Familie Tymoviridae
- Genus Maculavirus
- Genus Marafivirus
- Genus Tymovirus, mit Species Wild cucumber mosaic virus, sowie Gelber Rüben-Mosaikvirus – en. Turnip yellow mosaic virus (TYMV)
- Ordnung nicht bestimmt:
- Familie Alphatetraviridae
- Genus Betatetravirus
- Genus Omegatetravirus
- Familie Alvernaviridae
- Genus Dinornavirus
- Familie Alvernaviridae
- Genus Amalgavirus
- Genus Zybavirus[46]
- Familie Astroviridae
- Genus Avastrovirus
- Genus Mamastrovirus
- Genus ‚Bastrovirus‘[47]
- Familie Barnaviridae
- Genus Barnavirus
- Familie Benyviridae
- Genus Benyvirus, mit Species Beet necrotic yellow vein virus (satelliten-ähnliche RNA)
- Familie Botourmiaviridae
- Genus Botoulivirus
- Genus Magoulivirus
- Genus Ourmiavirus
- Genus Scleroulivirus
- Familie Bromoviridae
- Genus Alfamovirus
- Genus Anulavirus
- Genus Bromovirus (früher Tricornavirus)
- Genus Cucumovirus, mit Species Peanut stunt virus, sowie Gurkenmosaikvirus – en. Cucumber mosaic virus
- Genus Ilarovirus
- Genus Oleavirus
- Familie Caliciviridae
- Genus Lagovirus
- Genus Nebovirus
- Genus Norovirus, mit Species Norwalk-Virus
- Genus Sapovirus
- Genus Vesivirus
- Familie Carmotetraviridae
- Genus Alphacarmotetravirus
- Familie Closteroviridae
- Genus Ampelovirus
- Genus Closterovirus
- Genus Crinivirus
- Genus Velarivirus
- Familie Endornaviridae (früher als dsRNA klassifiziert)
- Genus Alphaendornavirus
- Genus Betaendornavirus
- Familie Flaviviridae
- Genus Flavivirus, mit Species West-Nil-Virus – en. West nile virus (WNV), Gadgets-Gully-Virus – en. Gadgets Gully virus (GGYV), FSME-Virus – en. Tick-borne encephalitis virus (TBEV), Dengue-Virus – en. Dengue virus (DENV), St.-Louis-Enzephalitis-Virus – en. Saint Louis encephalitis virus (SLEV), Usutu-Virus – en. Usutu virus (USUV), Zika-Virus – en. Zika virus (ZIKV), sowie Gelbfieber-Virus – en. Yellow fever virus (YFV)
- Genus Hepacivirus, mit Species Hepatitis-C-Virus
- Genus Pegivirus
- Genus Pestivirus, mit Species Pestivirus C – syn. Klassisches Schweinepest-Virus – en. Classical swine fever virus (CSFV) oder Hog cholera virus
- Familie Hepeviridae
- Genus Orthohepevirus
- Genus Piscihepevirus
- Familie Hypoviridae
- Genus Hypovirus
- Familie Kitaviridae[51]
- Genus Blunervirus, mit Species Blueberry necrotic ring blotch virus (BNRBV)
- Genus Cilevirus (CiLV)
- Genus Higrevirus, mit Species Hibiscus green spot virus 2 (HGSV-2)
- Familie Leviviridae
- Genus Allolevivirus, mit Species Escherichia virus Qbeta
- Genus Levivirus, mit Species Escherichia virus MS2
- Familie Luteoviridae
- Genus Enamovirus
- Genus Luteovirus, mit den Barley-Yellow-Dwarf-Viren – en. Barley yellow dwarf virus MAV (BYDV-MAV), Barley yellow dwarf virus PAV (BYDV-PAV), sowie Barley yellow dwarf virus PAS (BYDV-PAS)
- Genus Polerovirus, mit Species Cereal yellow dwarf virus RPV (CYDV-RPV), sowie Cereal yellow dwarf virus RPS (CYDV-RPS)
- Familie Matonaviridae
- Genus Rubivirus (früher zu Togaviridae), mit einziger Species Rötelnvirus, en. Rubella virus (RUBV)
- Familie Narnaviridae
- Genus Mitovirus
- Genus Narnavirus
- Familie Nodaviridae
- Genus Alphanodavirus
- Genus Betanodavirus
- Familie Permutotetraviridae
- Genus Alphapermutotetravirus
- Familie Potyviridae
- Genus Bevemovirus
- Genus Brambyvirus
- Genus Bymovirus
- Genus Ipomovirus
- Genus Macluravirus
- Genus Poacevirus
- Genus Potyvirus, mit Species Meerrettich-Mosaikvirus – en. Turnip mosaic virus, Hirsemosaikvirus – en. Sorghum mosaic virus (SrMV), Lily-Mottle-Virus – en. Lily mottle virus (LMoV), sowie Sellerie-Virus Y – en. Apium virus Y (ApVY)
- Genus Roymovirus
- Genus Rymovirus
- Genus Tritimovirus
- Familie Sarthroviridae[52][53] (ssRNA(+), laut Vorschlag sind dies Satelliten-Viren)
- Genus Macronovirus,[54] mit einziger Species Macrobrachium satellite virus 1
- Familie Solemoviridae
- Genus Polemovirus
- Genus Sobemovirus, mit Species Cymbidium ringspot virus
- Familie Solinviviridae
- Genus Invictavirus, mit Species Solenopsis invicta virus 3
- Genus Nyfulvavirus, mit Species Solenopsis invicta virus 3
- Familie Togaviridae
- Genus Alphavirus, mit Species Chikungunya-Virus (CHIKV), Mayaro-Virus (MAYV), Ross-River-Virus (RRV), sowie Sindbis-Virus (SINV)
- Familie Tombusviridae
- Unterfamilie Calvusvirinae
- Genus Umbravirus, mit Species Erdnuss-Rosetten-Virus – en. Groundnut rosette virus
- Unterfamilie Procedovirinae
- Genus Alphacarmovirus
- Genus Betacarmovirus, mit Species Turnip crinkle virus
- Genus Gammacarmovirus
- Genus Alphanecrovirus, mit Species Tabak-Nekrose-Virus A und D – en. Tobacco necrosis virus A und D
- Genus Aureusvirus, mit Species Maize white line mosaic virus
- Genus Betanecrovirus
- Genus Gallantivirus
- Genus Macanavirus
- Genus Machlomovirus
- Genus Panicovirus, mit Species Hirse-Mosaik-Virus – en. Panicum mosaic virus
- Genus Pelarspovirus
- Genus Tombusvirus
- Genus Zeavirus
- Unterfamilie Regressovirinae
- Genus Dianthovirus
- Unterfamilie nicht bestimmt:
- Genus Avenavirus
- Familie Virgaviridae[55]
- Genus Furovirus
- Genus Goravirus
- Genus Hordeivirus
- Genus Pecluvirus
- Genus Pomovirus
- Genus Tobamovirus, mit Species Tabakmosaikvirus – en. Tobacco mosaic virus (TMV)
- Genus Tobravirus, mit Species Tabak-Rattle-Virus – en. Tobacco rattle virus (TRV)
- Familie nicht bestimmt:
- Genus Jingmenvirus mit Species Jingmen tick virus (JMTV, ?Fam. Flaviviridae)[56]
- Genus Ideaovirus, mit Species Privet idaeovirus
- Genus Sinaivirus, mit Species Lake Sinai virus 1 und 2[57]
- Genus ‚Negevirus‘, mit Species ‚Blackford virus‘, ‚Bofa virus‘, ‚Buckhurst virus‘, ‚Marsac virus‘, sowie ‚Muthill virus‘[58]
- Genus Albetovirus (Satellit?[59] Siehe unten)
- Genus Aumaivirus (Satellit?[59] Siehe unten)
- Genus Papanivirus (Satellit?[59] Siehe unten)
- Genus Virtovirus (Satellit?[59] Siehe unten)
- Genus nicht bestimmt und nicht in ICTV Master Species List (Ausgabe #34)
- ‚Acyrthosiphon pisum virus‘ (APV)[64]
- ‚Chara australis virus‘ (CAV), alias ‚Chara corallina virus‘[65]
- ‚Extra small virus‘ (XSV)[66][67]
- ‚Nesidiocoris tenuis virus 1‘ (NtV-1)[68]
- ‚Plasmopara halstedii virus‘ (PhV)[69]
- ‚Rosellinia necatrix fusarivirus 1‘ (RnFV1)[70]
- ‚Kelp fly virus‘ (KFV)[71]
- ‚Chronic bee paralysis virus‘ (CBPV)[72]
Subvirale Erreger
Viroide
Die Taxonomie für Viroide nach ICTV folgt denselben Regeln wie für Viren, nur dass der Namensbestandteil „viroid“ enthält. Die Abkürzungen enden auf „Vd“ statt „V“, ggf. gefolgt von einer Nummer (nicht römisch, sondern arabisch).
Satelliten
Das ICTV hat im November 2018 erstmals eine Taxonomie auch für Satelliten-Viren herausgegeben, ebenfalls mit demselben Aufbau, jedoch mit Namensbestandteil „satellit“.
Weitere Informationen befinden sich unter:
- Satellite Nucleic Acids (Satelliten-Nukleinsäuren)
Prionen
- Vertebrate Prions (Wirbeltier-Prionen)
- Fungal Prions (Pilz-Prionen)
Vorgeschlagene oder nicht-offizielle Virustaxa
Die Virus-Taxonomie unterliegt ständigen Veränderungen, die einerseits durch neu entdeckte Viren (speziell in marinen und extremen Lebensräumen) bedingt sind, andererseits dadurch, dass aufgrund immer umfangreicherer Sequenzdaten neue verwandtschaftliche Beziehungen erschlossen werden. In der Regel werden Vorschläge für neue Virustaxa von internationalen Arbeitsgruppen dem ICTV unterbreitet, die allerdings schon vor ihrer offiziellen Übernahme in Publikationen oder Datenbanken (auch denen des NCBI) verwendet werden. Meist sind dies serologisch begründete Serogruppen innerhalb von Gattungen und Familien, aber auch neu geschaffene Virusordnungen, -familien (z. B. „Pithoviridae“) oder -unterfamilien (z. B. „Klosneuvirinae“).
Literatur
- C. M. Fauquet, M. A. Mayo et al.: Eighth Report of the International Committee on Taxonomy of Viruses. London, San Diego, 2012, ISBN 0-12-249951-4
- A. M. Q. King, M. J. Adams, E. B. Carstens, E. J. Lefkowitz (Hrsg.): Virus Taxonomy. Ninth Report of the International Committee on Taxonomy of Viruses. Amsterdam 2005, ISBN 978-0-12-384684-6
- Eugene V. Koonin, Tatiana G. Senkevich, Valerian V. Dolja: The ancient Virus World and evolution of cells, in: Biol Direct 1:29, 19. September 2006, doi:10.1186/1745-6150-1-29, PMID 16984643, PMC 1594570 (freier Volltext)
Weblinks
Einzelnachweise und Anmerkungen
- Lars Gerdes, Ulrich Busch, Sven Pecoraro: Parallele Quantifizierung von gentechnisch veränderten Organismen (GVO), in: Schriftenreihe Gentechnik für Umwelt und Verbraucherschutz, Band 8, 5. Fachtagung "Gentechnik für Umwelt- und Verbraucherschutz", Oberschleißheim, November 2013: Conference Paper, hier: Abb. 6.1: Baltimore-Klassifikation und Virusfamilien
- E. J. Lefkowitz, D. M. Dempsey, R. C. Hendrickson, R. J. Orton, S. G. Siddell, D. B. Smith: Virus taxonomy: the database of the International Committee on Taxonomy of Viruses (ICTV). In: Nucleic Acids Research. 46, Nr. D1, Januar 2018, S. D708–D717. doi:10.1093/nar/gkx932. PMID 29040670. PMC 5753373 (freier Volltext).
- ICTV: How to write virus, species, and other taxa names, ICTV online
- ICTV Code. In: talk.ictvonline.org. International Committee on Taxonomy of Viruses.
- Antwort der Bayerischen Staatsministerin für Gesundheit und Pflege zur schriftlichen Anfrage der Abgeordneten Franz Bergmüller et al. München, 16. Oktober 2020
- International Committee on Taxonomy of Viruses Executive Committe, Virus Taxonomy: 2018 Release, How to write virus and species names
- International Committee on Taxonomy of Viruses Executive Committee: The new scope of virus taxonomy: partitioning the virosphere into 15 hierarchical ranks, in: Nature Microbiology Band 5, S. 668–674 vom 27. April 2020, doi:10.1038/s41564-020-0709-x; sowie Nadja Podbregar: Ein Stammbaum für die Virosphäre, auf: scinexx.de vom 29. April 2020. Beide Artikel haben inhaltlich den Stand von Januar 2020, d. h. Einzelheiten der Master Species List (MSL) Nr. 35 des ICTV vom März 2020 sind noch nicht alle berücksichtigt. Für die grundsätzliche Intention des ICTV hat das aber keine Bedeutung, die Entwicklung ist mit der MSL#35 lediglich in der vorgegebenen Richtung schon wieder weitergegangen.
- ICTV: Virus Taxonomy 2020 Release
- Simon J. Anthony et al.: A Strategy To Estimate Unknown Viral Diversity in Mammals. In: mBio. 3. September 2013, doi:10.1128/mBio.00598-13.
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- Aufgrund struktureller Ähnlichkeiten wurde vorgeschlagen, die Ordnung Ligamenvirales und die Familie Tristromaviridae in einer Klasse Tokiviricetes zu vereinen (toki bedeutet "Faden" auf Georgisch und -viricetes ist das offizielle Suffix für Virusklassen). Referenz: F. Wang, D. P. Baquero, Z. Su, T. Osinski, D. Prangishvili, E. H. Egelman, M. Krupovic: Structure of a filamentous virus uncovers familial ties within the archaeal virosphere. In: Virus Evolution. 6, Nr. 1, Januar 2020, S. veaa023. doi:10.1093/ve/veaa023. PMID 32368353. PMC 7189273 (freier Volltext).
- Koonin EV, Dolja VV, Krupovic M, Varsani A, Wolf YI, Yutin N, Zerbini M, Kuhn JH: Create a megataxonomic framework for DNA viruses encoding vertical jelly roll-type major capsid proteins filling all principal taxonomic ranks. ICTV Proposal 2019.003G, April–Juli 2019
- SIB:Double Strand DNA Viruses, auf: ViralZone
- SIB: Turriviridae, auf: ViralZone
- SIB: Alphaturrivirus, auf: ViralZone
- SIB: Clavaviridae, auf: ViralZone
- SIB: Clavavirus, auf: ViralZone
- SIB: Globuloviridae, auf: ViralZone
- SIB: Globulovirus, auf: ViralZone
- Dupuy C, Huguet E, Drezen JM: Unfolding the evolutionary story of polydnaviruses. In: Virus Res. 117, Nr. 1, 2006, S. 81–89. doi:10.1016/j.virusres.2006.01.001. PMID 16460826.
- SIB: Tristromaviridae, auf: ViralZone
- H. Ogata, K. Toyoda, Y. Tomaru, N. Nakayama, Y. Shirai, J. M. Claverie, K. Nagasaki: Remarkable sequence similarity between the dinoflagellate-infecting marine girus and the terrestrial pathogen African swine fever virus. In: Virol J. 6, 2009, S. 178.
- SIB: Rhizidiovirus, auf: ViralZone.
- SIB: Single Strand DNA Viruses, auf: ViralZone.
- SIB: Bacilladnaviridae.
- SIB: Bidnaviridae.
- NCBI: Sclerotinia sclerotiorum hypovirulence associated DNA virus 1 (no rank)
- Hongxiang Zhang, Jiatao Xie, Yanping Fu, Binnian Tian, David B. Collinge, Daohong Jiang, et al.: A 2-kb Mycovirus Converts a Pathogenic Fungus into a Beneficial Endophyte for Brassica Protection and Yield Enhancement. In: Molecular Plant. Band 13, Nr. 10, 5. Oktober 2020, ISSN 1674-2052, S. 1420–1433, doi:10.1016/j.molp.2020.08.016.
Martin Vieweg: Pilz-Virus verwandelt Feind in Freund, auf: wissenschaft.de vom 1. Oktober 2020. - NCBI: Smacoviridae (family)
- Sencilo A, Paulin L, Kellner S, Helm M, Roine E: Related haloarchaeal pleomorphic viruses contain different genome types. In: Nucleic Acids Research. 40, Nr. 12, Juli 2012, S. 5523–34. doi:10.1093/nar/gks215. PMID 22396526. PMC 3384331 (freier Volltext).
- Pietilä MK, Roine E, Sencilo A, Bamford DH, Oksanen HM: Pleolipoviridae, a newly proposed family comprising archaeal pleomorphic viruses with single-stranded or double-stranded DNA genomes. In: Archives of Virology. 161, Nr. 1, Januar 2016, S. 249–56. doi:10.1007/s00705-015-2613-x. PMID 26459284.
- Pietilä MK, Atanasova NS, Manole V, Liljeroos L, Butcher SJ, Oksanen HM, Bamford DH: Virion architecture unifies globally distributed pleolipoviruses infecting halophilic archaea. In: Journal of Virology. 86, Nr. 9, Mai 2012, S. 5067–79. doi:10.1128/JVI.06915-11. PMID 22357279. PMC 3347350 (freier Volltext).
- SIB: Reverse Transcribing Viruses, auf: ViralZone
- ICTV Master Species List 2018b v2 MSL #34v, März 2019
- SIB: Double Strand RNA Viruses, auf: ViralZone
- SIB: Double Stranded RNA Viruses, auf: ViralZone
- Circulifer tenellus virus 1, auf: Virus-Host DB
- Henxia Xia et al.: A dsRNA virus with filamentous viral particled, in: Nature Communicationsvolume 8, Nr. 168 (2017), doi:10.1038/s41467-017-00237-9
- Spissistilus festinus virus 1, auf: Virus-Host DB
- SIB: Negative Strand RNA Viruses, auf: ViralZone
- Gaya K. Amarasinghe et al.: Taxonomy of the order Mononegavirales: update 2018, Archives of Virology, Band 163, Nr. 8, August 2018, S. 2283–2294
- Claudio L. Afonso et al.: Taxonomy of the order Mononegavirales: update 2016, in: Archives of Virology, Volume 161, Nr. 8, 1. August 2016, S. 2351–2360
- J. O. Bool et al.: Identification and partial characterization of Taastrup virus: a newly identified member species of the Mononegavirales, in: Virology. 2004 Feb 5;319(1):49-59, PMID 14967487, doi:10.1016/j.virol.2003.10.017
- ICTV Emaravirus
- SIB: Positive Strand RNA Viruses, auf: ViralZone
- SIB: Double Stranded RNA Viruses, auf: ViralZone
- Karoline dos Anjos, Tatsuya Nagata, Fernando Lucas de Melo: Complete Genome Sequence of a Novel Bastrovirus Isolated from Raw Sewage. In: Genome announcements. Band 5, Nummer 40, Oktober 2017, S. , doi:10.1128/genomeA.01010-17, PMID 28982994, PMC 5629051 (freier Volltext).
- Fusariviridae (FAMILY), auf: UniProt Taxonom
- FUSARIVIRIDAE, auf: PLANOSPHERE
- Fusariviridae, auf: NCBI Genomes
- D. F. Quito-Avila, P. M. Brannen, W. O. Cline, P. F. Harmon, R. R. Martin: Genetic characterization of Blueberry necrotic ring blotch virus, a novel RNA virus with unique genetic features, in: J Gen Virol. 2013 Jun;94(Pt 6):1426-34. doi:10.1099/vir.0.050393-0, PMID 23486668
- ICTV: Sarthroviridae, Virus Taxonomy: 2019 Release EC 51, Berlin, Germany, July 2019 (MSL #35).
- NCBI: Sarthroviridae (family)
- SIB: Macronovirus, auf: ExPASy: ViralZone
- Adams MJ, Antoniw JF, Kreuze J: Virgaviridae: a new Familie of rod-shaped plant viruses. In: Arch Virol. 154, Nr. 12, 2009, S. 1967–72. doi:10.1007/s00705-009-0506-6. PMID 19862474.
- Xin-Cheng Qin et al.: A tick-borne segmented RNA virus contains genome segments derived from unsegmented viral ancestors, in: PNAS 111 (18), 6. Mai 2014, S. 6744-6749, doi:10.1073/pnas.1324194111
- SIB: Double Stranded RNA Viruses, auf: ViralZone
- Claire L. Webster, Ben Longdon, Samuel H. Lewis, Darren J. Obbard: Twenty-Five New Viruses Associated with the Drosophilidae (Diptera), in: Evol Bioinform Online 12(Suppl 2), 2016, S. 13–25, doi:10.4137/EBO.S39454, PMID 27375356, PMC 4915790 (freier Volltext).
- Krupovic M, Kuhn JH, Fischer MG (2016) A classification system for virophages and satellite viruses. Arch Virol 161(1), S. 233-247, doi:10.1007/s00705-015-2622-9
- Carl J. Franz, Guoyan Zhao, Marie-Anne Félix, David Wang: Complete Genome Sequence of Le Blanc Virus, a Third Caenorhabditis Nematode-Infecting Virus, in: Journal of Virology, 2012, doi:10.1128/JVI.02025-12, PMID 23043172, PMC 3486331 (freier Volltext).
- Hongbing Jiang,a Carl J. Franz, Guang Wu, Hilary Renshaw, Guoyan Zhao, Andrew E. Firth, David Wang: Orsay virus utilizes ribosomal frameshifting to express a novel protein that is incorporated into virions, in: Virology 450-451(100), Februar 2014, S. 213–221. doi:10.1016/j.virol.2013.12.016, PMID 24503084, PMC 3969245 (freier Volltext).
- Carl J.Franz, Hilary Renshaw, Lise Frezal, Yanfang Jiang, Marie-Anne Félix, David Wang: Orsay, Santeuil and Le Blanc viruses primarily infect intestinal cells in Caenorhabditis nematodes, in: Virology, Volume 448, 5. Januar 2014, S. 255-264, doi:10.1016/j.virol.2013.09.024
- Orsay virus. Virus-Host DB
- J. F. van den Heuvel, H. R. Hummelen, Martin Verbeek, Annette Dullemans: Characteristics of Acyrthosiphon pisum Virus, a Newly Identified Virus Infecting the Pea Aphid, in: J Invertebr Pathol. November/Dezember 1997;70(3):169-76., doi:10.1006/jipa.1997.4691, PMID 9367722.
- A. J. Gibbs, M. Torronen, A. M. Mackenzie, J. T. Wood 2nd, J. S. Armstrong, H. Kondo, T. Tamada, P. L. Keese: The enigmatic genome of Chara australis virus, in: J Gen Virol. 92(Pt 11), November 2011, S. 2679-2690, doi:10.1099/vir.0.033852-0, PMID 21733884.
- D. Qian et al.: Extra small virus-like particles (XSV) and nodavirus associated with whitish muscle disease in the giant freshwater prawn, Macrobrachium rosenbergii, in: J Fish Dis. 26(9), September 2003, S. 521-527, PMID 14575370, doi:10.1046/j.1365-2761.2003.00486.x
- J. S. Widada, J. R. Bonami: Characteristics of the monocistronic genome of extra small virus, a virus-like particle associated with Macrobrachium rosenbergii nodavirus: possible candidate for a new species of satellite virus, in: J Gen Virol. 85(Pt 3), März 2004, S. 643-646, PMID 14993649, doi:10.1099/vir.0.79777-0
- Nesidiocoris tenuis virus, auf: Virus-Host DB
- Marion Heller-Dohmen et al.: The nucleotide sequence and genome organization of Plasmopara halstedii virus, in: Virol J. 2011; 8: 123, doi:10.1186/1743-422X-8-123, PMC 3069955 (freier Volltext), PMID 21410989
- Rosellinia necatrix fusarivirus 1, auf: Virus-Host DB
- Kelp fly virus, auf: Virus-Host DB
- Eric Dubois et al.: Effect of pollen traps on the relapse of chronic bee paralysis virus in honeybee (Apis mellifera) colonies, in: Apidologie April 2018, Volume 49, Issue 2, S. 235–242, doi:10.1007/s13592-017-0547-x