Anelloviridae
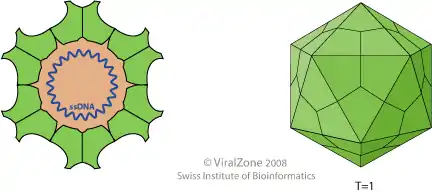
Die Anelloviridae sind eine Familie von DNA-Viren. Ihr Genom besteht aus einer ringförmigen (zirkulären) Einzelstrang-DNA negativer Polarität. Sie haben ein nicht umhülltes Kapsid von abgerundet ikosaedrischer Geometrie mit T=3-Symmetrie. Ihre Wirte sind Wirbeltiere. Es sind etwa 200 verschiedene Arten bekannt.[1][2] Die Typusart ist das 1997 entdeckte Torque-Teno-Virus 1 (TTV-1) aus der Gattung Alphatorquevirus.[3]
| Anelloviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
Anelloviridae | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Anelloviridae | ||||||||||||||
| Links | ||||||||||||||
|
Die Namensbezeichnung ist abgeleitet von italienisch anello ‚Ring‘, was sich auf das zirkuläre Genom der Anelloviridae bezieht.
Genom

.png.webp)
Das Genom ist nicht segmentiert (monopartit), es enthält ein einzelnes Molekül zirkulärer einzelsträngiger DNA mit negativem Polarität. Das gesamte Genom ist 3000–4000 nt lang. Das Genom hat offene Leserahmen (englisch open reading frames, ORFs) und einen hohen Grad an genetischer Diversität.[5][2]
Replikation
Der Replikationsmechanismus der Anelloviridae scheint die Rolling-circle-Replikation zu sein. Für die Replikation ist eine Wirtspolymerase erforderlich. Das Genom des Virus selbst kodiert nämlich nicht für eine virale Polymerase, weshalb sich die Anelloviren im Zellkern replizieren müssen.[2]
Pathologie
Anelloviren sind weit verbreitet und genetisch vielfältig.
Mindestens 200 verschiedene Arten kommen bei Mensch und Tier vor.
Sie verursachen chronische menschliche Virusinfektionen, die bisher noch nicht mit einer Krankheit in Verbindung gebracht wurden.
Drei Gattungen der Anelloviridae sind mit Infektionen des Menschen verbunden:[1]
● das Alphatorquevirus (veraltet Torque-Teno-Virus, TTV)
● das Betatorquevirus (veraltet Torque-Teno-Mini-Virus, TTMV)
● das Gammatorquevirus (veraltet Torque-Teno-Midi-Virus, TTMDV)
Systematik
Die innere Systematik der Anelloviridae ist nach dem International Committee on Taxonomy of Viruses (ICTV) mit Stand März 2019 wie folgt:[6]
Familie Anelloviridae
- Gattung Alphatorquevirus (veraltet Anellovirus, Torque-Teno-Virus, TTV)
- Spezies Torque teno virus 1 (Torque-Teno-Virus 1, TTV-1, Typusspezies)
- Spezies „Simian torque teno virus 30“ Chlorocebus aethiops
- Spezies „Simian torque teno virus 31“
- Spezies „Simian torque teno virus 32“
- Spezies „Simian torque teno virus 33“
- Spezies „Simian torque teno virus 34“
- Gattung Betatorquevirus (veraltet Torque-Teno-Mini-Virus, 13 TTMV-Spezies)
- Spezies Torque teno mini virus 1 (Torque-Teno-Mini-Virus, TTMV-1, Typusspezies)
- Gattung Gammatorquevirus (veraltet Torque-Teno-Midi-Virus, TTMDV)
- Spezies Torque teno midi virus 1 (Torque-Teno-Midi-Virus 1, TTMDV-1, Typusspezies)
- Gattung Deltatorquevirus
- Spezies Tupaia-Torque-Teno-Virus (Torque teno tupaia virus, TTV14-Tbc, Typusspezies)
- Gattung Epsilontorquevirus
- Spezies Torque teno tamarin virus (Typusspezies)
- Gattung Zetatorquevirus
- Spezies Torque teno douroucouli virus (Typusspezies)
- Gattung Etatorquevirus
- Spezies Torque teno felis virus (Typusspezies)
- Gattung Thetatorquevirus
- Spezies Hunde-Torque-Teno-Virus (Torque teno canis virus, TTV10-Cf, Typusspezies)
- Gattung Iotatorquevirus
- Spezies Schweine-Torque-Teno-Virus (Torque teno sus virus 1a, Porcine torque teno virus 1, TTV31-Sd, Typusspezies)
- Spezies Torque teno sus virus 1b (Porcine torque teno virus 2)
- Gattung Kappatorquevirus
- Gattung Lambdatorquevirus
- Spezies Torque teno zalophus virus 1 (Typusspezies)
- Gattung Mutorquevirus
- Spezies Torque teno equus virus 1 (Typusspezies)
- Gattung Nutorquevirusv
- Spezies Torque teno seal virus 4 (Typusspezies)
- Gattung Gyrovirus[8]
- ohne Gattungszuordnung (doppelte Anführungszeichen für Vorschläge):
- Spezies „Katzen-Torque-Teno-Virus“ („Feline anellovirus“, TTV4-Fc)
- Spezies „Nachtaffen-Torque-Teno-Virus“ (TTV3-At)
- Spezies „Tamarin-Torque-Teno-Virus“ (TTV2-So)
Weblinks
Einzelnachweise
- F. Bernardin, E. Operskalski, M. Busch, E. Delwart: Transfusion transmission of highly prevalent commensal human viruses. In: Transfusion. Band 50, Nr. 11, Mai 2010, S. 2474–2483, doi:10.1111/j.1537-2995.2010.02699.x, PMID 20497515.
- ViralZone: Anelloviridae. ExPASy. Abgerufen am 31. August 2019.
- Vasilyev EV, Trofimov DY, Tonevitsky AG, Ilinsky VV, Korostin DO, Rebrikov DV: Torque Teno Virus (TTV) distribution in healthy Russian population. In: Virology Journal. 6, 2009, S. 134. doi:10.1186/1743-422X-6-134. PMID 19735552. PMC 2745379 (freier Volltext).
- Caroline Tochetto, Ana Paula Muterle Varela, Diane Alves de Lima, Márcia Regina Loiko, Camila Mengue Scheffer, Willian Pinto Paim, Cristine Cerva, Candice Schmidt, Samuel Paulo Cibulski, Lucía Cano Ortiz, Sidia Maria Callegari Jacques, Ana Cláudia Franco, Fabiana Quoos Mayer, Paulo Michel Roehe: Viral DNA genomes in sera of farrowing sows with or without stillbirths, in: PLoS ONE 15(3), e0230714, 26. März 2020, doi:10.1371/journal.pone.0230714
- ICTVdB Management (2006). 00.107.0.01. Anellovirus. In: Büchen-Osmond, C. (Hrsg.): ICTVdB—The Universal Virus Database, version 4. Columbia University, New York, USA
- ICTV: ICTV MSL #34v 2018b.v2, März 2019
- NCBI: Torque teno sus virus k2a (species)
- SIB: Gyrovirus, auf: ViralZone
- Science Direct: Anelloviridae. „Members of the family Anelloviridae have features in common with Chicken anaemia virus, the type species of genus Gyrovirus, family Circoviridae“
- Michael J. Tisza, Diana V. Pastrana, Nicole L. Welch, Brittany Stewart, Alberto Peretti, Gabriel J. Starrett, Yuk-Ying S. Pang, Siddharth R. Krishnamurthy, Patricia A. Pesavento, David H. McDermott, Philip M. Murphy, Jessica L. Whited, Bess Miller, Jason Brenchley, Stephan P. Rosshart, Barbara Rehermann, John Doorbar, Blake A. Ta'ala, Olga Pletnikova, Juan C. Troncoso, Susan M. Resnick, Ben Bolduc, Matthew B. Sullivan, Arvind Varsani, Anca M. Segall, Christopher B. Buck: Discovery of several thousand highly diverse circular DNA viruses, in: eLife 9, 4. Februar 2020, e51971; doi:10.7554/eLife.51971. Fig. 2