Pithovirus
„Pithovirus“ (von altgriechisch πίθος píthos, deutsch ‚Vorratsgefäß‘, siehe auch die – nicht ganz korrekte Übersetzung – Büchse der Pandora) ist eine (mit Stand April 2020) noch nicht vom International Committee on Taxonomy of Viruses (ICTV) offiziell anerkannte Gattung innerhalb einer eigenen vorgeschlagenen Familie „Pithoviridae“ von Riesenviren des Phylums Negarnaviricota (veraltet Nucleocytoplasmic large DNA viruses, NCLDV). Die Namensgebung der Ordnung Pimascovirales (veraltet: PMI-Gruppe oder MAPI-Superklade)[4] – das ‚P‘ steht immer für ‚Pitho-‘ – hat das ICTV aber zu erkennen gegeben, dass es entsprechend allgemeiner Ansicht die Pithoviren als dieser Gruppe (Ordnung) zugehörig ansieht.
| „Pithovirus“ | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|

Skizze von „Pithovirus sibericum“ | ||||||||||||||||
| Systematik | ||||||||||||||||
| ||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||
| ||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||
| „Pithovirus“ | ||||||||||||||||
| Links | ||||||||||||||||
|
Entdeckung
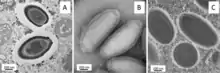
Die erste Spezies „Pithovirus sibericum“ der Gattung wurde 2014 aus über 30.000 Jahre altem sibirischem Permafrostboden isoliert. Es ist bis dato das älteste Eukaryoten infizierende DNA-Virus, das wieder zu einer Infektion gebracht werden konnte. Dies lässt vermuten, dass aus dem auf Grund der globalen Erwärmung auftauenden Permafrostboden noch weitere infektiöse Viren oder Mikroorganismen freigesetzt werden könnten. Um im Boden nach noch infektiösen Viren zu suchen, wurde eine Probe aus spät-pleistozänen Sedimenten des Kolyma-Tieflands steril entnommen und diese mit einer Kultur von Acanthamoeba castellanii als möglichem Wirt für Viren inkubiert. Nach der Vermehrung in Acanthamoeba wurden die Viruspartikel gereinigt und mittels Transmissionselektronenmikroskopie und Genomsequenzierung analysiert.
Levasseur und Kollegen haben 2016 eine weitere Spezies in dieser Gattung, „P. massiliensis“, isoliert.[5]
Eigenschaften
Aufbau
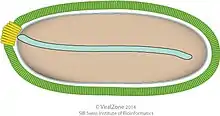
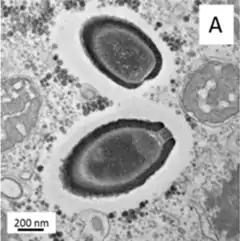
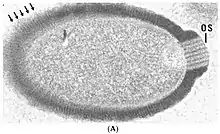
„P. sibericum“ ist bereits lichtmikroskopisch als eiförmige Partikel zu erkennen. Diese besitzen eine Länge von etwa 1,5 µm und eine Breite von etwa 500 nm. Elektronenmikroskopisch lässt sich eine etwa 60 nm dicke Virushülle erkennen, die parallele, aufrechte Streifen aufweist. Die Öffnung an der Spitze der Virushülle wird durch eine etwa 160 nm breite und 80 nm dicke Struktur aus 15 nm breiten Stäben verschlossen, die in der Aufsicht ein hexagonales Gittermuster aufweist und mit einer aufgerollten Membran im Inneren verbunden zu sein scheint. Im Inneren der Virushülle liegt ein membranumschlossenes Kompartiment, das keine inneren Strukturen aufweist außer einer gelegentlich sichtbaren 50 nm durchmessenden elektronendichten Kugel und einer schlauchförmigen Struktur entlang der Längsachse des Virus.
„P. sibericum“ unterscheidet sich damit von den äußerlich ähnlichen Pandoraviren durch die korkenartige Struktur an der Hüllenspitze und das deutlich kleinere, AT-reichere Genom mit weniger Genen.
Lebenszyklus
Der Lebenszyklus von „P. sibericum“ ist aus Kulturen mit Acanthamoeba bekannt und beginnt mit der Phagocytose der Viruspartikel durch den Wirt. Nach dem Verlust der korkenartigen Struktur fusioniert die virale Membran mit der Vakuolenmembran des Wirts, so dass sich eine kanalartige Verbindung des viralen Kompartiments mit dem Wirtscytoplasma bildet. Vier bis sechs Stunden nach der Infektion wird eine klare Zone im Wirtscytoplasma sichtbar, in der sich zahlreiche Vesikel sammeln. Die Virushülle und das innere Kompartiment werden gemeinsam gebildet, wobei zuerst rechteckige, geschlossene Partikel, bereits mit der charakteristischen korkenartigen Struktur, entstehen, deren äußere Wand sich später verdickt und die dann ihre eiförmige Gestalt annehmen. Die gestreifte, äußerste Schicht wird am Ende und in einzelnen Stücken aufgebaut. Am Ende wird die Wirtszelle aufgelöst und hunderte von Viruspartikeln (Virionen) werden entlassen.
Systematik
Innere Systematik
Die folgende Liste umfasst lediglich die oben erwähnten Vorschläge für zugehörige Virenspezies. Weder die Gattung als solche, noch einzelne Spezies sind bisher vom ICTV offiziell anerkannt (Stand April 2019):
- Familie „Pithoviridae“
Äußere Systematik
Inzwischen wurden weitere Viren gefunden, die offenbar ins Umfeld der Gattung „Pithovirus“ gehören (auch wenn sie vom ICTV mit Stand vom November 2018 ebenfalls noch nicht offiziell aufgenommen wurden):
- Gattung „Cedratvirus“ (mit Spezies „Cedratvirus A11“)[13][10]
- Gattung „Orpheovirus“ (mit Spezies „Orpheovirus IHUMI-LCC2“)[8][10]
- Spezies „Solivirus“[14][3] und
- Spezies „Solumvirus“.[15][3] sind vorgeschlagene Spezies aus einer Metagenomanalyse von Waldbodenproben durch F. Schulz und Kollegen 2018.[16]
- Spezies „Sissivirus S55“[3]
- Spezies „Misannotatedvirus“[3] alias „misidentified virus“[10]
- Aus Metagenomanalysen vom Schwarzen Raucher Lokis Schloss (englisch Loki’s castle) stammen weitere Vertreter (LCVs: Loki’s Castle Viruses, Pitho-like), die sich teilweise zu Kladen gruppieren. Es sind die folgenden vier Kladen: Klade 1: LCPAC101, LCPAC102, LCPAC103; Klade 2: LCPAC104, LCPAC201, LCPAC102; Klade 3: LCPAC302, LCPAC304; Klade 4: LCPAC401, LCPAC403, LCPAC404, LCPAC406; sowie jeweils für sich LCPAC001, LCDPAC01 und LCDPAC02.[17]
Für die um diese Funde erweiterte Familie der „Pithoviridae“ schlagen Schulz et al. folgende Systematik innerhalb der NCLDV vor:[16]
| „Pithovirus-like viruses“[4] („Pithoviridae“[18] s. l.) |
| ||||||||||||||||||||||||
Dies ist in Übereinstimmung mit der Systematik bei CNRS (2018)[19] und Rolland et al. (2019) Fig. 2a. – Fig. 2b zeigt dort ein etwas anderes Bild:[3]
| „Pithovirus-like viruses“[4] („Pithoviridae“[18]s. l.) |
| ||||||||||||||||||||||||||||||||||||
Hier steht jetzt nicht mehr „Solivirus“, sondern „Solumnvirus“ basal, was zeigt, dass 2019 offenbar noch keine stabile Aussage getroffen werden konnte.
Ähnlich wie Mimivirus war „Misannotatedvirus“ alias „misidentified virus“ wegen seiner Größe zunächst für ein Bakterium (zu Rickettsiales) gehalten worden, woraus sich der aktuelle Namensvorschlag ableitet.[3][10]
Einige Autoren rechnen Orpheovirus und Verwandte ebenfalls zu den Pithoviridae, d. h. interpretieren diese Familie im weiteren Sinn identisch mit Pithovirus-like viruses im obigen Kladogramm.[20]
Zur phylogenetischen Stellung siehe Disa Bäckström et al. (2019), Fig. 3.[17]
Literatur
- Matthieu Legendre, Julia Bartoli, Lyubov Shmakova, Sandra Jeudy, Karine Labadie, Annie Adrait, Magali Lescot, Olivier Poirot, Lionel Bertaux, Christophe Bruley, Yohann Couté, Elizaveta Rivkina, Chantal Abergel, Jean-Michel Claveri: Thirty-thousand-year-old distant relative of giant icosahedral DNA viruses with a pandoravirus morphology. In: PNAS. Band 111, Nr. 11, 2014, S. 4274–4279, doi:10.1073/pnas.1320670111 (englisch, freier Volltext).
Weblinks
- Julia Merlot: Sibirien: 30.000 Jahre eingefrorenes Virus zum Leben erweckt (Spiegel Online)
- Kenta Okamoto, Naoyuki Miyazaki, Chihong Song, Filipe R. N. C. Maia, Hemanth K. N. Reddy, Chantal Abergel, Jean-Michel Claverie, Janos Hajdu, Martin Svenda, Kazuyoshi Murata: Structural variability and complexity of the giant Pithovirus sibericum particle revealed by high-voltage electron cryo-tomography and energy-filtered electron cryo-microscopy, in: Scientific Reports, Band 7, Nr. 13291, 16. Oktober 2017, doi:10.1038/s41598-017-13390-4

Anmerkungen
- Das Material wurde von dieser Quelle kopiert, die unter einer Creative Commons Attribution 4.0 International License verfügbar ist.
Einzelnachweise
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- Eugene V. Koonin, Natalya Yutin: Evolution of the Large Nucleocytoplasmatic DNA Viruses of Eukaryotes and Convergent Origins of Viral Gigantism, in: Advances in Virus Research, Band 103, AP 21. Januar 2019, doi:10.1016/bs.aivir.2018.09.002, S. 167–202.
- Clara Rolland, Julien Andreani, Amina Cherif Louazani, Sarah Aherfi, Rania Francis, Rodrigo Rodrigues, Ludmila Santos Silva, Dehia Sahmi, Said Mougari, Nisrine Chelkha, Meriem Bekliz, Lorena Silva, Felipe Assis, Fábio Dornas, Jacques Yaacoub Bou Khalil, Isabelle Pagnier, Christelle Desnues, Anthony Levasseur, Philippe Colson, Jônatas Abrahão, Bernard La Scola: Discovery and Further Studies on Giant Viruses at the IHU Mediterranee Infection That Modified the Perception of the Virosphere, in: Viruses 11(4), März/April 2019, pii: E312, doi:10.3390/v11040312, PMC 6520786 (freier Volltext), PMID 30935049.
 Siehe insbes. Fig. 2a und b.
Siehe insbes. Fig. 2a und b. - Julien Guglielmini, Anthony C. Woo, Mart Krupovic, Patrick Forterre, Morgan Gaia: Diversification of giant and large eukaryotic dsDNnA viruses predated the origin of modern eukaryotes, in: PNAS, Band 116, Nr. 39, 10./24. September 2019, S. 19585–19592, doi:10.1073/pnas.1912006116, PMID 31506349, Fig. 2
- Anthony Levasseur, Julien Andreani, Jeremy Delerce, Jacques Bou Khalil, Catherine Robert, Bernard La Scola, Didier Raoult: Comparison of a Modern and Fossil Pithovirus Reveals Its Genetic Conservation and Evolution. In: Genome Biology and Evolution. Band 8, Nr. 8, Juli 2016, ISSN 1759-6653, S. 2333–2339, doi:10.1093/gbe/evw153, PMID 27389688, PMC 5010891 (freier Volltext) – (englisch).
-
Steven W. Wilhelm, Jordan T. Bird, Kyle S. Bonifer, Benjamin C. Calfee, Tian Chen, Samantha R. Coy, P. Jackson Gainer, Eric R. Gann, Huston T. Heatherly, Jasper Lee, Xiaolong Liang, Jiang Liu, April C. Armes, Mohammad Moniruzzaman, J. Hunter Rice, Joshua M. A. Stough, Robert N. Tams, Evan P. Williams, Gary R. LeCleir: A Student’s Guide to Giant Viruses Infecting Small Eukaryotes: From Acanthamoeba to Zooxanthellae, in: Viruses, Band 9, Nr. 3, doi:10.3390/v9030046.

- David M. Needham, Alexandra Z. Worden et al.: A distinct lineage of giant viruses brings a rhodopsin photosystem to unicellular marine predators, in: PNAS, 23. September 2019, doi:10.1073/pnas.1907517116, ISSN 0027-8424, hier: Supplement 1 (xlsx)
- Julien Andreani, Jacques Y. B. Khalil, Emeline Baptiste, Issam Hasni, Caroline Michelle, Didier Raoult, Anthony Levasseur, Bernard La Scola: Orpheovirus IHUMI-LCC2: A New Virus among the Giant Viruses. In: Frontiers in Microbiology. Band 8, 22. Januar 2018, ISSN 1664-302X, doi:10.3389/fmicb.2017.02643 (englisch).
- NCBI: Pithovirus sp. LC8 (species)
- Julien Andreani, Jonathan Verneau, Didier Raoult, Anthony Levasseurn Bernard La Scola: Deciphering viral presences: two novel partial giant viruses detected in marine metagenome and in a mine drainage metagenome, in: Virology Journal, Band 15, Nr. 66, 10. April 2018, doi:10.1186/s12985-018-0976-9
- NCBI: Pithovirus sibericum (species)
- NCBI: Pithovirus lacustris (species)
- Julien Andreani, Sarah Aherfi, Jacques Yaacoub Bou Khalil, Fabrizio Di Pinto, Idir Bitam, Didier Raoult, Philippe Colson, Bernard La Scola: Cedratvirus, a Double-Cork Structured Giant Virus, is a Distant Relative of Pithoviruses. In: Viruses / Molecular Diversity Preservation International. Band 8, Nr. 11, November 2016, ISSN 1999-4915, S. 300, doi:10.3390/v8110300, PMID 27827884, PMC 5127014 (freier Volltext) – (englisch).
- NCBI: Solivirus (species)
- NCBI: Solumvirus (species)
- Frederik Schulz, Lauren Alteio, Danielle Goudeau, Elizabeth M. Ryan, Feiqiao B. Yu, Rex R. Malmstrom, Jeffrey Blanchard, Tanja Woyke: Hidden diversity of soil giant viruses. In: Nature Communications. Band 9, Nr. 1, 2018, ISSN 2041-1723, Artikel 4881, Anmerkung 38, doi:10.1038/s41467-018-07335-2 (englisch, nature.com).
- Disa Bäckström, Natalya Yutin, Steffen L. Jørgensen, Jennah Dharamshi, Felix Homa, Katarzyna Zaremba-Niedwiedzka, Anja Spang, Yuri I. Wolf, Eugene V. Koonin, Thijs J. G. Ettema; Richard P. Novick (Hrsg.): Virus Genomes from Deep Sea Sediments Expand the Ocean Megavirome and Support Independent Origins of Viral Gigantism, in: mBio Band 10, Nr. 2, 5. März 2019, doi:10.1128/mBio.02497-18, PMID 30837339
- Eugene V. Koonin, Natalya Yutin: Multiple evolutionary origins of giant viruses, in: F1000 Research, 22. November 2018, doi:10.12688/f1000research.16248.1, version 1
- Centre national de la recherche scientifique: List of the main “giant” viruses known as of today, Université Aix Marseille, 18. April 2018
- NCBI: Pithoviridae (family)