Carlavirus
Carlavirus, früher bekannt als Carnation latent virus group und Carlavirus group, ist eine Gattung von Viren in der Ordnung Tymovirales, Familie Betaflexiviridae, Unterfamilie Quinvirinae. Das Genom ist eine Einzelstrang-RNA mit positiver Polarität, (+)ssRNA (Gruppe IV der Baltimore-Klassifikation). Die Virionen (Viruspartikel) sind gerade oder leicht gewundene Filamente. Als natürliche Wirte dienen Pflanzen (Pflanzen- oder Phytoviren). Derzeit (Stand März 2021) gibt in dieser Gattung es vom International Committee on Taxonomy of Viruses (ICTV) bestätigt 53 Spezies (Arten), einschließlich der Typusart Carnation latent virus. Zu den mit dieser Gattung assoziierten Krankheiten gehören: Mosaik- und Ringspot-Symptome.[3][2]
| Carlavirus | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
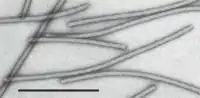
Virionen von Carnation latent virus (CLV) | ||||||||||||||||||
| Systematik | ||||||||||||||||||
| ||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||
| ||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||
| Carlavirus | ||||||||||||||||||
| Links | ||||||||||||||||||
|
_p28_Fig._21.png.webp)
Etymologie
Der Gattungsname Carlavirus ist eine Kombination aus der ersten Silbe der ersten beiden Wortteile im Namen der Typusart Carnation latent virus.
Beschreibung
Die Gattung Carlavirus wird im 9. Bericht des ICTV (2009) beschrieben.
Morphologie
Die Virionen (Virusteilchen) der Gattung sind nicht umhüllt, fadenförmig (filamentös) gerade oder leicht gebogen mit Helixsymmetrie. Ihre Länge beträgt grob 470–700 nm (im Extremfall 310–1000 nm, die Angaben schwanken je nach Autor) und 12–15 nm im Durchmesser bei einer Ganghöhe von etwa 3,4 nm.[4][3]
Genom
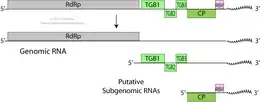
Das Genom besteht aus einem einzelsträngigen RNA-Molekül positiver Polarität von üblicherweise 7,4–7,7 kb (Kilobasen), ggf. auch (je nach Autor) 5,8–9,0 kb.[4][3]
Die Gattung zeichnet sich dadurch aus, dass sie sechs Offenen Leserahmen (Open Reading Frames, ORFs) mit kurzen untranslatierten Regionen (UTRs) an den Enden: Der 3'-Terminus ist polyadenyliert, bei einigen Arten ist das 5'-Ende mit einer Cap-Struktur versehen.[4][3]
Das Genom kodiert für 3 bis 6 Proteine, darunter ein Kapsidprotein, das sich am 3'-Ende befindet, und eine RNA-abhängige RNA-Polymerase, die sich am 5'-Ende des Genoms befindet. Beim Kartoffel-M-Virus kodiert ORF1 für ein Polypeptid mit 223 kDa (Kilodalton), das die Replikase des Virus darstellt.[4][3]
Die sich überlappenden Gene/ORFs 2, 3 und 4 bilden einen Block (Triple Gene Block, TGB) und kodieren für Polypeptide von 25, 12 und 7 kDa. Diese Triple-Gen-Block-Proteine (Triple Gene Block Proteins, TGBp: TGBp1, TGBp2 und TGBp3) ermöglichen oder erleichtern als Movement-Proteine die Bewegung der Virionen von Zelle zu Zelle und über große Distanzen. Einen solchen TGB findet man bei allen Betaflexiviridae mit Ausnahme der Gattungen Capillovirus, Citrivirus, Trichovirus, und Vitivirus, die ein einfaches Movement-Protein (MP) haben.[4][3]
ORF5 kodiert für ein Kapsidprotein (CP) mit 34 kDa und überlappt mit ORF6, der für ein Cystein-reichen Protein mit 11–16 kDa kodiert.[4][3]
RBP ist ein RNA-bindendes Protein:[3] ORF 5 überlappt mit ORF 6, der für ein Cystein-reichen Polypeptids mit 11–16 kDa kodiert, dessen Funktion noch nicht ganz geklärt ist. Seine Fähigkeit, Nukleinsäure zu binden, lässt vermuten, dass es die Übertragung durch Blattläuse erleichtern, oder am Gen-Silencing des Wirts oder der viralen RNA-Replikation beteiligt sein könnte.[4]
Replikationszyklus
Die virale Replikation erfolgt im Zytoplasma der Wirtszelle und ist lysogen. Der Eintritt in die Wirtszelle erfolgt durch Eindringen in diese. Die Replikation folgt dem Modell der Replikation von Einzelstrang-RNA-Viren positiver Polarität. Die Transkription erfolgt nach dem Modell von Einzelstrang-RNA-Viren positiver Polarität.
Übertragung
Die Viren werden durch Insekten (Blattläuse) als Vektoren übertragen.[4][3] Die Infektion wird manchmal durch Blattläuse in einem semi-persistenten Modus verbreitet, d. h. der Vektor ist für einige Stunden infektiös.[5] Einige Arten werden durch die Tabakmottenschildlaus (Bemisia tabaci) in einem semi-persistenten Modus oder durch das Saatgut übertragen.[6] Die meisten Arten infizieren nur einige wenige Wirte und verursachen Infektionen mit wenigen oder keinen Symptomen, z. B. American hop latent virus (AHLV) und Lily symptomless virus (LSV). Andere, wie das Blueberry scorch virus (BlSV) und das Poplar mosaic virus (PMV, Pappel-Mosaik-Virus), verursachen aber schwere Krankheiten.[7]
Systematik
Die Gattung wurde erstmals im ersten Bericht der ICTV 1971 als Carnation latent virus group vorgeschlagen, wurde aber 1975 in Carlavirus group und 1995 (6. Bericht) in die Gattung Carlavirus umbenannt. Im Jahr 2005 (8. Bericht) wurde es in die Familie der Flexiviridae gestellt, nachdem es zuvor nicht zugeordnet war.[8] Die aktuelle Position im 9. Bericht (2009) als Gattung der Familie Betaflexiviridae ergibt sich aus der späteren Unterteilung der Flexiviridae.[2]
Ordnung: Tymovirales
Familie: Betaflexiviridae
● Unterfamilie: Quinvirinae
- Gattung: Carlavirus
- Spezies: Aconitum latent virus
- Spezies: American hop latent virus (AHLV)
- Spezies: Atractylodes mottle virus
- Spezies: Blueberry scorch virus (BlSV)
- Spezies: Butterbur mosaic virus (Pestwurz-Mosaikvirus)
- Spezies: Cactus virus 2
- Spezies: Caper latent virus
- Spezies: Carnation latent virus (CLV, Typus)[9]
- Spezies: Chrysanthemum virus B
- Spezies: Cole latent virus
- Spezies: Coleus vein necrosis virus
- Spezies: Cowpea mild mottle virus (CPMMV)
- Spezies: Cucumber vein-clearing virus
- Spezies: Daphne virus S (Seidelbastvirus S)
- Spezies: Gaillardia latent virus
- Spezies: Garlic common latent virus
- Spezies: Helenium virus S (Sonnenbrautvirus S)
- Spezies: Helleborus mosaic virus (Nieswurz-Mosaikvirus)
- Spezies: Helleborus net necrosis virus
- Spezies: Hippeastrum latent virus
- Spezies: Hop latent virus (HPLV, unterscheide: Hop latent viroid, HpLVd, Gattung Cocadviroid)
- Spezies: Hop mosaic virus (HpMV, Hopfen-Mosaikvirus)
- Spezies: Hydrangea chlorotic mottle virus (HdCMV)
- Spezies: Kalanchoe latent virus
- Spezies: Ligustrum necrotic ringspot virus
- Spezies: Ligustrum virus A (Ligustervirus A)
- Spezies: Lily symptomless virus (LSV, Lily-symptomless-Virus, Symptomloses Lilienvirus)
- Spezies: Melon yellowing-associated virus
- Spezies: Mirabilis jalapa mottle virus
- Spezies: Narcissus common latent virus
- Spezies: Nerine latent virus
- Spezies: Passiflora latent virus
- Spezies: Pea streak virus (PeSV)
- Spezies: Phlox virus B (Phloxvirus B)
- Spezies: Phlox virus M (Phloxvirus M)
- Spezies: Phlox virus S (Phloxvirus S)
- Spezies: Poplar mosaic virus (PMV, Pappel-Mosaikvirus)
- Spezies: Potato latent virus (PotLV)
- Spezies: Potato virus H (PVH, Kartoffelvirus H)
- Spezies: Potato virus M (PVM, Kartoffelvirus M)
- Spezies: Potato virus P (PVP, Kartoffelvirus P)
- Spezies: Potato virus S (PVS, Kartoffelvirus S)
- Spezies: Red clover vein mosaic virus (RCVMV, Rotklee-Adernmosaikvirus)
- Spezies: Sambucus virus C (Holundervirus C)
- Spezies: Sambucus virus D (Holundervirus D)
- Spezies: Sambucus virus E (Holundervirus E)
- Spezies: Shallot latent virus (SLV, SLV000)
- Spezies: Sint-Jan onion latent virus (SjoLV, SJOLV0)
- Spezies: Strawberry pseudo mild yellow edge virus
- Spezies: Sweet potato C6 virus (SPC6V, SPC6V0, Süßkartoffelvirus C6)
- Spezies: Sweet potato chlorotic fleck virus (SPCFV)
- Spezies: Verbena latent virus
- Spezies: Yam latent virus
Literatur
- Giovanni P. Martelli, Pasquale Saldarelli: Carlavirus. In: Christian Tidona, Gholamreza Darai (Hrsg.): The Springer Index of Viruses. Springer Science & Business Media, New York 2012, ISBN 978-0-387-95919-1, S. 521–532, doi:10.1007/978-0-387-95919-1_75 (eingeschränkte Vorschau in der Google-Buchsuche – Leseprobe).
- Suzanne Astier, Josette Albouy, Yves Maury: Principles of Plant Virology: Genome, Pathogenicity, Virus Ecology. Science Publishers, Enfield 2007, ISBN 978-1-57808-503-3, S. 78.
- Carlavirus Isolation and RNA Extraction. In: Gary D. Foster, Sally C. Taylor (Hrsg.): Plant Virology Protocols: From Virus Isolation to Transgenic Resistance. Humana Press, New York 1998, ISBN 0-89603-385-6, S. 145, doi:10.1385/0-89603-385-6:145.
- David Pimentel: Encyclopedia of Pest Management. Marcel Dekker, New York 2002, ISBN 0-8247-0632-3, S. 407 (eingeschränkte Vorschau in der Google-Buchsuche – Leseprobe).
- Andrew M. Q. King: Genus Carlavirus. In: Virus Taxonomy: Ninth Report of the International Committee on Taxonomy of Viruses. Ninth report of the International Committee on Taxonomy of Viruses. Elsevier, London 2012, ISBN 978-0-12-384684-6, S. 924 (eingeschränkte Vorschau in der Google-Buchsuche).
Weblinks
- Taxon: Genus Carlavirus (virus) The Taxonomicon
- Viralzone: Carlavirus
- (Seite nicht mehr abrufbar, Suche in Webarchiven: ICTV)
Einzelnachweise
- ICTV Master Species List 2018b v1 MSL #34, Feb. 2019
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- Viral Zone. ExPASy. Abgerufen am 21. April 2021.
- ICTV: ICTV 9th Report (2011) – Betaflexiviridae
- David Pimentel: Encyclopedia of Pest Management. Marcel Dekker, New York 2002, ISBN 0-8247-0632-3, S. 407 (eingeschränkte Vorschau in der Google-Buchsuche – Leseprobe).
- Suzanne Astier, Josette Albouy, Yves Maury: Principles of Plant Virology: Genome, Pathogenicity, Virus Ecology. Science Publishers, Enfield 2007, ISBN 978-1-57808-503-3, S. 78.
- Carlavirus Isolation and RNA Extraction. In: Gary D. Foster, Sally C. Taylor (Hrsg.): Plant Virology Protocols: From Virus Isolation to Transgenic Resistance. Humana Press, New York 1998, ISBN 0-89603-385-6, S. 145, doi:10.1385/0-89603-385-6:145.
- M. J. Adams, J. F. Antoniw, M. Bar-Joseph, A. A. Brunt, T. Candresse, G. D. Foster, G. P. Martelli, R. G. Milne, S. K. Zavriev, C. M. Fauquet: The new plant virus family Flexiviridae and assessment of molecular criteria for species demarcation. In: Archives of Virology. Band 149, Nr. 5, Mai 2004, ISSN 0304-8608, S. 1045–1060, doi:10.1007/s00705-004-0304-0, PMID 15098118.
- ICTVdB: Carnation latent virus (via WebArchiv vom 11. September 2006)


