Flügeldeformationsvirus
Das Flügeldeformationsvirus (wissenschaftlich englisch Deformed Wing Virus, DWV) ist ein RNA-Virus aus der Gattung der Iflaviren (Iflaviridae), das über ein einsträngiges RNA-Genom verfügt und pathologische Effekte bei Honigbienen auslöst, weshalb das Virus auch Gegenstand eingehender wissenschaftlicher Untersuchungen ist. Dieser Umstand ist nicht zuletzt darauf zurückzuführen, dass es ganze Bienenkolonien vernichten und dadurch erhebliche wirtschaftliche Schäden verursachen kann. Es ist einer von 22 bekannten pathogenen Viren der Honigbiene (siehe auch Liste von Bienenkrankheiten) und weiterer Bienenarten (z. B. Dunkle Erdhummel und Ackerhummel).[3] Symptome bei adulten Bienen sind Verformungen der Flügel, aufgeblähter Hinterleib und sichtbare Verfärbungen.[4] Infizierte Larven sind nicht lebensfähig und sterben unmittelbar nach dem Schlüpfen. Entdeckt wurde das Virus in den frühen 1980er Jahren in Japan.[3]
| Flügeldeformationsvirus | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
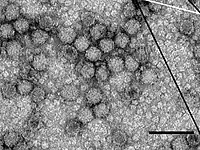
TEM-Aufnahme von Virionen des Flügeldeformationsvirus (DWV), Skalenlänge 100 nm | ||||||||||||||||||
| Systematik | ||||||||||||||||||
| ||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||
| ||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||
| Deformed wing virus | ||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||
| DWV | ||||||||||||||||||
| Links | ||||||||||||||||||
|


Beschreibung
Das Virus bildet einen 10140 Nukleotide langen, einsträngigen RNA-Strang mit einem einheitlichen Leseraster, das beiderseits von nicht-kodierenden Sequenzabschnitten eingefasst wird. Die Genabfolge auf der RNA entspricht derjenigen von Picornaviren der Säugetiere (Syntänie). Die RNA wird translatiert in ein einheitliches Polyprotein, das später durch spezifische Proteasen in die funktionalen Proteine zerlegt wird. Wie bei anderen Picornaviren sitzen dabei die Strukturproteine hintereinander nahe dem N-Terminus des Proteins, die nichtstrukturellen Proteine nahe dem C-Terminus. In der infektiösen Form bildet das Virus Kapside als Ikosaeder-förmige Partikel von etwa 30 Nanometer Durchmesser, deren Hülle aus drei Hüllproteinen aufgebaut wird.[5]
Auslöser
Bei Honigbienen wird das Virus neben anderen Viren über die parasitäre Varroamilbe verbreitet. Inzwischen geht man davon aus, dass das Agens (hauptsächliche Ursache) der Varroose (Varroamilbenbefall) der Hämolymphverlust nicht durch die parasitären Varroamilben, sondern durch die Virusinfektion ist.
Honigbienen können das Flügeldeformationsvirus auf Wildbienen und Hummeln übertragen.[6][7]
Alle Entwicklungsstadien der Biene können befallen sein. Adulte (Erwachsene) zeigen bei Erstinfektion keine Symptome, dienen dem Erreger aber als Reservoir (es gibt Hinweise auf eine vertikale Infektion, also auf eine Infektion von Biene zu Biene). Morphologische Auffälligkeiten zeigen Adulte, die im Puppenstadium infiziert waren. Diese Tiere weisen eine Verkürzung des Hinterleibs (Abdomen), Verfärbungen und die namensgebenden Flügel-Verstümmelungen auf. Infizierte Tiere haben eine geringere Lebenserwartung.
Eine ursächliche Behandlung bei einer Infektion gibt es nicht. Indirekt können die üblichen Maßnahmen zur Eindämmung des Varroabefalls getroffen werden.[8]
Varianten und Abgrenzung
Das Flügeldeformationsvirus zeigt sehr hohe Übereinstimmung in der Sequenz zu anderen bienenpathogenen Viren wie Varroa destructor virus 1[9] (VDV-1) und dem Kakugo-Virus (KV, ein Subtyp des DWV): Viren, die alle miteinander rekombinieren.[10] Das Virus ist, in nicht-pathogener Form, in Honigbienen weltweit verbreitet. In dieser Form bleibt die Infektion symptomlos, das Virus wird vertikal, als Provirus eingebaut im Genom der Wirtsart, weitergegeben. Infektiöse Stämme des Virus treten, soweit bekannt, ausschließlich in Präsenz von Varroa auf. Der Grund für dieses Verhalten ist noch nicht mit Sicherheit bekannt. Nachgewiesen wurde, dass sich die infektiöse Form, die durch weitaus höhere Gehalte viraler RNA im Wirt gekennzeichnet ist, nur bei direkter Übertragung in die Hämolymphe der Biene ausbilden kann. Neben der Verletzung durch den Stich von Varroa kann dies auch durch experimentelle Injektion erfolgen.[11]
Seit 2016 unterscheidet man die Gen-Varianten DWV-A und DWV-B. Letztere tötet Bienen schneller als DWV-A und ist zudem ansteckender als die verbreitete Form DWV-A.[12]
Das Kakugo-Virus besitzt gegenüber DWV-Viren eine RNA-Sequenz mit rund 98 % Übereinstimmung. Es befällt den Pilzkörper, eine paarig organisierte anatomische Struktur im Zentralhirn der Bienen. Er dient als Riechzentrum, spielt aber auch eine wichtige Rolle bei höheren integrativen Leistungen wie Lernen und Gedächtnis. Mit dem Virus infizierte Bienen zeigen abnormal aggressives Verhalten.
Literatur
- Gaetana Lanzi, Joachim R. de Miranda, Maria Beatrice Boniotti, Craig E. Cameron, Antonio Lavazza, Lorenzo Capucci, Scott M. Camazine, Cesare Rossi: Molecular and Biological Characterization of Deformed Wing Virus of Honeybees (Apis mellifera L.). In: Journal of Virology. Band 80, Nr. 10, 15. Mai 2006, S. 4998–5009, doi:10.1128/JVI.80.10.4998-5009.2006 (englisch, asm.org [PDF]).
Weblinks
- Stellungnahme des Bundesamts für Verbraucherschutz und Lebensmittelsicherheit, abgerufen am 18. April 2016.
- Martin Vieweg: Bienenerkrankung: Hoffnung im Kampf gegen Bienen-Virus, auf: wissenschaft.de vom 28. September 2021
Einzelnachweise
- ICTV Master Species List 2018b.v2. MSL #34, März 2019
- ICTV: ICTV Taxonomy history: Enterovirus C, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- Elke Genersch, Constanze Yue, Ingemar Fries, Joachim R. de Miranda: Detection of Deformed wing virus, a honey bee viral pathogen, in bumble bees (Bombus terrestris and Bombus pascuorum) with wing deformities. In: Journal of Invertebrate Pathology. Band 91, Nr. 1, Januar 2006, S. 61–63, doi:10.1016/j.jip.2005.10.002 (PDF).
- Joachim R. de Miranda, Elke Genersch: Deformed wing virus. In: Journal of Invertebrate Pathology. 103 (Supplement), Januar 2010, S. S48–S61, doi:10.1016/j.jip.2009.06.012.
- Gaetana Lanzi, Joachim R. de Miranda, Maria Beatrice Boniotti, Craig E. Cameron, Antonio Lavazza, Lorenzo Capucci, Scott M. Camazine, Cesare Rossi: Molecular and Biological Characterization of Deformed Wing Virus of Honeybees (Apis mellifera L.). In: Journal of Virology. Band 80, Nr. 10, 15. Mai 2006, S. 4998–5009, doi:10.1128/JVI.80.10.4998-5009.2006 (PDF).
- Robyn Manley, Ben Temperton, Toby Doyle, Daisy Gates, Sophie Hedges, Michael Boots, Lena Wilfert, Hillary Young: Knock‐on community impacts of a novel vector: spillover of emerging DWV‐B from ‐infested honeybees to wild bumblebees . In: Ecology Letters. 2019, doi:10.1111/ele.13323 (dt. Beitrag).
- Samantha A. Alger, P. Alexander Burnham, Humberto F. Boncristiani, Alison K. Brody, Olav Rueppell: RNA virus spillover from managed honeybees (Apis mellifera) to wild bumblebees (Bombus spp.). In: PLOS ONE. 14, 2019, S. e0217822, doi:10.1371/journal.pone.0217822 (dt. Beitrag).
- Stellungnahme der ZKBS zur Risikobewertung von Paenibacillus larvae ssp. larvae und des Flügeldeformationsvirus (DWV) gemäß § 5 Absatz 1 GenTSV vom 8. August 2006, abgerufen am 4. August 2016.
- ICTV: Master Species List 2018a v1 MSL including all taxa updates since the 2017 release. Fall 2018 (MSL #33)
- Siehe auch: Tomoko Fujiyuki et al.: Prevalence and phylogeny of Kakugo virus identified from aggressive worker honeybees (Apis mellifera L.) under various colony conditions, in: The IUSSI 2006 Congress, 1. August 2006
- Eugene V. Ryabov, Graham R. Wood, Jessica M. Fannon, Jonathan D. Moore, James C. Bull, Dave Chandler, Andrew Mead, Nigel Burroughs, David J. Evans (2014): A Virulent Strain of Deformed Wing Virus (DWV) of Honeybees (Apis mellifera) Prevails after Varroa destructor-Mediated, or In Vitro, Transmission. PLOS Pathogens 10(6): e1004230. doi:10.1371/journal.ppat.1004230
- McMahon, D.P., Natsopoulou, M.E., Doublet, V., Fürst, M.A., Weging, S., Brown, M.J.F., Gogol-Döring, A., Paxton, R.J.: Elevated virulence of an emerging viral genotype as a driver of honeybee loss. In: Proceedings of the Royal Society of London – Biological Sciences 20160811