Reverse Transkriptase
Reverse Transkriptasen (RT) sind enzymatisch wirksame Proteine, die als RNA-abhängige DNA-Polymerasen eine Transkription in umgekehrter Richtung (revers), nämlich von RNA in DNA, katalysieren; damit kann genetische Information von RNA in DNA umgeschrieben werden.[1]
| Reverse Transkriptase | ||
|---|---|---|
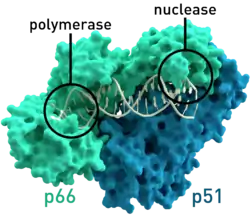
| ||
| Modell der reversen Transkriptase (Dimer) des HIV-1 | ||
| Enzymklassifikation | ||
| EC, Kategorie | 2.7.7.49, Nukleotidyltransferase | |
| Substrat | Deoxynucleosid-Triphosphat + DNA(n) | |
| Produkte | Diphosphat + DNA(n+1) | |
Biochemische Aspekte
Mittels ihrer RNA-abhängigen DNA-Polymerase-Aktivität wird zunächst nach Vorlage einer einzelsträngigen RNA durch Verknüpfung von komplementär gepaarten DNA-Bausteinen (Desoxyribonukleotide) ein Hybrid-Doppelstrang aus RNA und DNA aufgebaut. Danach wird dessen RNA-Anteil weitgehend abgebaut, vermittels einer RNase-H-Aktivität eines besonderen Abschnitts des Proteins. Der verbliebene DNA-Einzelstrang wird schließlich zum DNA-Doppelstrang ergänzt, katalysiert durch eine zusätzliche inhärente DNA-abhängige DNA-Polymerase-Aktivität der Reversen Transkriptase.
Die Fehlerhäufigkeit der Reversen Transkriptase liegt wegen einer fehlenden Korrekturfunktion[2] (proof-reading) bei 1:103 bis 1:104 und führt zu einer sehr hohen Mutationsrate.
Geschichte
Die Reverse Transkriptase aus Retroviren wurde erstmals 1970 sowohl von Howard Temin als auch, unabhängig, von David Baltimore beschrieben.[3][4] Sie erhielten 1975 für diese Entdeckung zusammen mit Renato Dulbecco den Nobelpreis für Physiologie oder Medizin. Der Zusatz revers kennzeichnet das eigenartige Vermögen dieses Enzyms, nach einer RNA-Vorlage eine DNA aufzubauen. Die unerwartete Prozessrichtung entkräftete die bis dahin vertretene Lehrmeinung, das sogenannte Zentrale Dogma der Molekularbiologie, der genetische Informationsfluss verlaufe ausschließlich in der Richtung DNA → RNA → Protein, niemals umgekehrt.[5][6]
Vorkommen
Die Reverse Transkriptase wurde zuerst in Retroviren (z. B. HIV, HTLV, SIV) entdeckt. Diese Viren mit RNA-Genom verwenden die RT, um ihr Genom in DNA umzuschreiben. Die RT erfüllt damit eine entscheidende Funktion bei der Vermehrung des Virus. Daneben enthalten auch bestimmte DNA-Viren wie die Hepadnaviren (z. B. der Erreger der Hepatitis B (HBV das Protein P), oder die bei Pflanzen auftretenden Caulimoviren) eine RT. Von ehemaligen, mutierten Retroviren stammen auch die Klasse-I-Transposons, auch Retroelemente genannt, ab. Diese benötigen für ihre Replikation eine RT. Diese wird entweder von ihnen selbst codiert (autonome LINEs und LTR-Retrotransposons) oder muss zur Verfügung gestellt werden (z. B. bei SINEs).
Gruppe II Introns codieren ebenfalls für eine Reverse Transkriptase, die eine enzymatisch aktive Intron-RNA (Ribozym) stabilisiert und die integrierte RNA in DNA umschreibt.[7] Die RNA katalysiert bei dem Vorgang das Spleißen. Gruppe II Introns wurden in Prokaryonten und in den Genomen von Organellen in Pilzen und Pflanzen nachgewiesen.
Eine Reverse Transkriptase ist ebenfalls Bestandteil der Telomerase von Eukaryoten, wo sie die im Zuge einer Replikation verkürzten Telomere wieder auf die ursprüngliche Länge erweitert und somit den Prozess der Zellalterung verzögert.[8] Die genauere Bezeichnung ist telomerase reverse transcriptase (TERT), wie z. B. für die humane telomerase reverse transcriptase (hTERT).[9]
Reverse Transkription
Das RNA-Genom von RNA-Viren, beispielsweise Retroviren, wird in doppelsträngige DNA umgeschrieben. Dieser Vorgang wird reverse Transkription genannt. Das Virus bringt die hierfür notwendige Reverse Transkriptase in seinen Viruspartikeln mit. Diese schreibt mithilfe eines tRNA-Primers die einzelsträngige RNA des Virus zunächst in einen komplementären DNA-Strang um (Aktivität als RNA-abhängige DNA-Polymerase). Danach wird die RNA abgebaut bis auf ein Fragment, das als zweiter Primer dient (Aktivität als Ribonuklease H). Damit entsteht anschließend die doppelsträngige DNA (Aktivität als DNA-abhängige DNA-Polymerase).
Das virale Genom der RNA liegt dann doppelsträngig als DNA-Kopie vor. Zudem wurden bei der reversen Transkription an beiden Enden der DNA-Stränge sogenannte LTR-Sequenzen generiert, die für den weiteren Ablauf der Infektion essenziell sind. Sie ermöglichen die Integration in das DNA-Genom der Wirtszelle – durch ein weiteres Enzym von Retroviren, eine Integrase.
Biotechnologische Anwendungen
Die Fehlerhäufigkeit der viralen Reversen Transkriptase erschwert die Bekämpfung von Retroviren wie HIV. Die Hemmung der Reversen Transkriptase ist ein Ziel der Kombinationstherapie und durch verschiedene Wirkstoffe (NNRTI, NRTI) möglich. Solche Reverse-Transkriptase-Hemmer waren die ersten und bis 1994 einzigen zur Behandlung der HIV-Infektion zugelassenen wirksamen Medikamente.
Die künstlich erzeugte Reverse Transkriptase "Xenopolymerase” ist nun in der Lage, Korrektur zu lesen und somit die Fehlerrate zu minimieren.[2]
Reverse Transkriptasen werden in der Molekularbiologie und in der molekularen Diagnostik eingesetzt, beispielsweise bei der RT-PCR oder um eine cDNA-Bank zu erstellen. Hierfür werden virale Reverse Transkriptasen aus dem Murine Leukemia Virus (MLV) oder dem Avian Myeloblastosis Virus (AMV) genutzt. In der Regel werden aber keine nativen Enzyme, sondern gentechnisch erzeugte Varianten mit geringerer Fehlerrate, niedrigerer RNase-H-Aktivität und höherer Temperaturstabilität eingesetzt.[10][11] Nachdem die Isolierung rekombinanter Gruppe II Intron-Reverse Transcriptasen gelungen ist, wird erwartet, dass diese aufgrund ihrer geringeren Fehlerrate, höheren Geschwindigkeit und Temperaturstabilität, insbesondere aber auch der Template-Switch-Aktivität, dem direkten Wechsel an das 3'-Ende neuer RNAs, an Bedeutung gewinnen.[12]
Einzelnachweise
- Racaniello, V. R. (Vincent R.), Rall, Glenn F., Skalka, Anna Marie, Enquist, L. W. (Lynn W.),: Principles of virology. 4. Auflage. Washington, DC, ISBN 978-1-55581-933-0, S. 189.
- Reverse Transcriptase with Proofreading Capabilities Created. Abgerufen am 27. Februar 2020 (englisch).
- H. M. Temin, S. Mizutani: RNA-dependent DNA polymerase in virions of Rous sarcoma virus. In: Nature. Band 226, Nummer 5252, Juni 1970, S. 1211–1213, PMID 4316301.
- D. Baltimore: RNA-dependent DNA polymerase in virions of RNA tumour viruses. In: Nature. Band 226, Nummer 5252, Juni 1970, S. 1209–1211, PMID 4316300.
- John M. Coffin, Stephen H. Hughes, Harold E. Varmus: The Place of Retroviruses in Biology. In: Retroviruses. Cold Spring Harbor Laboratory Press, 1997, ISBN 0-87969-571-4.
- Central dogma reversed. In: Nature. Band 226, Nummer 5252, Juni 1970, S. 1198–1199, PMID 5422595.
- A. M. Lambowitz, S. Zimmerly: Group II introns: mobile ribozymes that invade DNA. In: Cold Spring Harbor perspectives in biology. Band 3, Nummer 8, August 2011, S. a003616, doi:10.1101/cshperspect.a003616, PMID 20463000, PMC 3140690 (freier Volltext) (Review).
- Jeremy Cherfas: Hayflick Licked: Telomerase Lengthens Life of Normal Human Cells (Memento vom 8. August 2012 im Internet Archive)
- Witzany G (August 2008). The Viral Origins of Telomeres and Telomerases and their Important Role in Eukaryogenesis and Genome Maintenance. (PDF; 263 kB). In: Biosemiotics 1 (2): 191–206. doi:10.1007/s12304-008-9018-0
- M. J. Wacker, M. P. Godard: Analysis of one-step and two-step real-time RT-PCR using SuperScript III. In: Journal of biomolecular techniques : JBT. Band 16, Nummer 3, September 2005, S. 266–271, PMID 16461951, PMC 2291734 (freier Volltext).
- A. Baranauskas, S. Paliksa, G. Alzbutas, M. Vaitkevicius, J. Lubiene, V. Letukiene, S. Burinskas, G. Sasnauskas, R. Skirgaila: Generation and characterization of new highly thermostable and processive M-MuLV reverse transcriptase variants. In: Protein engineering, design & selection : PEDS. Band 25, Nummer 10, Oktober 2012, S. 657–668, doi:10.1093/protein/gzs034, PMID 22691702.
- S. Mohr, E. Ghanem, W. Smith, D. Sheeter, Y. Qin, O. King, D. Polioudakis, V. R. Iyer, S. Hunicke-Smith, S. Swamy, S. Kuersten, A. M. Lambowitz: Thermostable group II intron reverse transcriptase fusion proteins and their use in cDNA synthesis and next-generation RNA sequencing. In: RNA. Band 19, Nummer 7, Juli 2013, S. 958–970, doi:10.1261/rna.039743.113, PMID 23697550, PMC 3683930 (freier Volltext).
Weblinks
- Die Reverse Transkriptase von HIV
- Interaktive Präsentation der RT von HIV im Online Macromolecular Museum (engl., die Darstellung der PDB-Datei erfolgt über einen JavaScript-Viewer)