Bacilladnaviridae
Bacilladnaviridae ist die Bezeichnung für eine Familie von Viren mit DNA-Genom im Phylum Cressdnaviricota (en. circular REP-encoding single-stranded DNA viruses, informell CRESS-DNA-Viren). Ihre natürlichen Wirte sind hauptsächlich Kieselalgen (Diatomeen alias Bacillariophyta) wie Chaetoceros. Sie haben ein unsegmentiertes, zirkuläres Genom, das zu einem kleinen Teil Doppelstrang-DNA, ansonsten aber Einzelstrang-DNA ist.[4][5]
| Bacilladnaviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|

Virion der Familie Bacilladnaviridae | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Bacilladnaviridae | ||||||||||||||
| Links | ||||||||||||||
|
Die Bezeichnung Bacilladnaviridae ist zusammengesetzt aus der Bezeichnung Bacillariophyta für die Wirte, DNA, und der Endung -viridae für Virusfamilien.[6]
Beschreibung
.png.webp)
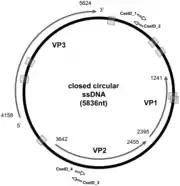
Die Virionen haben einen Durchmesser von etwa 34 nm – bei Chaetoceros setoensis DNA virus (CsetDNAV, Spezies Chaetoceros diatodnavirus 1) beispielsweise 33 nm[7] – und sammeln sich im Zellkern an.
Das Genom dieser Viren scheint einzigartig zu sein. Es besteht aus einem einzigen Molekül kovalent geschlossener zirkulärer einzelsträngiger DNA von 4,5–6 kb (Kilobasen) sowie (normalerweise) einem Segment (Fragment) bezeichnet linearer ssDNA von (insgesamt) ca. 0,6[7] bis 1 kb. In der Baltimore-Klassifikation weist sie dies als Mitglieder der Gruppe 2 (ssDNA-Viren) aus. Die linearen Fragmente sind aber komplementär zu einem Teil des geschlossenen Rings, wodurch in diesem teilweise doppelsträngige Regionen (dsDNA) entstehen, eine Konfiguration, die nach Baltimore nicht klassifizierbar wäre. Ausnahmen von der üblichen Zahl von einem komplementären Fragment sind beispielsweise:
● Bei Chaetoceros setoensis DNA virus (CsetDNAV) hat die zirkuläre DNA eine Länge von 5836 nt (Nukleotiden der Basen), und es gibt sogar acht kurze Segmente linearer komplementärer ssDNA mit jeweils nur 67 bis 145 nt.
● Bei dem als Mitglied vorgeschlagenen Spezies „Chaetoceros debilis DNA virus“ (CdebDNAV) gibt es offenbar keine solchen linearen Fragmente.[7]
Es gibt mindestens drei große Offene Leserahmen (englisch open reading frames, ORFs) in diesem Genom: VP1, VP2, VP3 (der Größe nach aufsteigend). D. h. es kodiert für (mindestens) drei Proteine.[6]
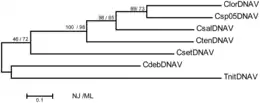
Wahrscheinlich replizieren Bacilladnaviridae ähnlich wie andere eukaryotische ssDNA-Viren ihr Genom per Rolling-Circle-Mechanismus, der durch die im Virusgenom kodierte Endonuklease Rep (kodiert durch VP3)[1] initiiert wird. Dieses Protein weist bei den Bacilladnaviridae jedoch unter den konservierten Sequenzmotiven für diese spezifische Besonderheiten auf und bildet in phylogenetischen Bäumen eine monophyletische Klade, die von anderen Gruppen von ssDNA-Viren getrennt ist.
Das Kapsidprotein CP (kodiert durch VP2)[1] hat eine Jelly-Roll-Faltung (englisch single jelly roll fold, [en]) und ist am engsten mit den entsprechenden Proteinen von Mitgliedern der Familie Nodaviridae [en] verwandt, die allerdings ein Einzelstrang-RNA-Genom besitzen. Offenbar ist es im Laufe der Evolution der ssDNA-Bacilladnaviridae zu einer Übernahme des Kapsid-Gens von den ssRNA-Nodaviridae gekommen.[1]
Reife Virionen werden an Ende durch Lyse der Wirtszelle freigesetzt.[6]
Systematik
Die Systematik der Familie Bacilladnaviridae gestaltet sich nach ICTV – ergänzt um einige Vorschläge in doppelten Anführungszeichen (nach NCBI wo nicht anders vermerkt) – ist mit Stand 4. April 2021 wie folgt:
- Klasse: Arfiviricetes, Ordnung: Baphyvirales, Familie: Bacilladnaviridae
- Gattung: Diatodnavirus,
- Spezies: Chaetoceros diatodnavirus 1 (Typus) – mit Chaetoceros setoense DNA virus alias Chaetoceros setoensis DNA virus (CsetDNAV)[7] – 5836:67+70+72+78+90+107+109+145 nt
- Gattung: Kieseladnavirus
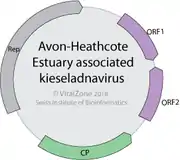
- Spezies: Avon-Heathcote Estuary associated kieseladnavirus (Typus) – mit Avon-Heathcote estuary associated bacilladnavirus (AHEaBV) – 4742 nt
- Gattung: Protobacilladnavirus (veraltet Bacilladnavirus)[9]
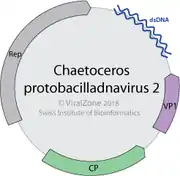
- Spezies: Chaetoceros protobacilladnavirus 1 (veraltet: Chaetoceros salsugineum DNA virus 01, Chaetoceros nuclear inclusion virus, CsalDNAV bzw. CspNIV, Typus) – 6000:997 nt[10][11][12]
- Spezies: Chaetoceros protobacilladnavirus 2 – mit Chaetoceros sp. DNA virus 7
- Spezies: Chaetoceros protobacilladnavirus 3 – mit Chaetoceros lorenzianus DNA virus (ClorDNAV)[7]
- Spezies: Chaetoceros protobacilladnavirus 4
- Spezies: Marine protobacilladnavirus 1 – mit Bacillariodnavirus LDMD-2013
- Spezies: Snail associated protobacilladnavirus 1 – mit Amphibola crenata associated bacilladnavirus 1 (AcrBV1) – 4729 nt
- Spezies: Snail associated protobacilladnavirus 2 – mit Amphibola crenata associated bacilladnavirus 2 (AcrBV2) – 4576 nt
- Spezies: „Chaetoceros sp. strain TG07-C28 DNA virus“ (Csp05DNAV)[7][13][Anm. 3] – 5785:890 nt
- Spezies: „Chaetoceros tenuissimus DNA virus“ (CtenDNAV) – 5639:875 nt (Typ 1), 5570:669 nt (Typ 2)
- Spezies: „Chaetoceros debilis DNA virus“ (CdebDNAV)[7] – ca. 7000 nt;–
- Spezies: „Thalassionema nitzschioides DNA virus“ (TnitDNAV)[7][14][Anm. 4] – 5573:600 nt
- nicht-klassifizierte Bacilladnaviridae:
Oben mit angegeben sind die Genomgößen, ggf. (soweit bekannt) auch die Fragmentgrößen nach Arsenieff (2018)[18] und Kazlauskas et al. (2017)[1]
Literatur
- Yuji Tomaru, Kensuke Toyoda, Hidekazu Suzuki, Tamotsu Nagumo, Kei Kimura, Yoshitake Takao: New single-stranded DNA virus with a unique genomic structure that infects marine diatom Chaetoceros setoensis. In: Nature Scientific Reports. Band 3, Nr. 1, 26. November 2013, ISSN 2045-2322, S. 3337, doi:10.1038/srep03337, PMID 24275766.
Anmerkungen
- Weiße Pfeile mit schwarzem Rand zeigen PCR-Primer an. Graue Pfeile zeigen die Positionen der ORFs VP1, VP2 und VP3 an. Graue Kästen zeigen die Positionen der acht kleinen, komplementären ssDNA-Fragmente an.
- Akronyme: Chaetoceros debilis DNA virus (CdebDNAV), C. lorenzianus DNA virus (ClorDNAV), C. salsugineum DNA virus (CsalDNAV), C. setoensis DNA virus (CsetDNAV), C. tenuissimus DNA virus (CtenDNAV), Chaetoceros sp. strain TG07-C28 DNA virus (Csp05DNAV), Thalassionema nitzschioides DNA virus (TnitDNAV).
- Provisorische Zuordnung nächst ClorDNAV nach Tomaru et al. (2013) Fig. 6 (siehe Abb.)
- Provisorische Zuordnung nächst CdebDNAV nach Tomaru et al. (2013) Fig. 6 (siehe Abb.)
Einzelnachweise
- Darius Kazlauskas, Anisha Dayaram, Simona Kraberger, Sharyn Goldstien, Arvind Varsani, Mart Krupovic: Evolutionary history of ssDNA bacilladnaviruses features horizontal acquisition of the capsid gene from ssRNA nodaviruses. In: Virology. 504, 2017, S. 114–121. doi:10.1016/j.virol.2017.02.001. PMID 28189969.
- Krupovic et :al.: Proposal 2019.012D (accepted by ICTV)
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- Virus Taxonomy: 2019 Release. In: talk.ictvonline.org. International Committee on Taxonomy of Viruses. Abgerufen am 25. April 2020.
- Y. Tomaru, Y. Takao, H. Suzuki, T. Nagumo, K. Koike, K. Nagasaki: Isolation and Characterization of a Single-Stranded DNA Virus Infecting Chaetoceros lorenzianus Grunow. In: Applied and Environmental Microbiology. 77, Nr. 15, 2011, S. 5285–5293. doi:10.1128/AEM.00202-11. PMID 21666026. PMC 3147440 (freier Volltext).
- SIB: Bacilladnaviridae, auf: Expasy ViralZone
- Yuji Tomaru, Kensuke Toyoda, Hidekazu Suzuki, Tamotsu Nagumo, Kei Kimura, Yoshitake Takao: New single-stranded DNA virus with a unique genomic structure that infects marine diatom Chaetoceros setoensis. In: Nature Scientific Reports. Band 3, Nr. 1, 26. November 2013, ISSN 2045-2322, S. 3337, doi:10.1038/srep03337, PMID 24275766.
- SIB: Kieseladnavirus: Gemome: Avon-Heathcote Estuary associated kieseladnavirus, auf: Expasy ViralZone
- ICTV: ICTV Taxonomy history: Protobacilladnavirus
- NCBI: Chaetoceros salsugineum DNA virus, equivalent: Chaetoceros salsugineum nuclear inclusion virus (species)
- Y. Bettarel, J. Kan, K. Wang, Kurt E. Williamson, S. Cooney, S. Ribblett, F. Chen, K. Wommack, D. Coats: Isolation and preliminary characterisation of a small nuclear inclusion virus infecting the diatom Chaetoceros cf. gracilis, auf: Semantic Scholar – Biology – Aquatic Microbial Ecology, Corpus ID: 56157535, 2005, doi:10.3354/AME040103. „Chaetoceros nuclear inclusion virus: CspNIV“, „CspNIV shows some strong similarities with Heterosigma akashiwo nuclear inclusion virus (HaNIV)“
- Keizo Nagasaki: Dinoflagellates, diatoms, and their viruses, in: The Journal of Microbiology 46(3), S. 235–243, Juli 2008, doi:10.1007/s12275-008-0098-y. „CspNIV shows some strong similarities with Heterosigma akashiwo nuclear inclusion virus (HaNIV)“
- NCBI: Chaetoceros virus YT-2008 (species) – GenBank: AB647334.1
- NCBI: Thalassionema nitzschioides DNA virus (species)
- NCBI: Haslea ostrearia associated bacilladnavirus (species)
- Janice E. Lawrence, Amy M. Chan, Curtis A. Suttle: A novel virus (HaNIV) causes lysis of the toxic bloom-forming alga Heterosigma akashiwo (Raphidophyceae). In: Journal of Phycology. 37, Nr. 2, 1. Mai 2002, S. 216–222. doi:10.1046/j.1529-8817.2001.037002216.x.
- Stephanie Boone: Viruses and Algae, Februar 2004
- Laure Arsenieff: Parasitisme et contrôle des blooms de diatomées en Manche occidentale, Dissertation, Station Biologique de Roscoff, Biodiversité et Ecologie, Sorbonne Université, Frankreich, 2018 (HAL Id: tel-02864776) Epub 11. Juni 2020 (französisch)