Replikation
Replikation oder Reduplikation bezeichnet in der Biologie die Vervielfältigung der Nukleinsäuremoleküle als Träger der Erbinformation einer Zelle oder eines Virus, sei es des gesamten Genoms aus DNA bzw. RNA oder auch nur einzelner Chromosomen oder Segmente. Die Reduplikation genannte Verdopplung der DNA in der Synthese-Phase des Zellzyklus geht einer Mitose voraus und betrifft gewöhnlich den gesamten Chromosomensatz. Doch können in Zellzyklen mancher spezialisierter somatischer Zellen bei Eukaryoten auch Teile ihres Genoms unterschiedlich behandelt werden: bestimmte DNA-Sequenzen werden amplifiziert; andere DNAs werden nicht multipliziert und bleiben unterrepliziert in nachfolgenden Zyklen. In solchen Fällen der Differenzierung auf DNA-Ebene wird der allgemeinere Ausdruck Replikation für die zelluläre DNA-Synthese bevorzugt.
Der molekulare Mechanismus der Re(du)plikation doppelsträngiger DNA ist stets semikonservativ (von lateinisch semi ‚halb‘ und conservare ‚erhalten‘).[1] Die DNA-Doppelhelix wird enzymatisch in ihre beiden Stränge aufgetrennt; dann katalysieren DNA-Polymerasen an jedem Einzelstrang die komplementäre Ergänzung zu einer halbkonservativen (= halbneuen) DNA-Doppelhelix. „Komplementär“ bedeutet, dass der vereinzelte, konservative DNA-Strang die Basen-Abfolge (Sequenz) seines künftig gegenüberliegenden Stranges eindeutig bestimmt. Jede Base eines DNA-Nukleotids kann nach den Regeln der Basenpaarung nur mit einem festgelegten Partner über Wasserstoffbrücken stabil binden (Adenin ↔ Thymin; Guanin ↔ Cytosin).
Bei RNA-Viren[2] und Retroviren ist deren RNA in die Definition der Replikation eingeschlossen. Alle Viren verwenden zum Replizieren, da sie keinen eigenen Stoffwechsel haben, die notwendigen Ausgangsstoffe der Wirtszelle, teilweise auch deren Enzyme.[3]
Zusammenfassung des Replikationsvorgangs
Replikation ist die Vervielfältigung von Nukleinsäuremolekülen. In Zellen sind die in Form einer Doppelhelix als DNA-Doppelstrang vorliegenden Nukleinsäuremoleküle die Träger der Erbinformation. Sie liegen bei (eukaryonten) Zellen mit Zellkern im Kern (Nukleus) und werden hier für eine Kernteilung vor einer Zellteilung verdoppelt. Bei der Re(du)plikation entstehen aus einem jeweils zwei gleiche DNA-Doppelstrangmoleküle. Damit kann den beiden Kernen zweier Tochterzellen je die gleiche Erbinformation zugeteilt werden.
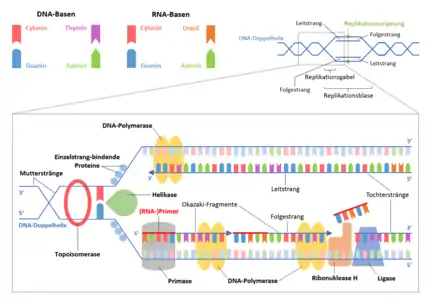
Der Vorgang der Replikation besteht molekularbiologisch aus einer Reihe von Schritten, die mittels verschiedener Enzyme gesteuert werden. Den Aufbau neuer DNA-Stränge bewirkt ein DNA-Polymerase genanntes Enzym, das hierfür einen Einzelstrang als Matrize benötigt. Denn erst anhand dieser Vorlage kann ein komplementärer DNA-Strang synthetisiert werden, indem jeweils das passende Nukleotid mit dem vorigen verknüpft wird. Zunächst muss daher am Ort des Replikationsursprungs (Origin) durch das Enzym Topoisomerase die Drehung der Helix entwunden werden. Dann kann der Doppelstrang durch das Enzym Helikase in diesem Bereich aufgetrennt werden – unter Lösen der Wasserstoffbrückenbindungen zwischen den Basenpaaren – in zwei einzelsträngige Abschnitte. Mit deren Aufspreizen wird eine sogenannte Replikationsgabel gebildet, die von einzelstrangbindenden Proteinen (SSB-Proteine) stabil gehalten wird. Nun kann eine Primase den beiden vorliegenden Einzelsträngen jeweils kurze RNA-Abschnitte anlagern, sogenannte Primer, mit denen die eigentliche Strangsynthese initiiert wird. Daran nämlich kann die DNA-Polymerase dann ansetzen und – den vorliegenden Einzelstrang als Matrize nutzend mittels Basenpaarung – fortlaufend passende Desoxyribonukleotide aneinanderknüpfen zum neuen komplementären Strang. Die DNA-Polymerase knüpft einen neuen Baustein des Polynukleotids an dessen 3′-Ende an, synthetisiert den neuen Strang also stets in 5′→3′-Richtung komplementär, während sie sich dementsprechend am antiparallelen Matrizenstrang dabei in 3′→5′-Richtung entlang bewegt.
Da die beiden jeweils als Matrize dienenden Einzelstränge des früheren Doppelstrangs auch einander komplementär sind und antiparallel zueinander verlaufen, bewegt sich eine synthetisierende DNA-Polymerase auf dem einen Matrizenstrang in Richtung der wandernden Replikationsgabel, und auf dem anderen in Gegenrichtung. An dem einen Matrizenstrang kann so der sogenannte Leitstrang (englisch leading strand) kontinuierlich synthetisiert werden. An dem anderen Matrizenstrang dagegen verläuft der Replikationsvorgang diskontinuierlich. Der gegenläufige neue Strang, Folgestrang (englisch lagging strand) genannt, muss stückweise von einer DNA-Polymerase synthetisiert werden. Dabei entstehen die sogenannten Okazaki-Fragmente. Deren RNA-Primer werden alsdann von einer anderen DNA-Polymerase durch DNA ersetzt. Anschließend können die Fragmente von einer DNA-Ligase zu einem Gesamtstrang verknüpft werden. Das Ergebnis der Replikation sind schließlich zwei (nahezu) identische DNA-Doppelstränge, bei denen jeweils eine Hälfte neu ist. Die Replikation erfolgt somit semi-konservativ, denn ein jeder Doppelstrang setzt sich aus einem vorbestehenden und einem neusynthetisierten Einzelstrang zusammen.
Semikonservatives Prinzip
Schon Watson und Crick erkannten, dass die im Doppelstrang gepaarten Basen eine Voraussetzung für die Bildung neuer DNA sind: „Es ist unserer Aufmerksamkeit nicht entgangen, dass die spezifische Paarbildung, die wir hier voraussetzen, sogleich an einen möglichen Kopiermechanismus für das genetische Material denken lässt.“[4][5] Drei Möglichkeiten der Replikation waren denkbar: dispersiv oder total konservativ oder halb konservativ. Letzteres Modell wurde durch den Meselson-Stahl-Versuch belegt.[6][7] Demnach wird der Original-Doppelstrang geöffnet, anschließend dienen beide Einzelstränge in der Replikation als Vorlage (als konservative Matrize). Der neue Strang entsteht nach den Regeln der Watson-Crick-Basenpaarung. Die Replikation nach dem semikonservativen Prinzip stellt den allgemein angenommenen Mechanismus dar. Alle anderen Prinzipien sind Sonderfälle, die jeweils nur zum Teil bewiesen sind.
Da alle Bakterien sowie die Zellkerne aller Eukaryoten doppelsträngige DNA (dsDNA) enthalten, kommt dieser Replikationsmechanismus in der Natur am häufigsten vor. Ausnahmen bilden einige Mitochondrien, bei denen ein anderer Mechanismus abläuft, sowie Plasmid- und Virengenome, bei denen die Erbinformation als DNA-Einzelstrang (ssDNA) vorliegen kann. Hier musste ein gänzlich anderer Mechanismus gefunden werden: der Rolling circle. Bei Retroviren, deren Erbinformation stets in Form eines RNA-Doppel- oder -Einzelstrangs vorliegt, wird die Replikation von der Wirtszelle übernommen, indem die RNA durch eine Reverse Transkriptase in DNA umgeschrieben und in das Wirtsgenom eingebaut wird.
Das Replikon
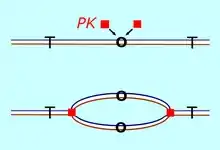
Unten: An den Wachstumsgabeln symmetrische DNA-Replikation; mögliche Endpunkte (Termini, T)
Das Replikon ist die elementare Einheit der Replikation.[8] Prokaryoten und Plasmide haben meist nur ein Replikon; dieses reicht aus, um die kleinen Genome in kurzer Zeit zu multiplizieren. Ausnahmen sind einige Bakterien und Archaeen. Die großen Genome der Eukaryoten sind in mehrere beziehungsweise viele Replikationsabschnitte gegliedert. Ein Replikon wird in der Regel symmetrisch aktiv, und zwar in der S-Phase, einem Zeitfenster des Zellzyklus für die DNA-Synthese.
Mitten im Replikon sitzt der Origin, der Ursprung der Replikation. Sobald der Origin aktiviert ist, wandert je eine Replikationsgabel (Wachstumsgabel) in die eine, die andere Gabel in die entgegengesetzte Richtung. Somit entspricht einer geöffneten Gabel ihre „Zwillings-Gabel“; beide entfernen sich (idealerweise) mit gleicher Geschwindigkeit vom gemeinsamen Origin. Da DNA-Polymerasen 5' → 3' replizieren, steht auf der einen Seite des Origins zuerst die HO-Matrize als Leitstrang zur Verfügung. Auf der anderen Seite des Origins wird unausweichlich der andere Strang zum Leitstrang. Das Replikon ist in beiden Richtungen vollständig repliziert, sobald jede der beiden Gabeln auf die Replikationsgabel ihres benachbarten Replikons trifft. Ein Replikon-Ende oder Terminus besitzt in der Regel keine definierte Sequenz, fordert lediglich für das Replikon eine endliche Länge.
Origin
Eukaryoten haben sehr aktive und weniger aktive Replikations-Startstellen. Dazu gibt es schlafende Origins, die für Notfälle vorgesehen sind.[9] Die Zahl der Replikations-Startpunkte entspricht der Größe des jeweiligen Genoms. Darmbakterium Escherichia coli: 1 Origin. Bäckerhefe Saccharomyces cerevisiae: ~350 Origins. Mensch Homo sapiens: 40000 bis 80000 Origins.[10][11]
Replikationsgabel
Mit Pulsen radioaktiver Markierung gelang es bei Zellen des Chinesischen Hamsters und bei HeLa-Zellen, die DNA-Synthese im Lichtmikroskop anschaulich zu machen. Die chromosomale DNA besteht aus Tandem-Abschnitten (Replikons), in denen die (Mutter-)DNA in gabelartigen Wachstumspunkten repliziert. Der Fortschritt einer Gabel wurde auf höchstens 2,5 Mikrometer in der Minute geschätzt. Wichtig ist auch, dass benachbarte Gabeln gleichzeitig aktiv werden.[12] Damit ist jedoch nicht ausgeschlossen, dass sich die Replikationzeit des Euchromatins von der des Heterochromatins unterscheidet.
Molekulargenetische Untersuchungen ergaben Modelle für die submikroskopischen Details der Gabel:
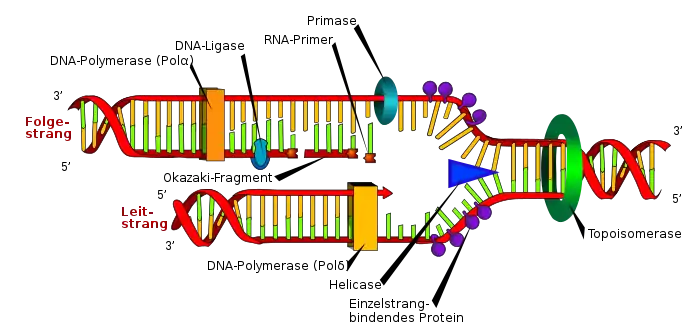
Das Schema zeigt an einer vom Origin aus nach rechts wandernden Replikationsgabel die beteiligten Enzyme bzw. Moleküle in Eukaryoten. Vorneweg arbeitet eine Topoisomerase; sie entwindet das DNA-Molekül aus komplexen Strukturen des Interphase-Chromatins. Eine Helikase trennt die beiden antipolaren Stränge der Doppelhelix. Proteingruppen stabilisieren die Einzelstränge, damit sich diese nicht sofort wieder komplementär paaren. Eine DNA-Polymerase (Pol δ) ergänzt seit ihrem Start am Origin kontinuierlich 3' → 5' den Leitstrang zur halb-konservativen, halb-neuen Doppelhelix.
Am Folgestrang geschieht die Replikation ebenfalls 3' → 5', allerdings diskontinuierlich. Zuerst katalysiert eine RNA/DNA-Primase (Pol α) einen RNA-Primer, der zu einem kurzen DNA-Stück, einem Okazaki-Fragment verlängert wird. Eine Pol δ übernimmt für eine kurze Strecke die Replikation des Folgestranges zur Doppelhelix. Dann katalysiert eine Pol α den nächsten Primer, das nächste Okazaki-Fragment folgt, und so weiter. Eine DNA-Ligase füllt mit Desoxynukleotiden die Lücken, die eine RNase H (nicht im Schema gezeigt) beim Abbau des RNA-Primers hinterließ.
Der koordinierte, gleichzeitige Fortschritt der Folgestrang-Replikation mit der des Leitstranges ist dem beschriebenen Gabelschema nicht anzusehen. Damit die Primase/Pol α am Folgestrang Gelegenheit bekommt, einen RNA-Primer zu katalysieren und die ersten Desoxynukleotiden anzuhängen, bildet der Folgestrang eine Schlaufe. In dieser ersetzt ein Molekül Pol δ die Primase/Pol α und verlängert das anfängliche RNA-DNA-Hybrid zu einem Okazaki-Fragment. Mit Hilfe der Schlaufe gelangt die Folgestrang-Pol δ gleichgerichtet an die kontinuierlich aktive Pol δ des Leitstranges. Beide Polymerasen verbinden sich und bewerkstelligen die Replikation synchron.[13] Dieses Modell erläutert die Bedeutung zweier weiterer Moleküle. Damit die Pol δ an die Matrize bindet, braucht es den Replikationsfaktor C (RFC), der auch das PCNA (das nukleäre Antigen proliferierender Zellen) auflädt. Die Klammer des PCNA verhindert, dass Pol δ von der Matrize abfällt.[14]
Da die aus demselben Origin entgegengesetzt laufende Replikationsgabel gleichfalls eine Schlaufe für ihren Folgestrang bildet, ist das Replikationsauge eine spannungsfreie, symmetrische Figur.
Terminus
Bei den Eukaryoten gibt es innerhalb der Chromosomen keine definierte Sequenz für die Beendigung der Replikation; der Terminus stellt lediglich eine molekulare morphologische Beschreibung dar. Dennoch: Keine Regel ohne Ausnahme. Es gibt sequenzspezifische Barrieren, die eine Replikationsgabel aufhalten, bevor die gegenläufige Gabel ankommt. Am besten untersucht sind die polaren Replikationsbarrieren, welche die ribosomale DNA begrenzen. Diese Termini sind die Voraussetzung für die lokale Amplifikation der rDNAs. Dann ist die spezifische Termination vor den Telomeren zu nennen; die Replikation stoppt, wenn die Gabel das Chromosomenende erreicht.[15][16]
Ablauf der Replikation
- Initiation stößt die Replikation an. Hier wird die DNA-Doppelhelix an einer bestimmten Stelle mit Hilfe der Helikase aufgebrochen und eine Polymerase lagert sich nach der Markierung durch eine Primase an die aufgebrochene DNA.[1]
- Elongation, in der die eigentliche Vervielfältigung vonstatten geht. Die beiden Stränge werden zeitgleich komplementär synthetisiert.
- Termination schließt die DNA-Synthese ab.
- DNA-Reparatur zur Korrektur eventuell entstandener Kopierfehler.[17]
Initiation
In der Zelle liegt die helikale DNA nicht geordnet ringförmig oder linear vor, sondern ist zusätzlich in sich verdrillt zu so genannten ‚supercoils‘: Während sich die DNA-Doppelhelix bei Eukaryoten um basische DNA-Strukturproteine wie Histone wickelt und der gesamte Komplex aus DNA-Molekül und zugehörigen Strukturproteinen zusätzlich durch Faltungen und Verdrehungen komprimiert und dadurch auch stabilisiert wird, ist das bei Prokaryoten (sowie Organellen mit eigener DNA wie z. B. Mitochondrien und Chloroplasten) nicht in gleicher Weise der Fall, da sie keine Histone besitzen. Man hat aber ‚histonähnliche Proteine‘ (englisch histone-like protein, HLP) gefunden, die für einen ähnlichen Effekt sorgen.[18][19][20] Um repliziert werden zu können, muss die DNA entwunden werden. Die Folge der Entwindung der DNA an einer Stelle ist die zunehmende Verdrillung des gesamten DNA-Doppelstranges. Um Torsionsspannungen bei der Entwindung entgegenzuwirken, läuft vor jeder Replikationsgabel eine Topoisomerase, die die Verdrillung vermindern kann (Entspannungsreaktion). Dazu ist die Spaltung der DNA-Stränge notwendig. Je nach Enzymtyp (Topoisomerase I oder II (II ist Gyrase in E.coli)) werden kontrolliert Einzel- oder Doppelstrangbrüche durchgeführt. Nach der Entwindung werden die zuvor gespaltenen Phosphorsäureesterbindungen des Zucker-Phosphat-Gerüsts der DNA durch das Enzym wieder geknüpft.
Für die Initiation der Replikation ist ein spezieller Ort, der Replikationsursprung (englisch Origin) auf der meist ringförmigen DNA notwendig, der den Startpunkt bestimmt. An dieser Stelle werden die Wasserstoffbrückenbindungen zwischen den Basen der beiden Einzelstränge aufgetrennt. Der sogenannte oriC umfasst 245 Basenpaare (bp) und enthält eine Tandemanordnung mit AT-reichen Sequenzen, die folgende Consensus-Sequenz aufweisen:
- 5′-GATCTNTTNTTTT-3′
- 3′-CTAGANAANAAAA-5′
In E.coli gibt es 5 Bindungsstellen für das DnaA-Hexamer. Zunächst wird das Initiatorprotein DnaA durch Adenosintriphosphat (ATP) aktiviert und an fünf jeweils 9 bp lange DnaA-Boxen gebunden. Insgesamt lagern sich etwa 20 DnaA-Proteine als Schleife um die DNA zusammen. Die Proteine IHF und FIS binden an spezifische Abschnitte des oriC und lösen die Beugung der DNA zu einer haarnadelähnlichen Struktur aus, welche auch die Bindung von DnaA unterstützt. Schließlich kann die Entwindung der Doppelhelix an drei aufeinanderfolgenden, 13 bp langen Adenin- und Thymin-reichen Sequenzen (auch ‚13-mer-Sequenzen‘ genannt) starten.
Dieser Vorgang wird von einer Helikase unter ATP-Verbrauch katalysiert (bei E. coli heißt diese DnaB und wird durch das Protein DnaC zum Origin geleitet). Die Helikase arbeitet von 5'-3'-Richtung. Durch die Auftrennung des Doppelstranges entstehen so am Origin zwei Replikationsgabeln, die während der Replikation bidirektional auseinanderlaufen. Damit sich die Basen nicht wieder über Wasserstoffbrückenbindungen paaren, halten sogenannte Einzelstrang-bindende Proteine (bei den Prokaryoten heißt dies „SSB-Protein“ für englisch single-strand-binding-protein) die einzelnen Stränge auseinander.
Im Anschluss der Öffnung folgt das Priming: An den nun freien Einzelsträngen wird durch eine RNA-Polymerase, die Primase (in E. coli DnaG), ein kurzes RNA-Stück, der Primer (unter zellulären Bedingungen etwa 10 Nukleotide), gesetzt. Dieser Komplex wird als ‚Primosom‘ bezeichnet und ist notwendig, da der Hauptproteinkomplex der Replikation, die DNA-Polymerase, mit der Synthese des jeweils zweiten Stranges DNA nur an einer freien 3′-OH-Gruppe beginnen kann. Das heißt: Die DNA-Polymerase benötigt den Primer als ‚Starthilfe‘ für die Replikation, auch wenn es sich dabei um RNA handelt. Die für den Primer eingesetzte RNA-Polymerase benötigt nur den Einzelstrang als Matrize. Hat die DNA-Polymerase dann aber erst mit der Synthese des zweiten Stranges (von 5′ nach 3′) begonnen, kann sie kaum unterbrochen werden und arbeitet bis zur Termination fort. Somit muss die Regulation der Replikation in der Initiationsphase geschehen.
Elongation
Nach der Initiation und der nun beginnenden Polymerisierung wird die Elongationsphase durchlaufen. Hier synthetisiert die DNA-Polymerase III die komplementären Stränge zu den Einzelsträngen: Die Basen der Einzelstränge werden nacheinander abgelesen und, nach dem Prinzip der komplementären Basenpaarung, im synthetisierten Strang entsprechend nacheinander eingebaut. Für die DNA-Synthese notwendige Bausteine liegen in Form der freien Nukleotide in der Zelle vor.
Dabei ergibt sich jedoch ein Problem: Die DNA-Polymerase kann nur in 5′→3′-Richtung synthetisieren. Da nun aber der DNA-Polymerasekomplex an beiden Strängen gleichzeitig synthetisiert, beide Stränge aber gemäß der doppelhelikalen Struktur entgegengesetzt orientiert sind, ergeben sich zwei gegenläufige (auch antiparallele) Stränge an der Replikationsgabel. Am Leitstrang kann nach einmaligem Priming bis zur Symmetrieachse der Replikationsgabel durchgehend repliziert werden, da dieser genau mit der Leserichtung des Polymerasekomplexes und in Laufrichtung der Replikationsgabel orientiert ist. Am Folgestrang ist eine kontinuierliche Replikation hingegen nicht möglich, da er in die „falsche Richtung“ verläuft. Dabei läuft die Polymerase bei dem ersten Lauf gegen den Primer des Leitstranges der zweiten Replikationsgabel, die in die andere Richtung verläuft. Nach der Unterbrechung muss ein erneutes Priming auf dem Folgestrang erfolgen, damit die Polymerase wieder erneut ansetzen kann. Dieses Priming erfolgt immer direkt der Helikase folgend. In den folgenden unregelmäßigen Polymerasezyklen am Folgestrang beendet die Polymerase die Synthetisierung immer am letzten RNA-Primer, also am 5′-Ende des vorhergehenden Fragments. Die so entstehenden DNA-Einzelfragmente werden auch als Okazaki-Fragmente bezeichnet. Die DNA-Polymerase hat verschiedene Domänen: die eigentliche Polymerase-Domäne zum Anhängen von Nukleotiden, die DNA-Klammer, um auf der DNA zu gleiten, ohne abzusetzen und die 5'-3'-Exonuklease-Domäne, welche der Korrekturlesefunktion direkt nach dem Einbau der Basen dient.
Es gibt einige gut belegte Hinweise darauf, dass der Bereich zwischen Primase und dem letzten Okazaki-Fragment als eine Schleife (auch als "Posaunen-Modell" bezeichnet) verdreht wird, so dass die Polymerase beide Stränge mit gleicher Laufrichtung bearbeiten kann.[21] Ist die Synthese der Schleife beendet, wird diese wieder aufgelöst und eine neue Schleife gebildet. Hier scheint wieder eine Topoisomerase mitzuwirken. Da die Verdoppelung an einem Strang kontinuierlich, am anderen mit Unterbrechungen verläuft, spricht man auch von einer semidiskontinuierlichen Verdoppelung.
Damit nun ein kontinuierlicher Strang entsteht, der keine RNA-Stücke enthält, tritt noch während der Replikation ein weiterer Mechanismus in Aktion: Eine RNase H entfernt die RNA-Primer und DNA-Polymerase I füllt die entstandene Lücke mit der jeweils komplementären DNA. DNA-Polymerase I kann auch selbst RNA entfernen.[22]
Die DNA-Ligase schließt dann die Bindung vom 3′-Ende des neuen zum 5′-Ende des alten DNA-Stückes, stellt also zwischen den neu synthetisierten DNA-Strängen die Phosphodiesterbindungen her.[22]
Termination
Bei den Prokaryoten mit einer ringförmig aufgebauten DNA sind gegenüber dem Origin gelegene Terminationssequenzen gefunden worden. Dabei handelt es sich genauer um zwei Sequenzen, jeweils eine für eine Replikationsgabel. Normalerweise muss die Termination nicht besonders ausgelöst werden, da, wenn zwei Replikationsgabeln aufeinanderlaufen bzw. die DNA, wie bei einer linearen Form, endet, die Replikation damit automatisch beendet wird. Es handelt sich hierbei um ein Kontrollelement, damit die Replikation bei verschiedenen Replikationsgeschwindigkeiten beider Replikationsgabeln kontrolliert an einem bestimmten Punkt endet. Die Terminationsstellen sind Bindeorte für das Protein Tus (englisch terminus utilizing substance). Dieses blockiert die replikative Helikase (DnaB) und bringt damit die Replikation zum Stillstand.[23]
Die replizierten ringförmigen Stränge bleiben bei Prokaryoten nach der Replikation noch für eine Weile miteinander verbunden, genau an dieser terminalen Stelle, damit sie nach der Zellteilung von weiteren Prozessen abschließend getrennt und aufgeteilt werden können. Ohne diese Verbindung scheint eine Kontrolle bei der Verteilung nicht vorhanden zu sein. Die Trennung der DNA-Ringe kann über zwei Mechanismen erfolgen, wobei entweder eine TypI- oder eine TypII-Topoisomerase beteiligt ist.
Eukaryotische Replikation
Die Replikation läuft bei Eukaryoten im Wesentlichen identisch ab. Allerdings gibt es einige Ausnahmen und Sonderfälle. So muss berücksichtigt werden, dass die DNA stärker „verpackt“ ist (z. B. beim Heterochromatin), die DNA-bindenden Proteine (die Histon- und Nicht-Histon-Proteine) stärkeren Einfluss haben und die DNA in linearer Form vorliegt. Zudem handelt es sich bei den beteiligten Proteinen in der Regel um solche mit gleicher Funktionalität, aber unterschiedlichem Aufbau.
Einer der wesentlichen Unterschiede liegt in der Initiation: Bei den Eukaryoten gibt es mehrere Origins. Der Vorteil dessen ist leicht ersichtlich, da zum einen die Replikation durch die mehr vorhandenen DNA-bindenden Proteine langsamer verläuft, zum anderen die eukaryotische Polymerase (verschiedene Polymerasen, welche mit griechischen Buchstaben bezeichnet werden und sich bei der Synthese von Leit- und Folgestrang – Polα respektive Polδ – unterscheiden), die einen komplexeren Reparaturmechanismus als bei Prokaryoten besitzt (englisch proofreading), langsamer fortschreitet. Die Polymerase schafft bei Eukaryoten ca. 50 bis 100 Nukleotide pro Sekunde, während bei Prokaryoten mehr als 1000 Nukleotide pro Sekunde ergänzt werden können. Des Weiteren ist die eukaryotische DNA in der Regel wesentlich größer als die der Prokaryoten (einigen Millionen Basenpaaren bei Prokaryoten gegenüber wenigen Milliarden bei Eukaryoten). Mehrere Origins und damit mehrere Replikationseinheiten verkürzen die Zeit, die benötigt wird, das gesamte Genom zu replizieren, auch wenn die Geschwindigkeit von Prokaryoten nicht erreicht wird (Die Replikationszeit der Prokaryoten liegt im Bereich von einigen Minuten, bei Eukaryoten beträgt sie mehrere Stunden).
Die Origins der Eukaryoten haben keine spezielle Sequenz, vielmehr handelt es sich wohl dabei um eine sogenannte Consensus-Sequenz, also eine Ähnlichkeitssequenz. Diese werden auch ARS-Elemente genannt. Andere Befunde gehen davon aus, dass große Bereiche der DNA, sogenannte Replikationszentren, als mögliche Startpunkte für die Replikation dienen können. Die, wie auch immer, als Origin erkannten Stellen werden durch einen Origin-Erkennungskomplex (englisch origin recognition complex, ORC), einen Cdc6-Protein und sogenannte MCM-Proteine (englisch minichromosome maintenance protein), welche als Helikasen dienen, markiert. Diese später wieder entfernten Proteine bilden quasi die Vorhut zur Replikation. Der ORC bindet an den Origin, weiterhin rekrutiert ORC andere Faktoren (Cdc6, Cdt1 und ‚Helicase Loading Proteins‘), daraufhin binden MCM-Helikasen, welche die DNA aufschmelzen. Nur einmal während der S-Phase wird die Replikation initiiert (trotz der 10.000 Origins auf dem eukaryotischen Genom). Cycline geben im Zellzyklus das Signal zur Replikation.
Der weitere Initiationsverlauf sowie die Elongation ist mit dem der Prokaryoten funktional identisch.
Terminale Sequenzen wurden bisher bei den Eukaryoten nicht entdeckt. Sie scheinen auch keine Bedeutung zu haben, da der Replikationsapparat automatisch beendet wird, sobald das Ende der DNA erreicht wird. Hierbei ergibt sich aber, im Gegensatz zur ringförmigen DNA-Struktur der Prokaryoten, ein Problem: Die DNA-Polymerase synthetisiert am Mutterstrang jeweils von 3′ nach 5′. (Der Tochterstrang hat also die Ausrichtung 5′→3′) Die Polymerase benötigt aber eine Primase als Starthilfe, um DNA duplizieren zu können. Die Primase ist ein Enzym, welches eine kurze Startsequenz der DNA als RNA repliziert. Dieses Anfangsstück präsentiert der DNA-Polymerase ein Nukleotid mit einem freien 3′-OH-Ende, an welches es weiter DNA-Nukleotide synthetisieren kann. Nach erfolgreicher Synthese werden die RNA-Primer von Enzymen (Flap-Endonukleasen) zerstört und hinterlassen so Lücken. An den Telomeren, den Enden der Chromosomen, können diese Lücken aber nicht geschlossen werden, da kein vorhergehendes 3′-Ende vorhanden ist. Ganz ähnlich entstehen die Okazaki-Fragmente bei der Synthese des Verzögerungsstrangs. Hierbei muss die Polymerase nämlich in die verkehrte Richtung arbeiten, weshalb viele Primasen verwendet werden müssen. Die Primase liegt bei Eukaryoten im Gegensatz zu Prokaryoten nicht als eigenes Enzym, sondern gebunden an die DNA-Polymerase alpha (Polα) vor. Die dabei entstehenden Lücken werden aber von Polδ gefüllt und mit DNA-Ligasen verbunden. Dies ist möglich, da hier immer ein vorhergehendes Nukleotid mit einem 3′-Ende vorhanden ist. Da an den Telomeren keine solchen Nukleotide vorhanden sind, ist die vollständige Synthese der Enden nicht möglich. So verkürzen sich bei jeder Chromosomenverdopplung die Telomere am 5′-Ende beider Tochterstränge. Da Telomere aus einer tandemartig-repetitiven Sequenz, also einer hintereinander sich wiederholenden Sequenz aufgebaut sind, die keine Strukturgene beinhaltet, ist ein Verlust bis zu einer gewissen Länge nicht von großem Nachteil. Man vermutet aber, dass die DNA mit zunehmender Replikationsanzahl instabiler wird, da die stabilisierende Wirkung der Telomere immer schwächer wird. Eventuell könnte dies ein genetisches Indiz für das Altern sein.
Außer bei Einzellern wie dem Wimpertierchen Tetrahymena[24] hat man bei Vielzellern in den Keimbahnzellen und Stammzellen sowie im Knochenmark (Blutbildung) und auch bei einigen Tumorzellen ein Enzym namens Telomerase entdeckt, welches diesen Verlust kompensiert. Dies ist eine Reverse Transkriptase (RNA-abhängige DNA-Polymerase), denn in ihr liegt die repetitive Sequenz als Matrize in RNA-Form vor. Sie verlängert den Leitstrang um einige Sequenzen, so dass die DNA-Polymerase nach erfolgtem Priming den Folgestrang synthetisieren kann.
Während der S-Phase des Zellzyklus bindet das Protein Cohesin die beiden Schwesterchromatiden der gesamten Länge nach aneinander. Während der Anaphase löst das Enzym Separase das Cohesin wieder auf, sodass die Schwesterchromatiden von den Spindelfasern zu den Zellpolen gezogen werden können.
Die Replikation wird in einem Abschnitt des Zellzyklus angestoßen: bei den Eukaryoten in der S-Phase (DNA-Synthesephase), die selbst zur Interphase gehört.
Reduplikation in Keimbahn und Stammzellen
Damit der genetische Bauplan einer biologischen Art und das Programm für seine zeitgerechte Verwirklichung erhalten bleibt, erfüllen die Zellen der Keimbahn eine Notwendigkeit: Sie müssen ihre Erbinformation vor jeder Kern- und Zell-Teilung exakt verdoppeln.[25] Die Chromosomen verwirklichen dieses Erfordernis, was an ihren Enden, den Telomeren, einer besonderen Lösung bedarf. Der strengen Regel für den mitotischen Zellzyklus gehorchen auch (pluripotente) Stammzellen.[26][27] An die identische, vollständige, semikonservative Replikation ist die Zellteilung stark gekoppelt.
Replikation in somatischen Zellkernen
- Endoreplikation: Im Soma vieler Organismen kommt es zu mehreren abfolgenden Zellzyklen mit genetischer Multiplikation, nach denen sich die vergrößerten Zellkerne, folglich auch die betreffenden Zellen, nicht teilen. Diese Art wiederholter DNA-Synthesen heißt Endoreplikation. Dabei mag es sich oft um vollständige Reduplikation[28] handeln; doch nicht selten ist die Endoreplikation ein selektiver Prozess, und zwar:
- Bei der Amplifikation werden bestimmte DNA-Sequenzen gegenüber dem restlichen Genom übermäßig repliziert.[29][30] Die Chorion-Gene bei Drosophila melanogaster amplifizieren in den Follikelzellen sechzigfach, bevor ihre Transkription beginnt. Solche Überreplikation garantiert eine große Menge an mRNA für die Hülle der Fliegeneier.[31][32]
- Bei der Unterreplikation dagegen werden bestimmte Sequenzen von den Verdoppelungsrunden ausgeschlossen. Das genetische Ergebnis ist durchaus mit dem der Elimination zu vergleichen.[33][34][35] In polytänen Chromosomen von D. melanogaster bleiben einzelne Replikationsgabeln in 20 % ihrer Banden stecken: Die Replikation wird stellenweise nicht beendet. Das ist allerdings eine unschädliche Art von Genom-Instabilität, da sie sich in somatischen Zellen, nämlich in den Speicheldrüsen, ereignet.[36]
- Diminution, Elimination: Verschiedene Eukaryoten verzichten ab einem gewissen embryonalen Entwicklungspunkt reguliert auf einen beträchtlichen Teil ihrer DNA. Besonders dramatisch sehen unter dem Lichtmikroskop jene Fälle aus, die Chromosomen ganz oder teilweise aus somatischen Zellkernen entfernen.[37][38] Die Fachbezeichnungen dafür sind Chromatin-Diminution oder Chromosomen-Elimination.[39][40][41][42][43]
Strukturelle Mechanismen
Die Replikation kann mit verschiedenen molekularen Mechanismen ablaufen, die von der Primärstruktur der Nukleinsäure abhängen. Neben dem symmetrischen Prozess der bidirektionalen Replikation, welcher bisher beschrieben wurde, gibt es asymmetrische Prozesse, nämlich: Telomer-Reproduktion, D-Loop-Prozess und Rolling-Circle-Prinzip.
Bidirektionale Replikation
Die Vervielfältigung der DNA in linearen Chromosomen des Kerngenoms einer eukaryoten Zelle sowie des Bakteriengenoms erfolgt zweiseitig symmetrisch. Die Replikation geht von vielen Startsequenzen („Origins“) aus, die über die DNA-Doppelhelix (eines Chromosoms) verteilt sind. Die Zahl der Origins entspricht der (möglichen) Anzahl der Replikationseinheiten („Replikons“). Die bidirektionale Replikation läuft von jedem Origin entgegengesetzt, in beide Richtungen, und zwar gleichzeitig an beiden Einzelsträngen der DNA-Doppelhelix. Das bidirektionale Prinzip kommt in der Natur am häufigsten vor.
Telomer-Reproduktion
Die identische Vervielfältigung der Telomere ist ein Problem linearer DNA-Moleküle bzw. ihrer Chromosomen. Die beiden Enden einer Doppelhelix erlauben keine bidirektionale Replikation, weil es am „Folgestrang“ schließlich keine Ansatzsequenz für einen RNA-Primer gibt. Deswegen bleibt am Chromosomenende der Folgestrang eine Primerlänge (20 bis 200 Nukleotide) kürzer als der „Leitstrang“. Da eine normale DNA-Replikation nicht möglich ist, setzt am Leitstrang die Telomerase an. Dieses (hybride) Enzym besteht aus einem Proteinteil und der RNA-Komponente 3'- CAACCCCAA-5' (bei Tetrahymena). Die neun RNA-Nukleotide enthalten die Matrize (fett), der entsprechend das einzelsträngige Telomer-Segment 5'-TTGGGG-3' wiederholt aneinander gereiht wird und den (ehemaligen) Leitstrang tandemartig verlängert.[44]
- Telomerase: Das Enzym wirkt an der 5'-3'-DNA-Matrize als Reverse Transkriptase, und zwar in drei Schritten: 1. Andocken mit der RNA-Sequenz an das 3'-Ende des chromosomalen DNA-Leitstranges; 2. Verlängern des 3'-Endes; 3. Vorrücken um ein Telomermotiv an das soeben gebildete 3'-Ende.[45] Das funktioniert wie der Bau einer Brücke, die mit einer selbsttragenden Konstruktion vorangetrieben wird. Zuletzt klappt die einzelsträngig verlängerte Telomer-DNA etwas um und bildet mit sich selbst außergewöhnliche GG-Basenpaare. In der entstandenen Schlaufe wird die einzelsträngige DNA von der (normalen) DNA-Polymerase zur Doppelhelix vervollständigt.[46]
Zellen mit aktiver Telomerase sind in der Lage, die Telomere vor jeder Teilung zu reproduzieren und zu bewahren. Notwendig ist dies vor allem in der Keimbahn und in Stammzellen.[47] Alternde somatische Zellen kommen dieser Anforderung nicht (mehr) nach, wenn ihre Telomere „verschleißen“.
- Telomeren-Schwund: Soweit der Sachverhalt zu messen ist, enthalten die Zellkerne nach jeder Teilung kürzere Telomere. Der Verlust an repetitiven Telomersequenzen gehört zum zellulären Alterungsprozess und fällt besonders in Zusammenhang mit vielen Krankheiten auf. Wenn die Telomersequenzen nicht mehr vollständig reproduziert werden, sind die Chromosomenenden irgendwann nicht mehr ausreichend geschützt. So kommt es zu genomischer Instabilität.[48][49]
- Telomer-Transposition: Die Entdeckung überraschte, dass ein Modellorganismus über keine Telomerase verfügt. Die Taufliegen der Art Drosophila vervollständigen die chromosomalen Telomere durch Transposition.[50] Während die Drosophila-Arten das Telomerase-Gen verloren haben, ist es bei anderen Organismen mit Telomer-Transposition noch vorhanden; dazu gehören der Seidenspinner Bombyx mori und der Rotbraune Reismehlkäfer Tribolium castaneum.[51]
D-Loop-Prozess
Manche Chloroplasten und Mitochondrien besitzen ringförmige DNA. Die Replikation beginnt an einem Strang in der großen, nicht-codierenden Region. Der Polymerasekomplex läuft nur in eine Richtung und produziert einen kurzen, dritten Strang, bekannt als 7S-DNA. Diese dreisträngige Struktur ist im Elektronenmikroskop als D-Loop zu erkennen. (D-Loop von engl. displacement loop: Ablöse- oder Verdrängungsschlaufe.)[52] Wenn der schon replizierte Strang mehr als zwei Drittel der ursprünglichen DNA verdrängt hat, löst er sich (von der Matrize). Der gelöste Strang wird unabhängig (zur dsDNA) repliziert.
Rolling-Circle-Replikation

Plasmide und viele zirkuläre ssDNA-Viren (die prototypischen Vertreter der Monodnaviria) zeigen ein besonderes Replikationsprinzip, die Rolling-Circle-Replikation (englisch rolling circle replication [en], RCR).[53][54][55][56] Liegt deren Nukleinsäure als Einzelstrang vor, wird sie komplementär zu einem Doppelstrang ergänzt. Ist das Erbmaterial (bereits) eine doppelsträngige Nukleinsäure, bricht eine Endonuklease einen Strang auf, indem die Verbindung zwischen zwei benachbarten Basen getrennt wird. An dieser geöffneten Stelle setzt ein Polymerasekomplex an, der nur in eine Richtung arbeitet. Das 3′-OH-Ende des geschnittenen Stranges dient dabei als Ansatzpunkt für einen sogenannten Primer, von dem aus der offene Strang verlängert wird. Für die einseitige Polymerisation dient der nicht aufgebrochene Ring als komplementäre Matrize. Die Replikationseinheit wandert um den inneren Strang herum wie um einen rollenden Kreis (Rolling circle).
Der innere Strang kann wiederholt als Matrize dienen, sodass mehrere Duplikate hintereinander entstehen, teilweise als Concatamere. Diese werden nach dem ersten Replikationsschritt zerlegt. Entweder bleiben die neuen Produkte einzelsträngig oder dienen als Matrize für einen zweiten Schritt, aus dem die replizierten doppelsträngigen Nukleinsäuren hervorgehen.
Das Rolling-circle-Prinzip scheint in der Natur in verschiedenen Formen zu existieren. Der beschriebene Ablauf stellt die verbreitetste Hypothese dar. – Erläuterung zu Viren: Diese lassen sich nach Art ihrer Nukleinsäuren in sechs Klassen einteilen. 1. Viren mit Doppelstrang-DNA; 2. mit Einzelstrang-DNA; 3. mit Doppelstrang-RNA; 4. mit positivem RNA-Einzelstrang; 5. mit negativem RNA-Einzelstrang; 6. Retroviren replizieren ihre RNA zu DNA, von der schließlich neue Virion-RNA multipliziert wird.[57]
Das Modell des rollenden Kreises kommt zum Beispiel bei der Konjugation zweier Bakterien vor. Dabei gibt ein Bakterium den Einzelstrang eines Plasmids an ein anderes Bakterium weiter, während es die eigene, ringförmige DNA des Plasmids behält. Bisher wenig erforschte ringförmige DNAs wurden extrachromosomal in menschlichen Krebszellen beobachtet.[58]
Literatur
- Rolf Knippers, Alfred Nordheim (Hrsg.): Molekulare Genetik. 10., vollst. überarb. und erw. Auflage. Thieme, Stuttgart 2015, ISBN 978-3-13-477010-0.
- Inge Kronberg: Genetik, 9 – DNA: Träger der Erbinformationen. Seiten 147–158. In: Jürgen Markl (Hsg.): Markl Biologie Oberstufe. Ernst Klett, Stuttgart, Leipzig 2010. ISBN 978-3-12-150010-9.
- Wilhelm Seyffert: Lehrbuch der Genetik. Gustav Fischer, Stuttgart etc. 1998. ISBN 3-437-25610-6. → 4. Kapitel: Weitergabe der genetischen Information. Seiten 59–80.
- Melvin L DePamphilis (Hsg.): DNA replication in eukaryotic cells. Cold Spring Harbor USA, CSH Laboratory 1996. ISBN 0-87969-459-9.
Weblinks
- David Hull und John S. Wilkins: Eintrag in Edward N. Zalta (Hrsg.): Stanford Encyclopedia of Philosophy.
- Maxanim animierte Darstellung der DNA-Replikation (engl.)
- Replikation der DNA mit Bildern und Beschreibung
- Animierte Darstellung der DNA-Replikation mit Erläuterungen
- „Forscher zeichnen zum ersten Mal auf, wie sich DNA verdoppelt – und es ist anders, als erwartet“, Galileo.tv, Juni 2016
- SIB: Viral replication/transcription/translation Viral replication/transcription/translation, §Replication, auf: Expasy ViralZone
Einzelnachweise
- R. Chaudhry, S. S. Bhimji: Biochemistry, DNA Replication. StatPearls Publishing; 2018. PMID 29489296.
- Craig E. Cameron: Viral Genome Replication. Springer Science & Business Media, 2009, ISBN 978-0-387-89456-0, S. 201.
- Tai-An Cha, Bruce M. Alberts: In vitro studies of the T4 bacteriophage DNA replication system. In: Thomas Kelly, Bruce Stillman (Hsg.): Cancer Cells. 6: Eukaryotic DNA replication. CSH Laboratory, Cold Spring Harbor 1988: 1–10. ISBN 0-87969-308-8.
- James D Watson: Die Doppel-Helix: Ein persönlicher Bericht über die Entdeckung der DNS-Struktur. Rowohlt, Reinbek bei Hamburg 1969; Seite 170. ISBN 3-499-16803-0.
- Gunther S Stent (Hsg.): James D Watson: The double helix. A personal account of the discovery of the structure of DNA. Norton & Company, New York, London. ISBN 0-393-95075-1. Seite 129: “It has not escaped our notice that the specific pairing we have postulated immediately suggests a possible copying mechanism for the genetic material.”
- Matthew Meselson, Franklin W Stahl: The replication of DNA in Escherichia coli. In: Proceedings of the National Academy of Sciences. Band 44, Nummer 7, Juli 1958, S. 671–682, doi:10.1073/pnas.44.7.671, PMID 16590258, PMC 528642 (freier Volltext).
- József Szeberényi: The meselson-stahl experiment. In: Biochemistry and Molecular Biology Education. 40, 2012, S. 209, doi:10.1002/bmb.20602. ← Verständnisfragen zum Experiment.
- François Jacob, Sydney Brenner: Sur la régulation de la synthèse du DNA chez les bacteries: l'hypothèse du replicon. In: C R Acad Sci Paris 256, 1963: 298–300.
- Marcelina W Musiałek, Dorota Rybaczek: Behavior of replication origins in Eukaryota – spatio-temporal dynamics of licensing and firing. In: Cell cycle. Band 14, Nummer 14, 2015, S. 2251–2264, doi:10.1080/15384101.2015.1056421, PMID 26030591, PMC 4614997 (freier Volltext) (Review).
- Rachel L Creager, Yulong Li, David M MacAlpine: Snapshot: Origin of DNA-replication. In: Cell 161, April 2015.
- Marie-Noëlle Prioleau, David M MacAlpine: DNA replication origins – where do we begin? In: Genes & development. Band 30, Nummer 15, 08 2016, S. 1683–1697, doi:10.1101/gad.285114.116, PMID 27542827, PMC 5002974 (freier Volltext) (Review).
- Joel A Huberman, Arthur D Riggs: On the mechanism of DNA replication in mammalian chromosomes. In: J Mol Biol 32, 2, 1968: 327–334. Abstract.
- Shou Waga, Bruce Stillman: Anatomy of a DNA replication fork revealed by reconstitution of SV40 DNA replication in vitro. In: Nature 369, 6477, 1994: 207–212. DOI: 10.1038/369207a0. → Schlaufen-Modell: S. 211, Fig. 6.
- Shou Waga, Bruce Stillman: The DNA replication fork in eukaryotic cells. In: Annu Rev Biochem 67, 1998: 721–751. PDF.
- Agnieszka Gambus: Termination of eukaryotic replication forks. In: Adv Exp Med Biol 1042, 2017: 163–187. doi: 10.1007/978-981-10-6955-0_8.
- James M Dewar, Johannes C Walter: Mechanisms of DNA replication termination. In: Nature reviews. Molecular cell biology. Band 18, Nummer 8, 08 2017, S. 507–516, doi:10.1038/nrm.2017.42, PMID 28537574, PMC 6386472 (freier Volltext) (Review).
- A Bębenek, I Ziuzia-Graczyk: Fidelity of DNA replication – a matter of proofreading. In: Current Genetics. [elektronische Veröffentlichung vor dem Druck] März 2018, doi:10.1007/s00294-018-0820-1, PMID 29500597.
- Neil A Campbell, Jane B Reece, Lisa A Urry, Michael L Cain, Steven A Wasserman, Petzer V Minorsky, Robert B Jackson: Biology. 8th Edition. Benjamin Cummings, Pearson Schweiz, Cham 2008. ISBN 978-0-321-54325-7.
- John M. Archibald: The Puzzle of Plastid Evolution. In: Current Biology. 19, Nr. 2, 2009, S. R81–8. doi:10.1016/j.cub.2008.11.067. PMID 19174147.
- T. Kobayashi, M. Takahara, S. Y. Miyagishima, H. Kuroiwa, N. Sasaki, N. Ohta, M. Matsuzaki, T. Kuroiwa: Detection and Localization of a Chloroplast-Encoded HU-Like Protein That Organizes Chloroplast Nucleoids. In: The Plant Cell Online. 14, Nr. 7, 2002, S. 1579–1589. doi:10.1105/tpc.002717. PMID 12119376. PMC 150708 (freier Volltext).
- Hans-Georg Koch, Jan Brix, Peter C. Heinrich: Replikation – Die Verdopplung der DNA. In: Löffler/Petrides Biochemie und Pathobiochemie (= Springer-Lehrbuch). Springer, Berlin, Heidelberg 2014, ISBN 978-3-642-17972-3, S. 545–558, doi:10.1007/978-3-642-17972-3_44.
- Replikation und Reparaturmechanismen der DNA - Wissen für Mediziner. Abgerufen am 18. Februar 2020.
- B A Berghuis, V S Raducanu, M M Elshenawy, (…), N H Dekker: What is all this fuss about Tus? Comparison of recent findings from biophysical and biochemical experiments. In: Crit Rev Biochem Mol Biol 53, 2018: 49–63. doi: 10.1080/10409238.2017.1394264.
- Carol W. Greider, Elizabeth H. Blackburn: Identification of a specific telomere terminal transferase activity in Tetrahymena extracts. In: Cell. Band 43, 1985, S. 405–413, doi:10.1016/0092-8674(85)90170-9, PMID 3907856.
- Lynne Suzanne Cox: Molecular Themes in DNA Replication. Royal Society of Chemistry, 2009, ISBN 978-0-85404-164-0, S. 156.
- Teresa Rivera, Candy Haggblom, Sandro Cosconati, Jan Karlseder: A balance between elongation and trimming regulates telomere stability in stem cellls. In: Nature structural & molecular biology. Band 24, Nummer 1, 01 2017, S. 30–39, doi:10.1038/nsmb.3335, PMID 27918544, PMC 5215970 (freier Volltext).
- Julia Su Zhou, Eros Lazzerini Denchi: How stem cells keep telomeres in check. In: Differentiation; research in biological diversity. Band 100, 2018 Mar - Apr, S. 21–25, doi:10.1016/j.diff.2018.01.004, PMID 29413749, PMC 5889314 (freier Volltext) (Review).
- Hewson Swift: The constancy of desoxyribose nucleic acid in plant nuclei. In: Proc Nat Acad Sci USA 36, 1950: 643–654.
- E R Gaginskaya, V L Kasyanov, G L Kogan: Amplification of ribosomal genes and formation of extrachromosomal nucleoli in oocytes of starfish Henricia hayashi (Asteroidea: Echinasteridae). In: Cell Differ 23, 1988: 53–60.
- Ulrich Scheer, Michael F Trendelenburg, G Krohne, Werner W Franke: Lengths and patterns of transcriptional units in the amplified nucleoli of oocytes of Xenopus laevis. In: Chromosoma 60, 2, 1977: 147–167.
- Allan C Spradling: The organizattion and amplification of two chromosomal domains containing Drosophila chorion genes. In: Cell 27, 1981: 193–201.
- Terry L Orr-Weaver, Allan C Spradling: Drosophila chorion gene amplification requires an upstream region regulating s18 transcription. In: Molecular and Cellular Biology. Band 6, Nummer 12, Dezember 1986, S. 4624–4633, doi:10.1128/mcb.6.12.4624, PMID 3099171, PMC 367247 (freier Volltext).
- E N Andreyeva, T D Kolesnikova, E S Belyaeva, R L Glaser, Igor F Zhimulev: Local DNA underreplication correlates with accumulation of phosphorylated H2Av in the Drosophila melanogaster polytene chromosomes. In: Chromosome Res 16, 2008: 851–862.
- Helmut Zacharias: Allocyclic behaviour and underreplication of the nucleolus chromosome in Pseudodiamesa (Chironomidae). In: Chromosoma 89, 1984: 263–273.
- Emil Heitz: Über α- und β-Heterochromatin sowie Konstanz und Bau der Chromomeren bei Drosophila. In: Biol Zentralblatt 54,1934: 588–609. → Seite 596: Beschreibt erstmals Unterreplikation des Heterochromatins bei Drosophila virilis.
- Allan C Spradling: Polytene chromosome structure and somatic genome instability. In: Cold Spring Harb Symp Quant Biol 82, 2017: 293–304. PDF.
- Theodor Boveri: Die Entwicklung von Ascaris megalocephala mit besonderer Rücksicht auf die Kernverhältnisse. In: Festschrift zum siebzigsten Geburtstag von Carl von Kupffer. Fischer, Jena 1899: 383–429.
- Theodor Boveri: Die Potenzen der Ascarisblastomeren bei abgeänderter Furchung. Zugleich ein Beitrag zur Frage qualitativ-ungleicher Chromosomen-Teilung. In: Festschrift 60. Geburtstag Richard Hertwigs. Vol III. Fischer, Jena 1910: 133–214.
- Sigrid Beermann: The diminution of heterochromatic chromosomal segments in Cyclops (Crustacea, Copepoda). In: Chromosoma 60, 1977: 297–344.
- R A Finch: Tissue-specific elimination of alternative whole parental genomes in one barley hybrid. In: Chromosoma 88, 1983: 386–393.
- Walter Nagl: Heterochromatin elimination in the orchid Dendrobium. In: Protoplasma 118, 1983: 234–237.
- Dieter Ammermann: Germ line specific DNA and chromosomes of the ciliate Stylonychia lemnae. In: Chromosoma 95, 1987: 37–43.
- Jianbin Wang, Shenghan Gao, Yulia Mostovoy, Yuanyuan Kang, Maxim Zagoskin, Yongqiao Sun, […], Richard E Davis: Comparative genome analysis of programmed DNA elimination in nematodes. In: Genome research. Band 27, Nummer 12, 12 2017, S. 2001–2014, doi:10.1101/gr.225730.117, PMID 29118011, PMC 5741062 (freier Volltext).
- Elizabeth H Blackburn: Telomerases. In: Annu Rev Biochem 1992.61, 1992: 113–129. PDF.
- Carol W Greider, Elizabeth H Blackburn: A telommeric sequence in the RNA of Tetrahymena telomerase required for telomere repeat synthesis. In: Nature 337, 6205, 1989: 331–337. → Seite 335: „The elongation translocation mechanism.“
- Helmut Zacharias, Inge Kronberg: Telomere: Ende gut, alles gut. In: Biologie in unserer Zeit 6, 2009: 366–367.
- David Van Ly, Ronnie Ren Jie Low, Sonja Frölich, Tara K Bartolec, Georgia R Kafer, Hilda A Pickett, Katharina Gaus, Anthony J Cesare: Telomere loop dynamics in chromosome end protection. In: Mol Cell 71, 4, 2018: 510–525.e6. PDF.
- Elizabeth H Blackburn, Carol W Greider, Jack W Szostak: Telomeres and telomerase: The path from maize, Tetrahymena and yeast to human cancer and aging. In: Nature Med 12, 2006: 1133–1138. DOI: 10.1038/nm1006-1133.
- Kara J Turner, Vimal Vasu, Darren K Griffin: Telomere biology and human phenotype. In: Cells. Band 8, Nummer 1, 01 2019, S. , doi:10.3390/cells8010073, PMID 30669451, PMC 6356320 (freier Volltext) (Review).
- Elena Casacuberta, Mary-Lou Pardue: HeT-A elements in Drosophila virilis: Retrotransposon telomeres are conserved across the Drosophila genus. In: Proceedings of the National Academy of Sciences. Band 100, Nummer 24, November 2003, S. 14091–14096, doi:10.1073/pnas.1936193100, PMID 14614149, PMC 283551 (freier Volltext).
- Elena Casacuberta: Drosophila: Retrotransposons making up telomeres. In: Viruses. Band 9, Nummer 7, 2017, doi:10.3390/v9070192, PMID 28753967, PMC 5537684 (freier Volltext) (Review). → Seite 10: Vergleicht Ähnlichkeiten der Telomer-Retrotransposons mit dem Telomerase-Mechanismus.
- Thomas J Nicholls, Michal Minczuk: In D-loop: 40 years of mitochondrial 7S DNA. In: Exp Gerontol 56, 2014: 175 –181. → Die Funktion der 7S-DNA steht hier immer noch zur Diskussion.
- ssDNA Rolling circle. In: ViralZone. Swiss Institute of Bioinformatics. Abgerufen am 29. März 2021.
- Josef Köck, Christine Rösler, Jing-Jing Zhang, Hubert E Blum, Michael Nassal, Christian Thoma: Generation of covalently closed circular DNA of hepatitis B viruses via intracellular recycling is regulated in a virus specific manner. In: PLoS pathogens. Band 6, Nummer 9, September 2010, S. e1001082, doi:10.1371/journal.ppat.1001082, PMID 20824087, PMC 2932716 (freier Volltext).
- J A Ruiz-Masó, C Machón, L Bordanaba-Ruiseco, M Espinosa, M Coll, G Del Solar: Plasmid rolling-circle replication. In: Microbiol Spectr 3, 1, 2015: PLAS-0035-2014. PDF.
- T Santos, P Pereira, J A Queiroz, C Cruz, F Sousa: Plasmid production and purification: An integrated experiment-based biochemistry and biotechnology laboratory course. In: Biochem Mol Biol Educ 2019 Nov; 47, 6, 2019: 638–643.
- Salvador Edward Luria, James E. Darnell Jr., David Baltimore, Allan Campbell: General Virology. 3rd Edition. John Wiley & Sons, New York etc. 1978. ISBN 0-471-55640-8. → Seiten 306f: Viral genetic systems: Classification of animal viruses.
- Eliszabeth Pennisi: Circular DNA throws biologists for a loop. In: Science 356, 6342, 2017: 996.