Reoviridae
Die Reoviridae bilden die größte Familie von unbehüllten Viren mit doppelsträngiger RNA (dsRNA). Zusammen mit den Birnaviridae und Partitiviridae sind sie die einzigen Viren, deren dsRNA-Genom segmentiert vorliegt. Zur Familie gehören so wichtige Erreger wie das Blauzungenvirus, die humanen Rotaviren oder das Colorado-Zeckenfieber-Virus. Der Name der Familie ist ein Akronym aus „respiratory, enteric, orphan“, da man der Ansicht war, dass Mitglieder dieser taxonomischen Gruppe nur Darmerkrankungen (enteric), Atemwegserkrankungen (respiratory) oder keine Erkrankung (orphan) hervorrufen würden. Tatsächlich wurden die Reoviridae bei vielen Säugetieren gefunden, ebenso bei Reptilien, Fischen, Krustentieren und Insekten. Drei Gattungen der Familie (Fijivirus, Phytoreovirus und Oryzavirus) umfassen Erreger bei Pflanzen (Pflanzenviren) und Pilzen (Mykoviren).
| Reoviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
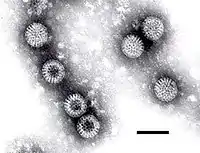
Rotaviren im TEM | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Reoviridae | ||||||||||||||
| Links | ||||||||||||||
|
Morphologie

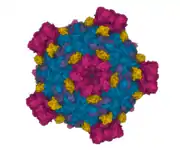
Die reifen Virusteilchenen (Virionen) der Reoviridae sind unbehüllte, ikosaedrische Kapside mit einem Durchmesser von etwa 60–80 nm. Charakteristisch für die meisten Gattungen der Familie (mit Ausnahme der Gattung Cypovirus) ist der Aufbau des Virions aus zwei ineinander geschachtelte und eng wechselwirkende Kapside (T=13), die wiederum eine innere, regelmäßige Corestruktur umgeben. Die so aus drei Lagen von Proteinen aufgebauten Virionen zeigen daher im Elektronenmikroskop eine sehr typische, dickwandige Struktur mit regelmäßigen, speichenförmigen Verdickungen. Von dieser Speichenstruktur abgeleitet erhielt die Gattung Rotavirus (lat. rota: Rad) ihren Namen. Diese Speichenstruktur entsteht durch 132 Öffnungen des äußeren Kapsids, die in ebenso viele Kanäle des inneren Kapsids münden. Diese Kanäle, die bis an das innere Core reichen, lassen eine Diffusion von Ionen in das Virion zu.
Dadurch, dass das innere Core und das innere Kapsid für die korrekte Verpackung der RNA-Segmente verantwortlich sind, kann das äußere Kapsid eine relativ große Variabilität der Oberflächenstrukturen aufweisen ohne die Stabilität des Virions und die Vollständigkeit des Genoms zu beeinträchtigen. Daher zeigen sich bei vielen Reoviren zahlreiche Spezies und Serotypen.
In der inneren Coreschale befindet sich ein Molekül der viralen RNA-Polymerase und Guanyltransferase. Bei einigen Gattungen sind Myristyl-Reste (Tetradecyl-Reste) kovalent an das Kapsid gebunden. Bei den Gattungen Rotavirus, Orbivirus und Coltivirus kommen unreife Virionen vor, die Reste einer Lipidhülle besitzen. Bei diesen Gattungen geschieht das Verlassen der Zelle durch Knospung an der Zellmembran (Orbivirus, Coltivirus) oder der Membran des Endoplasmatischen Retikulums (ER) (Rotavirus). Durch einen enzymatischen Reifungsschritt verlieren die Virionen die Lipidhülle wieder. Bei einigen Gattungen wird das äußere Kapsid durch wirtseigene Proteasen (z. B. Trypsin, Chymotrypsin) verändert, wodurch sogenannte „infectious subviral particles“ (ISVPs) entstehen. Nur die ISVPs sind infektiös und der Übergang von unreifen Virionen zu ISVPs spielt bei diesen Viren eine entscheidende Rolle in der Krankheitsentstehung und der unterschiedlichen Erscheinungsform der Virionen im Darm, Speichel oder Blutserum.
Genom
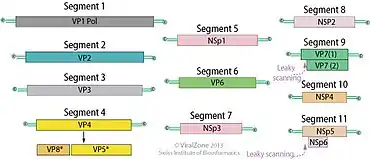
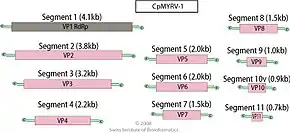
Das Genom besteht aus einer doppelsträngigen RNA, die je nach Gattung in 10 bis 12 Segmente unterteilt ist. Die Molmasse der Segmente reicht von 0,2 bis 3 × 106 Da. Die virale mRNA der Reoviridae besitzt keinen Poly-A-Schwanz. Bei einigen Virusspezies findet man kurze, einzelsträngige RNA-Oligonukleotide in der inneren Coreschale.
Systematik
Innere Systematik
Mit Stand November 2018 gliedern sich nach Maßgabe des ICTV die Reoviriridae wie folgt (neben den Typusspezies ist nur eine Auswahl weiterer Spezies angegeben):[3]
- Familie Reoviridae
- Unterfamilie Sedoreovirinae
- Genus Cardoreovirus
- Spezies Eriocheir-sinensis-Reovirus (en. Eirocheir sinensis reovirus, ESRV, Typusspezies) bei der Chinesischen Wollhandkrabbe[4]
- Spezies „Macropipus-depurator-Virus“ (en. „Macropipus-depurator-Virus“, MdRV, unbestätigt) bei Decapoda[4]
- Macropipus-depurator-P-Virus (MdRV-P, DpPV)
- Spezies „Carcinus-mediterraneus-Virus“ (en. „Carcinus mediterraneus reovirus“, CMRV, unbestätigt) bei Zehnfußkrebsen (Decapoda)[4]
- Carcinus-mediterraneus-W2-Virus (CmRV-W2, CcRV-W2)
- Genus Mimoreovirus (früher Mimovirus)[5] infiziert Micromonas pusilla.
- Spezies Afrikanisches Pferdepestvirus (en. African horse sickness virus, AHSV)
- Spezies Blauzungenvirus (en. Bluetongue virus, BTV, Typusspezies)
- Spezies Epizootische-Hämorrhagie-Virus (en. Epizootic hemorrhagic disease virus, EHDV)
- Spezies Spezies Great Island virus
- Kemerovo-Virus (en. Kemerovo virus, KEMV)
- Spezies Lebombo-Virus (en. Lebombo virus, LEBV)
- Spezies Lipovnik-Virus (en. Lipovnik virus, LIPV, Vektor: Zecken)
- Spezies Orungo-Virus (en. Orungo virus, ORUV)
- Spezies Palyam-Virus (en. Palyam virus, PALV)
- Chuzan-Virus (en. Chuzan virus, CHUV)
- Spezies Peruanisches Pferdepestvirus (en. Peruvian horse sickness virus, PHSV)
- Spezies St. Croix River-Virus (en. ‚St Croix River virus, SCRV)
- Spezies Tribec-Virus (auch Tribeč-Virus, en. Tribec virus, inoffiziell Tribeč virus, TRBV)
- Spezies Umatilla-Virus (en. Umatilla virus, UMAV)
- Genus Phytoreovirus
- Spezies Wound tumor virus (WTV, Typusspezies)
- Spezies Rice dwarf virus (RDV)
- Spezies Rotavirus A (RVA, Typusspezies)
- BoRV-A/UK
- SiRV-A/SA11
- Spezies Rotavirus B (RVB)
- Hu/MuRV-B/IDIR,
- Spezies Rotavirus C (RVC)
- PoRV-C/Co
- Spezies Rotavirus D bis I
- Genus Seadornavirus
- Spezies Banna-Virus (en. Banna virus, BAV, Typusspezies)
- Spezies Liaoning-Virus (en. Liao ning virus, LNV)
- Spezies Kadipiro-Virus (en. Kadipiro virus, KDV)
- Unterfamilie Spinareovirinae
- Genus Aquareovirus
- Spezies Aquareovirus A (Typusspezies)
- Spezies Aquareovirus B
- Spezies Aquareovirus C
- Golden shiner reovirus (GSRV)
- Grass carp reovirus (GCRV)
- Spezies Aquareovirus E
- Spezies Aquareovirus G
- Genus Coltivirus
- Spezies Colorado-Zeckenfieber-Virus (en. Colorado tick fever virus, CTFV, Typusspezies)
- Spezies Eyach-Virus (en. Eyach virus, EYAV)
- Genus Cypovirus[7]
- Spezies Cypovirus 1 (Typusspezies)
- Spezies Cypovirus 2 bis 16
- Genus Dinovernavirus
- Spezies Aedes-pseudoscutellaris-Virus (en. Aedes pseudoscutellaris virus, offiziell Aedes pseudoscutellaris reovirus, APRV, Typusspezies)
- Spezies „Fakovirus“ (Vorschlag)[8]
- Genus Fijivirus
- Spezies Fiji disease virus (Typusspezies)
- Genus Idnoreovirus
- Spezies Idnoreovirus 1 (Typusspezies)
- Spezies Idnoreovirus 2 bis 5
- Genus Mycoreovirus
- Spezies Mycoreovirus 1 (Typusspezies)
- Spezies Mycoreovirus 2 und 3
- Genus Orthoreovirus[9]
- Spezies Mammalian orthoreovirus (MRV, Typusspezies)
- Genus Oryzavirus
- Spezies Rice ragged stunt virus (RRSV, Typusspezies)
- Spezies Echinochloa ragged stunt virus
- Nicht-klassifizierte Virusspezies innerhalb der Familie Reoviridae:
- Spezies „Buthus-occitanus-Reovirus“ (en. „Buthus occitanus reovirus“, „BoRV“) bei Skorpionen der Familie Buthidae
- Spezies „Cimex-lectularis-Reovirus“ (en. „Cimex lactularius reovirus“, „ClRV“) bei der Bettwanze Cimex lectularius
- Spezies „Porcellio-dilatatus-Reovirus“ (en. „Porcellio dilatatus reovirus“, „PdRV“) bei Landasseln (Isopoda: Oniscidea) der Spezies Porcellio dilatatus
- Spezies „Scylla-serrata-Reovirus“ (en. „Scylla serrata reovirus“, „Mud crab reovirus“, „SsRV“ bzw. „MCRV“)
- Spezies „Operophtera-brumata-Reovirus“ (en. „Operophtera brumata reovirus“, „ObRV“) beim Kleinen Frostspanner
Das folgende Kladogramm gibt vereinfacht die Verwandtschaftsbeziehungen wieder, wie sie im 9. Report des International Committee on Taxonomy of Viruses (ICTV, 2011) und von MacLachlan/Dubovi (2016);[4][11] sowie fast übereinstimmend von Belhouchet et al. (2010)[12] vorgeschlagen wurde:
| Reoviridae |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Für einige der Gattungen sind die abgekürzten Speziesnamen angegeben, diese umfassen jeweils einen oder mehrere der untersuchten zugehörigen Stämme (englisch strains).
In älteren Arbeiten gibt es noch gewisse Abweichungen (z. B. bei Attoui et al. (2006)[13] bei Quito-Avila (2011)[14] und bei Ke et al. (2010).[15]
Äußere Systematik
Das ICTV hat mit der Master Species List #35 vom März 2020 die Reoviridae dem neu geschaffenen Phylum Duplornaviricota zugeordnet.[16] Eine Kladogramm findet sich bei Picornavirales §ICTV Master Species List #35.
Quellen
- P. P. C. Mertens, C. Wei, B. Hillmann: Family Reoviridae. In: C. M. Fauquet, M. A. Mayo et al.: Eighth Report of the International Committee on Taxonomy of Viruses. London, San Diego 2004, S. 447–555.
- S. Mordrow, D. Falke, U. Truyen: Molekulare Virologie. 2. Auflage, Heidelberg / Berlin 2003, ISBN 3-8274-1086-X.
Weblinks
- Reoviridae (NCBI)
Einzelnachweise
- ICTV Master Species List 2018b v1 MSL #34, Feb. 2019
- ICTV: Bluetongue virus, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- ICTV: Master Species List 2018a v1 MSL including all taxa updates since the 2017 release. Fall 2018 (MSL #33)
- ICTV: dsRNA Viruses: Reoviridae, in: ICTV 9th Report (2011), 9. Report des ICTV als Buch
- Joaquín Martínez Martínez, Arjan Boere, Ilana Gilg, Jan W. M. van Lent, Harry J. Witte, Judith D. L. van Bleijswijk, Corina P. D. Brussaard: New lipid envelope-containing dsDNA virus isolates infecting Micromonas pusilla reveal a separate phylogenetic group, in: AME Band 74, Nr. 1, S. 17–78, Januar 2015, doi:10.3354/ame01723, ResearchGate, PDF.
- SIB: Orbivirus, auf: ViralZone
- SIB: Cypovirus, auf: ViralZone
- NCBI: Fako virus (species)
- SIB: Orthoreovirus, auf: ViralZone
- Kelly Simone Bateman et al.: A taxonomic review of viruses infecting crustaceans with an emphasis on wild hosts. In: Journal of Invertebrate Pathology. 147, Januar 2017, doi:10.1016/j.jip.2017.01.010
- N. James MacLachlan, Edward J. Dubovi: Fenner’s Veterinary Virology. 5. Auflage 2016, ISBN 978-0-12-800946-8, doi:10.1016/C2013-0-06921-6.
- Mourad Belhouchet et al.: Complete sequence of Great Island virus and comparison with the T2 and outer-capsid proteins of Kemerovo, Lipovnik and Tribec viruses (genus Orbivirus, family Reoviridae). In: Journal of General Virology. 91(Pt 12): S. 2985–2993, Dezember 2010, doi:10.1099/vir.0.024760-0
- Houssam Attoui et al: Micromonas pusilla reovirus: A new member of the family Reoviridae assigned to a novel proposed genus (Mimoreovirus). In: Journal of General Virology. 87(Pt 5), S. 1375–1383, Juni 2006, doi:10.1099/vir.0.81584-0.
- Diego F. Quito-Avila et al.: Complete sequence and genetic characterization of Raspberry latent virus, a novel member of the family Reoviridae. In: Virus Research. Band 155, Nr. 2, Februar 2011, S. 397–405, doi:10.1016/j.virusres.2010.11.008.
- F. J. Ke, L. B. He et al.: Turbot reovirus (SMReV) genome encoding a FAST protein with a non-AUG start site. In: BMC Genomics. 2010, doi:10.1186/1471-2164-12-323.
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)