Duplodnaviria
Duplodnaviria ist ein Realm von Viren, der alle doppelsträngigen DNA-Viren umfasst, die für das Hauptkapsidprotein (englisch major capsid protein) HK94 (HK97-MCP) kodieren.[3] Frühere Bezeichnung für diese Gruppe waren HK97-like group oder HK97 major capsid protein supermodule.[4][3]
| Duplodnaviria | ||||||
|---|---|---|---|---|---|---|
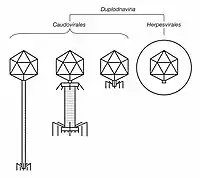
Illustrierte Beispiele von Virusteilchen der Duplodnaviria. | ||||||
| Systematik | ||||||
| ||||||
| Taxonomische Merkmale | ||||||
| ||||||
| Wissenschaftlicher Name | ||||||
| Duplodnaviria | ||||||
| Links | ||||||
|
Die Mitglieder dieses Realm haben auch eine Reihe anderer Proteine gemeinsam, die an der Struktur und dem Aufbau des viralen Kapsids mit ikosaedrischer Form beteiligt sind. Außer dem Kapsid mit HK97-MCP selbst, sind das die Portalproteine. Sie bilden die Öffnung des Kapsids, durch das die DNA (beim Zusammenbau der Virusteilchen) ein- und (nei der Infektion der Wirtszelle) austritt. Eine Kapsidreifungsprotease (englisch capsid maturation protease) leert das Innere des Kapsids, bevor virale DNA in die Wirtszelle injiziert wird, und ein Terminase-Enzym fügt die virale DNA beim Zusammenbau in das Kapsid ein, um vollständige Viruspartikel (Virionen), zusammenzusetzen.[3][5]
Im Realm Duplodnaviria gibt es nur ein Subtaxon, das Reich Heunggongvirae. Dieses hat zwei Phyla, die jeweils bis zum Rang der Ordnung monotypisch sind (Stand April 2020). Daher gibt es in den Duplodnaviria zur Zeit nur zwei Ordnungen, die Caudovirales (Bakteriophagen mit Kopf-Schwanz-Struktur, die nur Prokaryoten – Bakterien und Archaeen – infizieren) und die Herpesvirales (Herpesviren im weiteren Sinn, die nur Eukaryoten infizieren).[3][6]
Caudovirales sind weltweit allgegenwärtig und sehr vielfältig, weshalb ihre Vertreter möglicherweise zu den ältesten Viren überhaupt gehören. Wahrscheinlich stammen die Herpesviren entweder von einem gemeinsamen Vorfahren mit den Caudovirales ab, wenn dieser in seiner Evolution eine Abstammungslinie (Klade) hervorgebracht hat, die Eukaryoten infizieren konnte; oder sie stammen ab von einem divergierenden Zweig innerhalb der Caudovirales.[7]
Die verwandtschaftliche Beziehung zwischen den Schwanzphagen Caudovirales und den Herpesviren ist seit langem bekannt. Das International Committee on Taxonomy of Viruses (ICTV) hat diesen Sachverhalt 2019 mit der Anerkennung des Realm Duplodnaviria Rechnung getragen. Der erste Teil des Namens (duplo) kommt aus dem Lateinischen und bedeutet doppel; der mittlere Teil (dna) bezieht sich auf die Desoxyribonukleinsäure (DNA), da alle Mitglieder des Realm doppelsträngige DNA-Viren (dsDNA) sind; und der letzte Teil (viria) ist das Suffix für Realm.[3] Als dsDNA-Viren gehören die Mitglieder dieses Realm alle zur Baltimore-Gruppe I (dsDNA-Viren).
Die Baltimore-Klassifikation basiert auf der Art und Weise der Erzeugung der viralen Messenger-RNA (mRNA) und häufig zusammen mit der Standard-Virustaxonomie verwendet wird, die auf der Evolutionsgeschichte und damit Verwandtschaftsbeziehungen basiert. In der Virustaxonomie sind Realm die höchste Ebene (Rangstufe), und Duplodnaviria ist einer von derzeit (Stand April 2020) vier solchen Realm; die anderen sind Monodnaviria, Varidnaviria (beide beinhalten auch DNA-Viren) sowie Riboviria (einziger Realm mit RNA-Viren einschließlich revers transkribierender Viren wie die Retroviren).[6] Etliche Virentaxa sind jedoch bisher noch keinen Realm zugeordnet.
Aufbau
Alle Viren der Duplodnaviria weisen ein ikosaedrisches Kapsid auf, das aus einem Hauptkapsidprotein besteht mit einer spezifischen gefalteten Struktur, die als HK97-Faltung bezeichnet wird und nach dem HK97-Hauptkapsidprotein (HK97-MCP) der Phagenart Escherichia-Virus HK97 benannt ist. Trotz signifikanter Unterschiede innerhalb der Duplodnaviria ist die Grundstruktur des Proteins bei allen Arten in diesem Realm erhalten (konserviert). Andere für Struktur und Zusammenbau der Kapside verantwortliche gemeinsame Proteine umfassen:[4][3][8]
- ein Portalprotein, aus dem die Öffnung des Kapsids besteht,
- eine Protease, die das Kapsid entleert, bevor DNA eingefügt wird
- das Terminase-Enzym, das die DNA in das Kapsid einfügt.
Virusreplikation
Nachdem HK97-MCP durch die Ribosomen der Wirtszelle synthetisiert wurde, wird das Virus-Kapsid zusammengesetzt, indem die Proteine aneinander binden. Das Innere des Kapsids enthält Gerüstproteine (englisch scaffold proteins), die die Konstruktion des Kapsids in seiner geometrischen Form (die sog. Assemblierung) steuern. In Abwesenheit separater Gerüstproteine wirkt die Delta-Domäne von HK97-MCP, die zur Innenseite des Kapsids zeigt, ersatzweise als Gerüstprotein.[4][5][8]
Danach wird eine zylindrische Öffnung im Kapsid gebildet, die als Ein- und Ausgang für virale DNA dient, und daher als genannt Portal bezeichnet wird. Das Portal entspricht daher in der Funktion dem sog. Stargate, wie es bei manchen DNA-Riesenviren zu finden ist (z. B. Mimivirus). Das Portal wird mit Portalproteinen an einem der 12 Eckpunkte des Kapsids erzeugt. Das Gerüstprotein (ggf. die Delta-Domäne von HK97-MCP) wird von der Innenseite des Kapsids durch eine Kapsid-Reifungs-Protease (englisch capsid maturation protease) entfernt, die auch ein Teil des Gerüstes sein kann. Diese zerstückelt das Gerüstprotein (ggf. also sich selbst) in kleinere Moleküle (Proteolyse), so dass danach ein noch leeres Kapsid übrig bleibt.[5][8] Bei Herpesviren wird die Protease auch als Assemblin bezeichnet.
Gleichzeitig mit der Kapsid-Assemblierung erfolgt die Replikation der viralen DNA, wodurch Concatamere (englisch concatemer) entstehen, d. h. lange DNA-Moleküle, die zahlreiche Kopien des viralen Genoms enthalten. Ein Terminase genanntes Enzym, bestehend aus einer großen und einer kleinen Untereinheit, findet die virale DNA innerhalb der Zelle über ihre kleine Untereinheit, schneidet die Concatamere und erzeugt die Enden des Genoms (genannt Termini, Plural von Terminus). Die Terminase erkennt im Genom ein Verpackungssignal (als spezielle DNA-Sequenz) und schneidet die Nukleinsäure, wodurch ein freies Ende entsteht, an das sie bindet.[8]
Die jetzt an das Concatamer gebundene Terminase, bindet sich danach an das Kapsidportal und beginnt, die DNA von außerhalb des Kapsids nach innen zu verlagern. Dieser Vorgang verbraucht Energie, die vermöge einer ATP-Hydrolyse durch die große Untereinheit erzeugt wird. Je mehr DNA in das Kapsid eingefügt wird, desto mehr dehnt sich das Kapsid aus, womit dieses dünner wird und seine Oberfläche nähert sich zunehmend der ikosaedrischen Form an. Sobald sich das Genom vollständig im Inneren befindet, schneidet die Terminase das Concatamer erneut ab und vervollständigt damit den Verpackungsvorgang. Die Terminase löst sich dann vom Portal und wiederholt diesen Vorgang, bis alle DNA-Genome des Concatamers verpackt sind.[8]
Bei Bakteriophagen mit Kopf-Schwanz-Struktur (Caudovirales) wird nach der DNA-Verpackung der Schwanz des Virions, der zuvor separat zusammengesetzt wurde, am Portal an das Kapsid gebunden, das bei diesem Virustyp üblicherweise dann als Kopf bezeichnet wird. Bei diesen Viren finde man manchmal auch sog. Dekorations-Proteine, die sich an die Oberfläche des Kapsids anlagern, um die Struktur des Kapsids zu verstärken.
Nachdem das Virion vollständig in der Wirtszelle zusammengebaut ist, verlässt es die Zelle.[8]
Ätiologie
Bei den Schwanzphagen führt eine Infektion üblicherweise zum Zelltod des Wirtsbakteriums oder -archaeons. Herpesviren verursachen jedoch eine Vielzahl von Krankheiten in ihren Wirten. Beim Menschen gehören zu den durch Herpesviren verursachten Krankheiten Herpes simplex, Windpocken und Gürtelrose (Herpes Zoster), Roseola (Drei-Tage-Fieber) und verschiedene Krebsarten wie Lymphom, Nasopharynxkarzinom und Kaposi-Sarkom. Herpesviren sind auch dafür bekannt, dass sie dem Immunsystem ausweichen und eine latente Infektion verursachen können, die über lange Zeiträume andauert. Beispiele hierfür sind das Varicella-Zoster-Virus, das typischerweise früh im Leben Windpocken und häufig Jahrzehnte später Gürtelrose verursacht..[10]
Systematik
Der Realm Duplodnaviria beinhaltet derzeit (Stand April 2020) nur ein einziges Reich Heunggongvirae, das in zwei Phyla unterteilt ist, die bis zum Rang der Ordnung monotypisch sind, d. h. jeweils nur eine einzige Ordnung enthalten:[6]
- Realm: Duplodnaviria
- Reich: Heunggongvirae
- Phylum: Peploviricota
- Klasse: Herviviricetes
- Ordnung: Herpesvirales – Herpesviren (im weiteren Sinn) – infizieren nur Tiere, also Eukaryoten.
- Phylum: Uroviricota
- Klasse: Caudoviricetes
- Ordnung: Caudovirales – Phagen mit Kopf-Schwanz-Aufbau – infizieren nur Archaeen und Bakterien, d. h. Prokaryoten.
Entwicklungsgeschichte
Phagen mit Kopf-Schwanz-Struktur sind möglicherweise die älteste Virenlinie der Welt:
- sie sind auf der ganzen Erde allgegenwärtig
- sie infizieren nur Prokaryoten – Eukaryoten sind vermutlich erst später aus einem Zweig der (prokaryotischen) Archaeen entstanden, siehe Eozyten-Hypothese)
- sie haben ein hohes Maß an Diversität.
Die stark divergierenden Virionstrukturen könnten aber auch auf unterschiedliche Ursprünge hinweisen. Der Ursprung von Herpesvirales ist unklar, aber es gibt zwei wahrscheinliche Szenarien:
- Die Caudovirales könnten zu verschiedenen Zeiten Linie hervorgebracht haben, die in der Lage waren, Eukaryoten zu infizieren. Die starke Ähnlichkeit, die die Herpesvirales mit den Caudovirales aufweisen, könnte darauf hinweisen, dass es diese rezente Nachkommen einer solchen Linie sind.
- Das zweite mögliche Szenario ist, dass die Herpesvirales eine stark weiterentwickelte ‚Ausreißer‘-Clade aus dem Inneren der Caudovirales sind. Dies wird dadurch unterstützt, dass einige Familien der Caudovirales, wie die Myoviridae (ud insbesondere deren Unterfamilie Tevenvirinae, eine relativ hohe Ähnlichkeit zu Herpesviren auf Basis bestimmter Aminosäuresequenzen ihrer Proteine zeigen (Proteom)[7]
Die ATPase-Untereinheit der Duplodnaviria-Terminasen, die Energie zum Verpacken der viralen DNA erzeugt, hat das gleiche allgemeine strukturelle Design einer P-Loop-Faltung (auch Walker-Motif genannt) wie die Verpackungs-ATPasen von der DJR-Viren des Realm Varidnaviria (englisch double jelly roll fold); sie sind jedoch ansonsten nicht direkt miteinander verwandt. Während die Viren der Duplodnaviria die HK97-Faltung für ihre Hauptkapsidproteine verwenden, sind die Hauptkapsidproteine bei den Varidnaviria stattdessen durch vertikale Einzel- oder Doppel-Jelly-Roll-Falten gekennzeichnet.[3]
Einzelnachweise
- ICTV:ICTV Taxonomy history: Human alphaherpesvirus 1, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- ICRV: ICTV Master Species List 2020.v1, New MSL including all taxa updates since the 2019 release, March 2021 (MSL #36)
- Eugen V. Koonin, V. V. Dolja, M. Krupovic, A. Varsani, Y. I. Wolf, N. Yutin, M. Zerbini, J. H. Kuhn: Create a megataxonomic framework, filling all principal/primary taxonomic ranks, for dsDNA viruses encoding HK97-type major capsid proteins (en, docx) In: International Committee on Taxonomy of Viruses. 18. Oktober 2019.
- M. M. Suhanovsky, C. M. Teschke: Nature's favorite building block: Deciphering folding and capsid assembly of proteins with the HK97-fold. In: Virology. 479–480, Mai 2015, S. 479–480. doi:10.1016/j.virol.2015.02.055. PMID 25864106. PMC 4424165 (freier Volltext).
- R. I. Duda, B. Oh, R. W. Hendrix: Functional domains of the HK97 capsid maturation protease and the mechanisms of protein encapsidation. In: J Mol Biol. 425, Nr. 15, 9. August 2013, S. 2765–2781. doi:10.1016/j.jmb.2013.05.002. PMID 23688818. PMC 3709472 (freier Volltext).
- Virus Taxonomy: 2019 Release. In: talk.ictvonline.org. International Committee on Taxonomy of Viruses.
- J. S. Andrade-Martínez, J. L. Moreno-Gallego, A. Reyes: Defining a Core Genome for the Herpesvirales and Exploring their Evolutionary Relationship with the Caudovirales. In: Sci Rep. 9, Nr. 1, August 2019, S. 11342. bibcode:2019NatSR...911342A. doi:10.1038/s41598-019-47742-z. PMID 31383901. PMC 6683198 (freier Volltext).
- V. B. Rao, M. Feiss: Mechanisms of DNA Packaging by Large Double-Stranded DNA Viruses. In: Annu Rev Virol. 2, Nr. 1, November 2015, S. 351–378. doi:10.1146/annurev-virology-100114-055212. PMID 26958920. PMC 4785836 (freier Volltext).
- Myoviridae. In: ViralZone. Swiss Institute of Bioinformatics. Abgerufen am 19. Mai 2020.
- Herpesviridae. In: ViralZone. Swiss Institute of Bioinformatics. Abgerufen am 19. Mai 2020.
Literatur
- C. W. Ward: Progress towards a higher taxonomy of viruses. In: Research in Virology. 144, Nr. 6, 1993, S. 419–53. doi:10.1016/S0923-2516(06)80059-2. PMID 8140287. PMC 7135741 (freier Volltext).