Narnavirus
Narnavirus ist eine Gattung von Einzelstrang-RNA-Viren positiver Polarität in der Familie Narnaviridae. Als natürliche Wirte (zumindest der offiziell bestätigten Mitglieder der Gattung) dienen Pilze, was diese Viren als Mykoviren klassifiziert.[2][3][4]
| Narnavirus | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
Die Narnaviren haben kein Kapsid und keine Virushülle, RNA-Genom und RdRp bilden einen nackten Ribonukleoprotein-Komplex | ||||||||||||||||
| Systematik | ||||||||||||||||
| ||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||
| ||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||
| Narnavirus | ||||||||||||||||
| Links | ||||||||||||||||
|
Es gibt in der Gattung derzeit (Stand 15. Juni 2021) zwei vom International Committee on Taxonomy of Viruses (ICTV) offiziell bestätigte Spezies (Arten).[2][3][4] Man hat gezeigt, dass Mitgliedsviren für die sexuelle Fortpflanzung des Schimmelpilzes Rhizopus microsporus erforderlich sind..[5]
Die Narnaviridae haben ein nacktes RNA-Genom ohne Kapsid, d. h. keine echten Virionen (Viruspartikel) und leiten ihren Namen von dieser Eigenschaft ab (englisch naked RNA).[6]
Beschreibung
Aufbau
Narnaviren (im Sinn von Mitgliedern der Gattung Narnavirus) haben kein echtes Virion. Sie haben weder Strukturproteine noch ein Kapsid.[7]
Genome
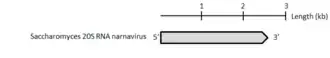
Das Genom der Narnaviren ist nicht segmentiert (monopartit) und besteht aus einem linearen RNA-Molekül positiver Polarität. Es gibt nur einen Offenen Leserahmen (en. open reading frame, ORF), der für die RNA-abhängige RNA-Polymerase (RdRp) kodiert. Das Genom ist mit der RdRp im Zytoplasma des Wirtspilzes assoziiert und bildet einen nackten Ribonukleoprotein-Komplex.[4]
Replikationszyklus
Die Replikation erfolgt im Zytoplasma. Sie folgt dem üblichen Modell der Replikation positiv-strängiger RNA-Viren; gleiches gilt für die Transkription. Das Virus verlässt die Wirtszelle durch Bewegung von Zelle zu Zelle. Nachgewiesen sind bisher (nur) Pilze als natürliche Wirte (näheres siehe unten). Die Übertragungswege sind vertikal (auf die Nachkommen des Wirts) und sexuell.[4]
Systematik
Innere Systematik
Die Gattung hat (mit Stand 15. Juni 2021) folgende vom ICTV bestätigten Mitgliedsspezies:[3][2]
- Saccharomyces 20S RNA narnavirus
- Saccharomyces 23S RNA narnavirus
Weitere vorgeschlagene Spezies nach NCBI sind (Auswahl):[8]
- „Aedes angustivittatus narnavirus“ (ATNV)[9]
- „Aedes taeniorhynchus narnavirus“ (AANV)[9]
- „Alternaria tenuissima narnavirus 1“
- „Aspergillus fumigatus narnavirus 1“
- „Aspergillus fumigatus narnavirus 2“
- „Beauveria bassiana narnavirus“
- „Blechmonas luni narnavirus 1“[10][11] (vermutlich Verschreiber für „Blechomonas luni narnavirus 1“: die Gattung Blechomonas – im Gegensatz zum Virus mit zwei ‚o‘ – ist Mitglied der Unterfamilie Blechomonadinae der Trypanosomatidae)[12][13][12]
- „Blechomonas maslovi narnavirus 1“[13]
- „Blechomonas wendygibsoni narnavirus 1“[13]
- „Botrytis cinerea narnavirus 2“
- „Botrytis cinerea narnavirus 4“
- „Bremia lactucae associated narnavirus 1“
- „Bremia lactucae associated narnavirus 2“
- „Bremia lactucae associated narnavirus 3“
- „Cladosporium tenuissimum narnavirus 1“
- „Coquillettidia venezuelensis narnavirus 1“ (CVNV1)
- „Coquillettidia venezuelensis narnavirus 2“ (CVNV2)
- „Erysiphe necator associated narnavirus 1“
- „Fusarium poae narnavirus 1“
- „Fusarium poae narnavirus 2“
- „Grapevine associated narnavirus-1“
- „Magnaporthe oryzae narnavirus 1“
- „Narnavirus I-329“
- „Neofusicoccum parvum narnavirus 1“
- „Neofusicoccum parvum narnavirus 2“
- „Neofusicoccum parvum narnavirus 3“
- „Oidiodendron maius splipalmivirus 1“ (Oidiodendron) ist eine Gattung von Echten Schlauchpilzen
- „Phytomonas serpens narnavirus 1“
- „Plasmopara viticola lesion associated narnavirus 1“
- „Psorophora varipes narnavirus“ (PVNV)
- „Puccinia striiformis narnavirus 1“
- „Sclerotinia sclerotiorum narnavirus 1“
- „Sclerotinia sclerotiorum narnavirus 2“
- „Tetranychus urticae-associated narnavirus“
Anmerkung: Die Namensgebungen weisen nicht alle auf Pilze hin. Entweder parasitieren die betreffenden Kandidatenmitglieder andere Eukaryoten (etwa Stechmücken): Bei Genomsequenzen aus der Metagenomik besteht jedoch auch die Möglichkeit, dass sie deswegen mit diesen vergesellschaftet (assoziiert) sind, weil ihr Wirt ein Pilz ist, der mit diesen Tieren, Pflanzen usw. in einer symbiotischen (vielleicht parasitären) Beziehung steht und auf oder in diesen lebt. Diese Frage ist in den betreffenden Fällen daher noch zu klären.[9]
Äußere Systematik
Zwar gibt es mit dem derzeitigen Stand vom 18. Juni 2021 in den Narnaviridae außerhalb der Gattung Narnavirus keine bestätigten Gattungen oder Spezies, das NCBI listet jedoch folgende Kandidaten (Auswahl):[14]
- Spezies „Aedes japonicus narnavirus 1“
- Spezies „Aedes japonicus narnavirus 1“
- Spezies „Annperkins narna-like virus“
- Spezies „Apple narna-like virus 1“
- Spezies „Apple narna-like virus 2“
- Spezies „Aspergillus lentulus narnavirus 1“
- Spezies „Barns Ness breadcrumb sponge narna-like virus 1“ bis „8“
- Spezies „Botryosphaeria dothidea narnavirus 1“
- Spezies „Botryosphaeria dothidea narnavirus 2“
- Spezies „Botryosphaeria dothidea narnavirus 3“
- Spezies „Botryosphaeria dothidea narnavirus 4“
- Spezies „Botrytis cinerea binarnavirus 1“
- Spezies „Botrytis cinerea binarnavirus 3“
- Spezies „Botrytis cinerea binarnavirus 5“
- Spezies „Culex narnavirus 1“
- Spezies „Cyathus narnavirus A“
- Spezies „Plasmodium vivax Narna-Like virus 1“
- Spezies „Puccinia narnavirus A“
- Spezies „Puccinia narnavirus B“
- Spezies „Puccinia narnavirus C“
- Spezies „Thrips tabaci associated narna-like virus 1“
- Spezies „Thrips tabaci narna-like virus 2“
Einzelnachweise
- ICTV Master Species List 2018b.v2. MSL #34, März 2019.
- ICTV: ICTV Master Species List 2020.v1, New MSL including all taxa updates since the 2019 release, March 2021 (MSL #36)
- Virus Taxonomy: 2020 Release. International Committee on Taxonomy of Viruses (ICTV). March 2021. Abgerufen am 15. Juni 2021.
- Viral Zone. ExPASy. Abgerufen am 15. Juni 2021.
- A. N. Espino-Vázquez, J. R. Bermúdez-Barrientos, J. F. Cabrera-Rangel, G. Córdova-López, F. Cardoso-Martínez, A. Martínez-Vázquez, D. A. Camarena-Pozos, S. J. Mondo, T. E. Pawlowska, C. Abreu-Goodger, L. P. Partida-Martínez: Narnaviruses: Novel players in fungal–bacterial symbioses. In: The ISME Journal. 2020. doi:10.1038/s41396-020-0638-y.
- Narnaviridae - Positive Sense RNA Viruses - Positive Sense RNA Viruses (2011) - ICTV. In: talk.ictvonline.org. Abgerufen am 15. Juni 2021.
- Valerian V. Dolja, Eugene Viktorovich Koonin: Capsid-Less RNA Viruses. In: ELS 2012, ISBN 978-0470016176, doi:10.1002/9780470015902.a0023269.
- NCBI: Narnavirus (genus), speziell unclassified Narnavirus (list)
- Joseph R. Fauver, Shamima Akter, Aldo Ivan Ortega Morales, William C. Black IV, Americo D. Rodriguez, Mark D. Stenglein, Gregory D. Ebel, James Weger-Lucarelli: A reverse-transcription/RNase H based protocol for depletion of mosquito ribosomal RNA facilitates viral intrahost evolution analysis, transcriptomics and pathogen discovery, in: Virology, Band 528, Februar 2019, S. 181–197, doi:10.1016/j.virol.2018.12.020
- Virus-Host DB: Blechmonas luni narnavirus 1
- UniProt: Taxonomy - Blechmonas luni narnavirus 1 (Species)
- NCBI: Blechmonas luni narnavirus 1 (species), NC_040829.1
- Danyil Grybchuk, Alexei Y. Kostygov, Diego H. Macedo, Jan Votýpka, Julius Lukeš, Vyacheslav Yurchenko: RNA Viruses in Blechomonas (Trypanosomatidae) and Evolution of Leishmaniavirus, in: ASM mBio, Band 9, 1. Oktober 2018, eissn: 2150-7511, doi:10.1128/mbio.01932-18, PMC 6191543 (freier Volltext), PMID 30327446
- NCBI: unclassified Narnaviridae (list)