Marnaviridae
Marnaviridae ist die Bezeichnung einer Familie von positiv-einzelsträngigen RNA-Viren in der Ordnung Picornavirales.[2] Die erste Art (Spezies) dieser Familie, die isoliert wurde (und daher die Typusart der Familie darstellt), ist das Heterosigma akashiwo RNA-Virus (HaRNAV) in der Gattung Marnavirus,[3] das die toxische, blütenbildende Raphidophyten-Alge Heterosigma akashiwo [en] infiziert.[4] Auf Basis von DNA-Sequenzierungen wurden weitere zwanzig marine RNA-Virusspezies in die Familie aufgenommen, verteilt auf sechs Gattungen.[5]
| Marnaviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
.png.webp)
EM-Aufnahme von Partikeln des | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Marnaviridae | ||||||||||||||
| Links | ||||||||||||||
|
HaRNAV wurde aus dem Wasser in der Straße von Georgia in British Columbia (Kanada) isoliert, aus einer konzentrierten Virusansammlung mit dem Wirt Heterosigma akashiwo (NEPCC 522).[6] HaRNAV darf nicht mit anderen Viren verwechselt werden, die diesen Wirt infizieren, wie z. B. dem dsDNA-Virus Heterosigma akashiwo virus 01 (HaV-1, mit Isolat HaV53) aus der Gattung Raphidovirus oder dem „Heterosigma akashiwo Nuclear Inclusion Virus“ (HaNIV, Vorschlag, ?Gattung Protobacilladnavirus).[7][8][9][10]
Beschreibung


Modifiziert nach ViralZone.[11]
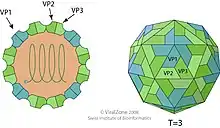
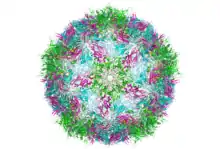
Die Viren der Marnaviridae sind nicht umhüllt und haben ikosaedrische Geometrie mit einer Triangulationszahl T=pseudo3. Der Durchmesser beträgt etwa 25 nm. Das Genom ist linear und monopartit (nicht segmentiert), mit einer Länge von ca. 8,6 kb (Kilobasen).[11] Das Kapsid besteht aus drei Hauptkapsidproteinen (major capsid proteins, MCPs: VP1, VP2, VP3), die jeweils eine Jelly-Roll-Faltung aufweisen, und einem Minor-Capsid-Protein (minor capsid proteins, mCP), das sich auf der Innenseite des Kapsids an den Polen der Fünffach-Achse befindet.[12]
Reproduktionszyklus
Die Replikation folgt dem üblichen Schema für (+)ssRNA-Viren (en. positive stranded RNA virus replication model). Die Transkription folgt ebenfalls dem üblichen Schema für (+)ssRNA-Viren (en. positive stranded RNA virus transcription). Die neu gebildeten Virionen verlassen die Wirtszelle durch tubulusgeführte Virusbewegung (en. tubule-guided viral movement).[11]
Als natürliche Wirte des einzigen gut bekannten Mitglieds HaRNAV der Familie Marnaviridae dient Heterosigma akashiwo [en] (Raphidophyceae). Es wird angenommen, dass auch die anderen Vertreter marines Phytoplankton infizieren.[11]
Systematik
Die Systematik der Marnaviridae ist nach ICTV mit Stand Anfang April 2021 wie folgt:[1]
Ordnung: Picornavirales
- Familie: Marnaviridae
- Gattung: Bacillarnavirus (drei Arten)
- Spezies: Chaetoceros socialis forma radians RNA virus 1 — Wirtsgattung Chaetoceros
- Spezies: Chaetoceros tenuissimus RNA virus 01 (CtenRNAV) — Wirtsgattung Chaetoceros
- Spezies: Rhizosolenia setigera RNA virus 01 — Wirtsgattung Rhizosolenia
- Gattung: Kusarnavirus (eine Art)
- Spezies: Astarnavirus mit Asterionellopsis glacialis RNA virus — Wirtsgattung Asterionellopsis
- Gattung: Labyrnavirus (eine Art)
- Spezies: Aurantiochytrium single-stranded RNA virus 01 — Wirtsgattung Aurantiochytrium
- Gattung: Locarnavirus (vier Arten)
- Spezies: Jericarnavirus B
- Spezies: Sanfarnavirus 1
- Spezies: Sanfarnavirus 2
- Spezies: Sanfarnavirus 3
- Gattung: Marnavirus (eine bestätigte Art, mehrere vorgeschlagene)[13]
- Spezies: Heterosigma akashiwo RNA virus (HaRNAV) mit Isolat SOG263[14][15] — Wirtsspezies Heterosigma akashiwo
- Spezies: „Forsythia suspensa marnavirus“ — Wirtsspezies Hänge-Forsythie
- Spezies: „Kummerowia striata marnavirus“ — Wirtsgattung Kummerowia
- Spezies: „Lactuca sativa marnavirus“ — Wirtsgattung Lactuca
- Spezies: „Lindernia crustacea marnavirus“ — Wirtsgattung Lindernia
- Spezies: „Trichosanthes kirilowii marnavirus“ — Wirtsspezies Trichosanthes kirilowii
- Spezies: „Zehneria japonica marnavirus“ — Wirtsgattung Zehneria
- Gattung: Salisharnavirus (vier Arten)
- Spezies: Britarnavirus 1
- Spezies: Britarnavirus 4
- Spezies: Palmarnavirus 128
- Spezies: Palmarnavirus 473
- Gattung: Sogarnavirus (sechs Arten)
- Spezies: Britarnavirus 2
- Spezies: Britarnavirus 3
- Spezies: Chaetarnavirus 2
- Spezies: Chaetenuissarnavirus II
- Spezies: Jericarnavirus A
- Spezies: Palmarnavirus 156
Einzelnachweise
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- Marnaviridae - Positive Sense RNA Viruses - Positive Sense RNA Viruses (2011) - International Committee on Taxonomy of Viruses (ICTV) (en) In: International Committee on Taxonomy of Viruses (ICTV). Abgerufen am 27. April 2017.
- ICTV: Virus Taxonomy: 2014 Release. Abgerufen am 15. Juni 2015.
- A. S. Lang, A. I. Culley, C. A. Suttle: Genome sequence and characterization of a virus (HaRNAV) related to picorna-like viruses that infects the marine toxic bloom-forming alga Heterosigma akashiwo. In: Virology. 320, Nr. 2, 2004, S. 206–217. doi:10.1016/j.virol.2003.10.015. PMID 15016544.
- M. Vlok, A. S. Lang, C. A. Suttle: Application of a sequence-based taxonomic classification method to uncultured and unclassified marine single-stranded RNA viruses in the order Picornavirales. In: Virus Evolution. 31;5(2):vez056, 2019. doi:10.1093/ve/vez056. PMID 31908848.
- Vera Tai, Janice E. Lawrence, Andrew S. Lang, Amy M. Chan, Alexander I. Culley, Curtis A. Suttle: Characterization of HaRNAV, a single-stranded RNA virus causing lysis of Heterosigma akashiwo (Raphidophyceae). In: Journal of Phycology. 39, Nr. 2, 28. März 2003, S. 206–217. doi:10.1046/j.1529-8817.2003.01162.x.
- Janice E. Lawrence, Amy M. Chan, Curtis A. Suttle: A novel virus (HaNIV) causes lysis of the toxic bloom-forming alga Heterosigma akashiwo (Raphidophyceae). In: Journal of Phycology. 37, Nr. 2, 1. Mai 2002, S. 216–222. doi:10.1046/j.1529-8817.2001.037002216.x.
- Y. Bettarel, J. Kan, K. Wang, Kurt E. Williamson, S. Cooney, S. Ribblett, F. Chen, K. Wommack, D. Coats: Isolation and preliminary characterisation of a small nuclear inclusion virus infecting the diatom Chaetoceros cf. gracilis, auf: Semantic Scholar – Biology – Aquatic Microbial Ecology, Corpus ID: 56157535, 2005, doi:10.3354/AME040103. „Chaetoceros nuclear inclusion virus: CspNIV“, „CspNIV shows some strong similarities with Heterosigma akashiwo nuclear inclusion virus (HaNIV)“
- Keizo Nagasaki: Dinoflagellates, diatoms, and their viruses, in: The Journal of Microbiology 46(3), Juli 2008, S. 235–243, doi:10.1007/s12275-008-0098-y. „CspNIV shows some strong similarities with Heterosigma akashiwo nuclear inclusion virus (HaNIV)“
- Stephanie Boone: Viruses and Algae, Februar 2004
- Viral Zone. ExPASy.
- Anna Munke, Kei Kimura, Yuji Tomaru, Kenta Okamoto: Capsid Structure of a Marine Algal Virus of the Order. In: Journal of Virology. 94, Nr. 9, 16. April 2020. doi:10.1128/JVI.01855-19.
- NCBI: Marnavirus (genus)
- NCBI: Heterosigma akashiwo RNA virus SOG263
- UniProt: UniProt: Proteomes - Heterosigma akashiwo RNA virus (strain SOG263)
Weblinks
- Viralzone: Marnaviridae
- ICTV
- NCBI: Marnaviridae (family)
- ICTV: Taxonomy History – Marnaviridae
- Janice E. Lawrence, Corina P. D. Brussaard, Curtis A. Suttle: Virus-Specific Responses of Heterosigma akashiwo to Infection, in: Appl Environ Microbiol, 72(12), Dezember 2006, S. 7829–7834, PMC 1694243 (freier Volltext), PMID 17041155, doi:10.1128/AEM.01207-06