Nanoviridae
Mit Nanoviridae wird eine Familie von Viren,[2] deren natürliche Wirte Pflanzen sind. Es gibt derzeit (23. Juni 2021) 14 vom International Committee on Taxonomy of Viruses (ICTV) bestätigte Spezies (Arten) in dieser Familie, die sich auf 2 Gattungen verteilen.[3][1]
| Nanoviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
Faba bean necrotic yellows virus (FBNYV) | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Nanoviridae | ||||||||||||||
| Links | ||||||||||||||
|
Bei der Gattung Nanovirus sind die Wirte Eudikotyledonen, vorwiegend Leguminosen (Familie Fabaceae); bei der Gattung Babuvirus dagegen Monokotyledonen (einkeimblättrige Pflanzen), vorwiegend Ingwerartige (Ordnung Zingiberales).
Die Infektion der Wirtspflanzen mit Viren der Nanoviridae führt zu verkümmertem Wachstum.[3][4][5]
Die Viren der Familie Nanoviridae benötigen einen virus-kodierten Hilfsfaktor für die Übertragung durch Blattläuse (Vektoren).
Etymologie und Bezeichnungen
Der Name der Familie leitet sich ab von griechisch νᾶνος nanos, deutsch ‚Zwerg‘, sowohl wegen ihres kleinen Genoms als auch wegen ihrer verkümmernden Wirkung auf infizierte Pflanzen.[3] Der Gattungsname Babuvirus ist eine Zusammenziehung aus der Bezeichnung der Typusspezies Banana bunchy top virus.
Im informellen Sprachgebrauch werden die Viren der Gattung Babuvirus als en. babuviruses ‚Babuviren‘, die der Gattung Nanovirus als en. nanoviruses ‚Nanoviren‘ bezeichnet, die Mitglieder der Familie Nanoviridae aber zur Unterscheidung als en. nanovirids ‚Nanoviriden‘ (in Analogie zu en. hominids ‚Hominiden‘ für die Mitglieder der Familie Hominidae).[6]
Aufbau und Genom
Viren der Familie Nanoviridae sind unbehüllt, mit ikosaedrischer oder runder Geometrie, ihre Symmetrie hat Triangulationszahl T=1. Der Durchmesser beträgt etwa 18–19 nm. Das Kapsid besteht aus 60 Einheiten (Kapsomeren).[3][4]
Das Genom besteht aus mehreren Segmenten einzelsträngiger zirkulärer DNA mit einer Länge von jeweils 917 bis 1114 nt (Nukleotiden oder Basen), bei der Gattung Babuvirus geringfügig mehr bei Nanovirus.[6] Die Zahl der Segmente schwankt je nach Gattung mit (in der Regel) sechs bei Babuvirus[Anm. 1] und acht bei Nanovirus;[6] bei der nicht-zugewiesenen Spezies Coconut foliar decay virus (CFDV) scheint es drei Segmente zu geben, die Zugehörigkeit zur Familie wurde aber zuletzt angezweifelt.[7] Jedes der kleinen Viruspartikel enthält nur ein einziges Genomsegment,[6] so dass ausreichend Virionen in einer Wirtszelle zusammenkommen müssen, damit dessen volle Funktionalität gegeben ist. Aufgrund des segmentierten Genoms ist bei den Nanoviridae ein Reassortment bei Doppelinfektion möglich.
Die Segmente der Nanoviridae haben eine ähnliche Organisationsstruktur, einschließlich konservierter invertierter Wiederholungssequenzen (englisch inverted repeats, inverted repeat sequences), die potenziell eine Haarnadelstruktur (en. stem-loop structure, CR-SL)[Anm. 2] bilden und den Ursprung der Replikation darstellen. Eine weitere gemeinsame DNA-Region aller Segmente wird als CR-M (bei Babuvirus) bzw. CR-II (bei Nanovirus) bezeichnet.
Die Virionen haben einen einzigen Typ von Kapsidprotein (CP oder Cap) von etwa 19 kDa (Kilo-Dalton). Darüber hinaus werden mindestens 5–7 nicht-strukturelle Proteine kodiert.[6]
In der Regel kodieren die DNA-Segmente für jeweils ein einziges Protein.[6]
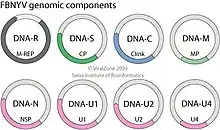
Zusätzlich sind viele Isolate von Nanoviridae mit einer unterschiedlichen Anzahl von separat eingekapselten, satellitenartigen sscDNAs (zyklische Einzelstrang-DNA), sog. Alphasatelliten, von etwa 1000–1100 nt assoziiert.[6] Mit diesen zusammen können bis zu etwa 11 verschiedene DNA-Segmente vorliegen. Die zusätzlichen satellitenartigen DNAs enthalten abweichende Haarnadelstrukturen (stem loops).
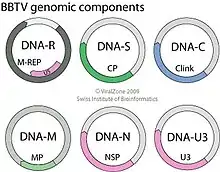
Die Viren der beiden Gattungen teilen einen Satz von fünf homologen DNA-Segmenten, die als DNA-R (kodierend für das Master Replication Initiator Protein, M-Rep), DNA-S (kodierend für das Kapsidprotein, CP), DNA-C (kodierend für das sog. Clink-Protein), DNA-M (kodierend für das Movement-Protein, MP) und DNA-N (kodierend für das sog. Nuclear Shuttle Protein – Kerntransport, NSP) bezeichnet werden.[6]
Einen zweiten Offenen Leserahmen (en. open reading frame, ORF) mit Bezeichnung U5, der vollständig im M-Rep verschachtelt ist, fand man unter den Spezies der Gattung Babuvirus beim Banana Bunchy Top Virus (BBTV) und beim Cardamom Bushy Dwarf Virus (CBDV), nicht aber beim Abaca Bunchy Top Virus (ABTV) – oder der Gattung Nanovirus.[6]
Replikationszyklus
Es wird angenommen, dass die Replikation von Nanoviriden im Zellkern durch einen Rolling-Circle-Replikationsmechanismus über eine dsDNA-Zwischenstufe (en. dsDNA intermediate) pro Segment erfolgt. Diese dienen dann als Vorlagen für die Transkription.[4][3]
Nach der Infektion einer Wirtszelle und Entfernung der Kapsidhülle wird die Synthese dieser viralen dsDNA-Zwischenstufe mit Hilfe von Wirts-DNA-Polymerase(n) gestartet über die kurzen DNA-Primers, die komplementär zur gemeinsamen Hauptregion (CR-SL) sind. Von diesen dsDNA-Zwischenstufen transkribiert die Wirts-[[{RNA-Polymerase]] dann (genauso wie von der genomischen dsDNA des Wirtszellkerns) mRNAs, die die viralen Proteine kodieren (en. DNA-templated transcription).
Die virale DNA-Replikation (als Schritt zur Erzeugung der Nachkommenschaft) wird durch das M-Rep-Protein initiiert. Sowohl für die Spezies Faba bean necrotic yellows virus (FBNYV) der Gattung Nanovirus als auch für die Typusspezies Banana bunchy top virus (BBTV) der Gattung Babuvirus gibt es experimentelle Hinweise, dass M-Rep die DNA-Spaltung (en. DNA cleavage) und auch den Nukleotidyltransfer katalysiert, und so die Replikation aller genomischen DNAs initiiert. M-Rep ist das einzige virale Protein, das für die Replikation der Nanoviriden essentiell ist. Von diesem angesehen werden alle anderen Replikationsproteine einschließlich der DNA-Polymerasen von der Wirtszelle bereitgestellt. Die virale DNA-Replikation wird verstärkt durch die Wirkung eines von Nanoviriden kodierten Zellzyklus-Modulator-Proteins (Clink).[3]
Das Virus verlässt die Wirtszelle durch per Export durch die Kernporen (en. nuclear pore export) und tubulusgesteuerte Virusbewegung (en. tubule-guided viral movement).[3][4]
Als natürliche Wirte dienen Pflanzen. Das Virus wird über einen Vektor (Blattläuse) übertragen.[3][4]
Systematik
Im Jahr 2021 hat das ICTV die neue Familie Metaxyviridae als Schwesterfamilie der Nanoviridae innerhalb der Ordnung Mulpavirales bestätigt. Die Metaxyviridae bestehen aus der einzigen Gattung Cofodevirus mit nur einer einzigen Spezies.[8][9]
Die vom ICTV bestätigten Taxonomie der Ordnung Mulpavirales und ist damit wie folgt:[3][1]
Ordnung Mulpavirales
- Familie Metaxyviridae
- Familie Nanoviridae
- Gattung Babuvirus
- Spezies Abaca bunchy top virus (ABTV)
- Spezies Banana bunchy top virus (BBTV), Typus – Büschelgipfelkrankheit der Banane
- Spezies Cardamom bushy dwarf virus (CBDV)
- Gattung Nanovirus
- Spezies Black medic leaf roll virus (BMLRV)
- Spezies Cow vetch latent virus (CvLV)
- Spezies Faba bean necrotic stunt virus (FBNSV)
- Spezies Faba bean necrotic yellows virus (FBNYV)
- Spezies Faba bean yellow leaf virus (FBYLV)
- Spezies Milk vetch dwarf virus (MDV)[10]
- Spezies Parsley severe stunt associated virus
- Spezies Pea necrotic yellow dwarf virus (PNYDV)
- Spezies Pea yellow stunt virus (PYSV)
- Spezies Sophora yellow stunt virus (SYSV)
- Spezies Subterranean clover stunt virus (SCSV), Typus
- Spezies „Grapevine-associated nanovirus“
- Spezies „Milk vetch chlorotic dwarf virus“ (MVCDV)
- Spezies „Picobiliphyte sp. MS584-5 nanovirus“
- Gattung Babuvirus
Helferviren
Die Mulpavirales können als Helferviren der Satellitenviren aus der Familie Alphasatellitidae dienen: die Nanoviridae für deren Unterfamilie Nanoalphasatellitinae, die Metaxyviridae für die Unterfamilie Petromoalphasatellitinae.[8]
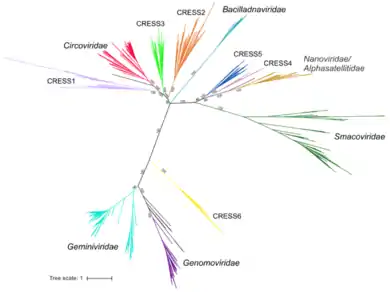
Evolution
Man nimmt an, dass die Alphasatellitidae sich aus den Nanoviridae entwickelt haben, und zwar zunächst die Unterfamilie Nanoalphasatellitinae, später nach einem Wechsel auf andere Helferviren (Geminiviridae) die Geminialphasatellitinae. Die Alphasatellitidae wären dann ebenfalls Mitglieder der Cressdnaviricota, wenn nicht gar der Mulpavirales. In diese Gruppe gehören offenbar zwei weitere Kladen als mutmaßliche Familien mit den provisorischen Bezeichnungen „CRESS4“ und „CRESS5“.[12][11][13] Jedoch sind derzeit noch weitere Forschungen nötig, bis eine offizielle Anerkennung erfolgen kann.
Anmerkungen
- acht bei Cardamom bushy dwarf virus (CBDV). Anisha Dayaram: Discovery of novel circular replication-associated protein encoding single-stranded DNA viruses in ecosystems using viral metagenomic approaches (PDF; 5,2 MB) Dissertation, University of Canterbury, New Zealand, 2014. Siehe Fig. 1.3
- Die stem-loop ist in vielen Darstellung als Ω- oder ߉-artige Ausstülpung außen am Genomring zu erkennen
Weblinks
- Nanoviridae. ICTV Report
- Stefan Boettcher, Allon Percus: Nature’s way of optimizing. In: Artificial Intelligence. 119, Nr. 1–2, 2000, S. 275–286. arxiv:cond-mat/9901351. doi:10.1016/S0004-3702(00)00007-2.
- Notes on Nanoviridae
- Nanoviridae. Viralzone
Einzelnachweise
- ICTV Master Species List 2020.v1. New MSL including all taxa updates since the 2019 release, ICTV, March 2021 (MSL #36)
- JE Thomas, B Gronenborn, RM Harding, B Mandal, I Grigoras, JW Randles, Y Sano, T Timchenko, HJ Vetten, HH Yeh, H Ziebell: ICTV Virus Taxonomy Profile: Nanoviridae. In: The Journal of General Virology, 12. Januar 2021. doi:10.1099/jgv.0.001544, PMID 33433311
- ICTV Report Nanoviridae.
- Viral Zone. ExPASy. Abgerufen am 2. April 2021.
- ICTV: Virus Taxonomy: 2014 Release. Abgerufen am 15. Juni 2015.
- John E. Thomas, Bruno Gronenborn, Robert M. Harding, Bikash Mandal, Ioana Grigoras, John W. Randles, Yoshitaka Sano, Tania Timchenko, H. Josef Vetten, Hsin-Hung Yeh, Heiko Ziebell: ssDNA Viruses > Nanoviridae. ICTV Virus Taxonomy Profile: Nanoviridae. In: Journal of General Virology, November 2020
- Unassigned species in family Nanoviridae
- Bruno Gronenborn, Arvind Varsani, J. W. Randles, H. J. Vetten, J. E. Thomas: Create a new subfamily (Petromoalphasatellitinae) with four new genera and six new species (Alphasatellitidae) (ZIP: DOCX, XLSX), Vorschlag 2020.001P.R.Petromoalphasatellitinae_nsf and das ICTV (angenommen)
- Coconut foliar decay virus (species). NCBI
- Simon Roux, François Enault, Gisèle Bronner, Daniel Vaulot, Patrick Forterre, Mart Krupovic: Chimeric viruses blur the borders between the major groups of eukaryotic single-stranded DNA viruses. In: Nature Commun, 4, 6. November 2013, S. 2700, doi:10.1038/ncomms3700. Siehe insbesondere Supplement 2 (XLS).
- Darius Kazlauskas, Arvind Varsani, Mart Krupovic: Pervasive Chimerism in the Replication-Associated Proteins of Uncultured Single-Stranded DNA Viruses. In: MDPI Viruses, Band 10, Nr. 4, Special Issue Viral Recombination: Ecology, Evolution and Pathogenesis, 10. April 2018, 187, doi:10.3390/v10040187; siehe auch Fig. S1 in Supplement S1 (ZIP: PDF, XLSX)
- Lele Zhao, Karyna Rosario, Mya Breitbart, Siobain Duffy: Chapter Three - Eukaryotic Circular Rep-Encoding Single-Stranded DNA (CRESS DNA) Viruses: Ubiquitous Viruses With Small Genomes and a Diverse Host Range. In: Advances in Virus Research, Band 103, 2019, S. 71–133, doi:10.1016/bs.aivir.2018.10.001, Epub 5. Dezember 2018. Insbes. siehe Fig. 3
- Hiroto Kaneko, Morgan Gaia, Rodrigo Hernández-Velázquez, Patrick Forterre, Curtis A. Suttle, Hiroyuki Ogata et al.: Eukaryotic virus composition can predict the efficiency of carbon export in the global ocean. In: iScience, Band 24, Nr. 1, 22. Januar 2021, 102002, Epub 29. Dezember 2020, doi:10.1016/j.isci.2020.102002, PMC 7811142 (freier Volltext), PMID 33490910; siehe Fig. 1(C).