SARS-CoV
SARS-CoV, auch als SARS-CoV-1 bezeichnet, ist ein Virus aus der Familie der Coronaviridae. Eine Infektion mit SARS-CoV verursacht das Schwere akute Atemwegssyndrom (SARS). Am 16. April 2003 gab die WHO bekannt, dass SARS-CoV die SARS-Pandemie 2002/2003 ausgelöst hat. Das Genom ist über 29,7 kbp groß und damit eines der umfangreichsten unter den RNA-Viren.[3]
Die als Name verwendete Abkürzung „SARS-CoV“ wurde aus der englische Wortgruppe „severe acute respiratory syndrome coronavirus“ abgeleitet.[A 1] Das SARS-CoV wird heute einer Klade zugeordnet,[4] die zur Spezies Severe acute respiratory syndrome-related coronavirus gehört, zu einer Art von Coronaviren, zu welcher weitere Viren bzw. Kladen gehören, z. B. SARS-CoV-2.[A 1] Der alternative Ausdruck SARS-CoV-1 wird vor allem deshalb anstelle von SARS-CoV verwendet, um das Virus besser von jenem anderen Virus zu unterscheiden, welches SARS-CoV-2 genannt wurde (und zur COVID-19-Pandemie geführt hat).[A 1]
Einzelne Isolate bzw. Virusstämme der Klade, welcher SARS-CoV angehört, werden beispielsweise „Tor2“ oder „PC4-227“ genannt; wobei empfohlen wird, das jeweilige Virus im Zweifelsfall durch zusätzliche Angaben genauer zu benennen.[A 1][4] In neueren Publikationen (2020) wurden die jeweiligen Virusisolate bspw. durch „SARS-CoV-1 Tor2 (AY274119.3)“[5] oder durch „SARS-CoV_PC4-227, AY613950.1“[4] genauer spezifiziert, wobei hier zusätzlich die jeweilige GenBank-Zugriffsnummer zu einer DNA-Sequenz des jeweiligen Virus-Genoms angegeben wurden.[A 1]
Einordnung und Benennung
Verschiedene Namen für das Virus, welches SARS verursachen kann („SARS-Coronavirus“,[6] „SCV“[7]), bezogen sich bis 2009 zugleich auf die gesamte taxonomische Spezies (damals „Severe acute respiratory syndrome coronavirus“), der dieses Virus angehörte.[8]
Es wurden Stämme von Coronaviren in anderen Säugetieren als dem Menschen gefunden, z. B. in Fledermäusen,[9] die dem SARS-verursachendem Virus beim Menschen ähneln. Seit 2009 heißt die Spezies Severe acute respiratory syndrome-related coronavirus (siehe dort für weitere Infos) und beherbergt (neben dem SARS-verursachenden Virus) auch weitere Stämme, die zwar verwandt sind, aber kein SARS beim Menschen auslösen.[8] In der folgenden Zeit wurden weitere Stämme entdeckt, ohne dass dies die Unterscheidung von SARS-verursachenden Viren und anderen Viren innerhalb dieser Spezies grundlegend erschwert hätte.[10][11][12]
Ein weiteres Virus, das vorläufig mit 2019-nCoV bezeichnet wurde,[13] eng mit SARS-CoV verwandt ist und zur gleichen Spezies gehört, wurde Anfang 2020 als SARS-CoV-2 bezeichnet.[A 2][14][4] Die Verwandtschaft zwischen SARS-CoV und SARS-CoV-2, dem zweiten Pandemie-verursachenden Virus in dieser Spezies, wird vor allem durch Genomanalysen kenntlich.[15]
In der gegenwärtig gültigen Liste für die Einordnung und Benennung von Viren, die durch das ICNV herausgegeben wurde, ist „SARS-CoV“ eine Kurzbezeichnung, die z. B. für zwei Virus-Isolate (bzw. Stämme) verwendet wird: „Tor2“ und „PC4-227“; der Ausdruck „SARS-CoV-1“ taucht dort nicht auf.[A 3][16][17]
Seitdem es die Idee gibt, das 2019 als Erreger einer Krankheit (COVID-19) neu in Erscheinung getretene Coronavirus „SARS-CoV-2“ zu nennen, gibt es auch die Idee, das lange vorher in Erscheinung getretene Coronavirus besser als „SARS-CoV-1“ zu bezeichnen (statt es „SARS-CoV“ zu nennen), um die Viren besser unterscheiden zu können; frühe Beispiele stammen aus Februar 2020.[A 4][18]
Eine schwierige Frage ist die nach der Einteilung von Viren, die zu einer einzelnen Spezies gehören, aber innerhalb dieser gruppiert werden müssen, da die Virus-Taxonomie (bzw. das ICTV) keine Ränge unterhalb der „species“ vorsieht.[4]
Zwei solcher Gruppen sind zum einen diejenigen Viren, die SARS auslösen können und zum anderen jene Viren, die COVID-19 auslösen können, wobei beide Gruppen jeweils mehrere verschiedene Isolate aufweisen (bspw. „BJ-01“ bis „BJ-04“ bei „SARS-CoV“[3]) und derselben Spezies angehören.[4] Die Arbeitsgruppe vom Internationalen Komitee für die Taxonomie der Viren, welche für die Coronaviridae zuständig ist („Coronaviridae Study Group of the International Committee on Taxonomy of Viruses“), bezeichnete die beiden Viren, „SARS-CoV“ (jenes Virus, das die Krankheit SARS hervorrufen kann) und „SARS-CoV-2“ (jenes Virus, das die Krankheit COVID-19 hervorrufen kann) als Kladen zugeordnete Viren, als „Schwesterkladen“ („sister clades“) innerhalb derselben Spezies („species“), Severe acute respiratory syndrome-related coronavirus.[4] Die Arbeitsgruppe gab die Empfehlung, künftig nicht nur die grundsätzliche Virus-Bezeichnung (also z. B. „SARS-CoV“ oder „SARS-CoV-2“) innerhalb von Publikationen anzugeben, sondern weitere Angaben hinsichtlich des jeweiligen Isolates bzw. Stammes zu machen (z. B. im Format Virus/Wirt/Ort/Isolat/Datum; ggf. Zugriffsnummer zur Genomsequenz).[4]
Bei vergleichenden Untersuchungen ist es praktikabel, sowohl die Gruppe von Krankheitserregern, als auch das konkret verwendete Isolat anzugeben; bei van Doremalen et al. (2020) wurden z. B. sowohl Ausdrücke für die beiden Gruppen („SARS-CoV-2“ und „SARS-CoV-1“), als auch konkrete Stammbezeichnungen („nCoV-WA1-2020“ und „Tor2“) sowie Zugriffsnummern für die jeweiligen Genomsequenzen („MN985325.1“ und „AY274119.3“) angegeben.[A 5][5]
In Publikationen werden die Kurzformen SARS-CoV, SARS-CoV-1 und SARS-CoV-2 zumeist als Namen verwendet, während die ausgeschriebenen Formen zumeist als Erklärungen für diese Namen zu finden sind. Bei Verwendung des Ausdrucks „SARS-CoV-1“ als Namen wurden unterschiedliche ausgeschriebene Formen zur Erklärung des Namens verwendet, solche mit Einbeziehung der Ziffer (die ersten beiden Beispiele) und solche ohne Einbeziehung der Ziffer (die letzten beiden Beispiele):
- „… data from severe acute respiratory syndrome coronavirus 1 (SARS-CoV-1), the virus responsible …“,[19]
- „… by its sister coronaviruses such as severe acute respiratory syndrome coronavirus-1 (SARS-COV-1) and …“,[20]
- „… candidates for severe acute respiratory syndrome (SARS-CoV-1) and provided a brief overview of …“,[21]
- „… Analysis of the first severe acute respiratory syndrome coronavirus (SARS-CoV-1) outcomes in 2003, and …“.[22]
Struktur
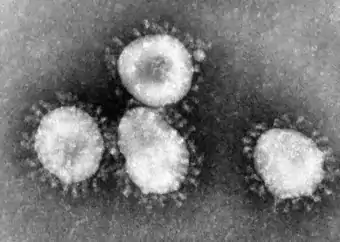
Das SARS-Coronavirus besitzt ein für die Virusfamilie typisches Genom und eine typische Struktur. Die Erbinformation ist in einem 29.751 Nukleotide langen einzelsträngigen RNA-Genom gespeichert.[23] Wie andere Coronaviren ist das Virion von SARS-CoV kugelförmig mit einem Durchmesser von rund 125 nm. Das Virus verfügt über vier Strukturproteine, Spike (S), Membrane (M), envelope (E) und nucleocapsid (N).
Herkunft
Aufgrund genetischer Untersuchungen von Betacoronaviren in Fledermäusen in Südostasien wurde eine evolutionäre Entwicklung des Virus bei der Übertragung über mehrere Wirte, von Fledermäusen bis hin zum Menschen, von einem Vorläufer des SARS-CoV bis zum humanpathogenen SARS-CoV postuliert.[25] Verschiedene Coronavirus-Isolate bei mehreren Fledermausarten und bei Larvenrollern wurden zusammen mit dem menschlichen SARS-CoV auf einen gemeinsamen Vorfahren zurückgeführt.[26] Eine genauere Untersuchung zu den Ursprüngen des SARS-CoV findet sich bei Xu et al. aus dem Jahr 2014 – der dem SARS-Virus am nächsten stehende Fledermaus-Coronavirus-Stamm ist Bt-SLCoV Rp3, dieser infiziert die Chinesische Hufeisennase (Rhinolophus sinicus).[27] Als Larvenroller infizierende Isolate wurden SARS-CoV SZ3 und SZ16 identifiziert.[28]
Im Jahr 2017 hatten Hu und Kollegen verschiedene Spezies von Fledermäusen aus einer Höhle in der chinesischen Provinz Yunnan untersucht. Sie wurden fündig in Hufeisennasen (Rhinolophidae) der Spezies R. sinicus, R. ferrumequinum, R. affinis und in Rundblattnasen (Hipposideridae) der Spezies Aselliscus stoliczkanus. Die Ergebnisse legten nahe, dass der bis dato dem SARS-CoV am nächsten stehende Vorläufer, das WIV16, eine Rekombinante aus drei Viren der Spezies SARS-assoziiertes Coronavirus (SARSr-CoV) ist (siehe Reassortment), die in Fledermäusen dieser Höhle vorkommen (WIV1, Rs4231 und Rs4081).[12] Es kann daher davon ausgegangen werden, dass auch bei Coronaviren eine Rekombination des Genoms zwischen verschiedenen Viren möglich ist, obwohl dieses unsegmentiert (monopartit) ist, d. h. aus einem einzigen Nukleinsäurestrang (hier ssRNA) besteht – im Gegensatz etwa zu Influenzaviren, deren Genom aus 8 Teilen besteht.
Übertragung
Die menschliche Krankheit SARS wird vor allem durch Aerosole in der Atemluft und Tröpfchen übertragen; das ist vor allem während der SARS-Pandemie 2002/2003 festgestellt worden.[29][30] Bei der Übertragung von Mensch zu Mensch nutzt das SARS-verursachende Virus, SARS-CoV-1, ein virales Oberflächenprotein (Spike-Protein), das an ein anderes Protein (ACE2-Rezeptor) auf der Oberfläche der jeweiligen menschlichen Wirtszelle bindet.[31]
Es gibt Hinweise, dass SARS einen zoonotischen Ursprung hat; z. B. traten die meisten Fälle in frühen Phasen der Epidemie der chinesischen Provinz Guangdong bei Lebensmittelhändlern auf, bei Personen, die Tiere schlachteten und mit lebenden Tieren und Fleisch Handel trieben sowie bei Personen, die Lebensmittel zubereiteten und servierten.[29][30] Auf welche Weise genau diese anfängliche Übertragung eines entsprechenden Virus vom Tier zum Menschen ablief, ist kaum bekannt.[32]
Die beiden in mancher Hinsicht ähnlichen Viren, SARS-CoV-1 und SARS-CoV-2, sind hinsichtlich der Übertragung verglichen worden.[33][34] Während SARS-CoV-1 vorrangig die unteren Atemwege befällt, also die Lunge, kann SARS-CoV-2 sowohl die oberen als auch die unteren Atemwege befallen; SARS-CoV-2 kann sich bspw. unabhängig von der Lunge im Rachen vermehren.[33][34] Diese unterschiedlich stark ausgeprägte Bevorzugung von verschiedenen Geweben durch SARS-CoV-1 und SARS-CoV-2 dürfte auch die Unterschiede bei der Übertragung auf Kontaktpersonen mit sich bringen.[33][34]
Die effektive Vermehrung von SARS-CoV-2 im Rachen könnte dazu führen, dass Personen ohne Krankheitsanzeichen die Infektion weitergeben.[33][34] Bei SARS-CoV-1 ist die effektive Vermehrung vermutlich auf die Lunge begrenzt und die Weitergabe der Infektion begann bei der SARS-Pandemie häufig erst dann, wenn bei einem Überträger bereits Symptome der Krankheit vorhanden waren, wodurch dieser möglicherweise unmittelbarer erkannt werden konnte, als dass das bei SARS-CoV-2 der Fall ist.[A 6]
Eine Hypothese dazu,[34] wie sich der erweiterte Tropismus (hin zum Rachen) von SARS-CoV-2 gegenüber SARS-CoV-1 erklären lassen könnte, bezieht sich darauf, dass im Spike-Protein von SARS-CoV-2 eine spezielle Spaltstelle vorhanden ist (polybasische Furin-Typ-Spaltstelle an der S1–S2-Verbindung), die es beim Spike-Protein von SARS-CoV-1 nicht gibt.[35] Es wurde gezeigt, dass es durch das Einfügen einer ähnlichen Spaltstelle in das Spike-Protein von SARS-CoV-1 (also durch Insertion einer polybasischen Spaltstelle in die S1–S2-Region) zu einer moderaten, aber erkennbaren Erhöhung der Fusionsaktivität kommt; das könnte zu einem verstärkten Eintritt von Viren in das jeweilige Gewebe führen, gerade bei geringer Expressionsdichte von ACE2.[36]
Studien im Gefolge der SARS-Pandemie ergaben, dass die meisten infizierten Personen relativ wenige Kontakte infizierten, während es durch manche Personen zu einem Superspreading kam.[37][38] In einer Studie zu einem Flug mit 112 Passagieren wurden 16 Erkrankungen dokumentiert, welche infolge eines bereits erkrankten Passagiers entstanden. Dabei wurde eine Häufung der Ansteckungen bis zu 3 Reihen vor dem Indexpatienten, in einem Abstand bis zu 2,30 m, festgestellt.[39]
Umweltstabilität
Unter Laborbedingungen konnte hinsichtlich der Tenazität nachgewiesen werden, dass verdünntes Sputum und verdünnter Stuhl mindestens 72 Stunden lang eine niedrige Infektiosität aufweisen. Auf Flächen unterschiedlicher Materialien konnte eine Infektionsfähigkeit des Virus nach rund 72 bis 96 Stunden nachgewiesen werden. Die Infektiosität nimmt bei Raumtemperatur nach rund zwei Stunden ab. Das Virus wird durch Erhitzen über 75 °C für 30 Minuten (alternativ: über 67 °C für 60 Minuten oder über 56 °C für 90 Minuten) sowie durch 60-minütige UV-Strahlung (Wellenlänge: 260 nm, Intensität: 90 µW/cm², Gesamtdosis: 324 mWs/cm²) vollständig inaktiviert.[40] SARS-CoV wird in Abwesenheit von anderen Proteinen bei 56 °C innerhalb von 30 Minuten mit einer Keimzahlreduktion auf das 10−5-fache inaktiviert werden.[41] In Anwesenheit von höheren Proteinkonzentrationen erfolgt eine Keimzahlreduktion auf das 10−5-fache bei 60 °C innerhalb von 30 Minuten.[41] SARS-CoV ist als behülltes Virus empfindlich gegen alkoholische Desinfektionsmittel mit einer Keimzahlreduktion auf das 10-2,78-fache nach 30 Sekunden.[41] Eine Inaktivierung in verdünnter Essigsäure (Weinessig) erfolgt mit einer Keimzahlreduktion auf das 10−3-fache nach 60 Sekunden.[41] Aldehyd-basierte Desinfektionsmittel (mit Formaldehyd oder Glutaraldehyd) führen zu einer Inaktivierung mit einer Keimzahlreduktion auf das 10−3-fache nach 120 Sekunden.[41] Mit Glucoprotaminlösungen erfolgt eine Inaktivierung mit einer Keimzahlreduktion auf das 10-1,68-fache nach 120 Sekunden.[41]
Immunologie und Impfstoffforschung
Bei einem Virusstamm aus der chinesischen Provinz Guangdong konnte das Auftreten infektionsverstärkender Antikörper nachgewiesen werden, welche mit dem ACE2-Rezeptor interagieren.[42] Bei drei genesenen Patienten der SARS-Pandemie konnte noch neun bis elf Jahre nach ihrer Infektion eine Immunantwort mittels T-Gedächtniszellen und zytotoxischer T-Zellen nachgewiesen werden. Diese waren gegen die Strukturproteine M und N gerichtet. Eine Kreuzreaktivität gegen das strukturverwandte MERS-CoV konnte nicht nachgewiesen werden.[43]
Im Jahr 2010 wurde im Tierversuch an Mäusen und Goldhamstern ein Impfstoff aus inaktiviertem SARS-CoV getestet. Es ließ sich eine begrenzte Immunität der Tiere nachweisen, die jedoch rasch abnahm. Die Mauspopulation verfügte nach achtzehn Wochen über keine Immunität mehr. Bei den Hamstern zeigte sich eine begrenzte Immunität noch achtzehn Wochen nach der zweiten Impfdosis.[44] Im Jahr 2012 wurde eine Studie veröffentlicht, welche neben inaktiviertem SARS-CoV auch Impfstoffe bestehend aus Teilkomponenten an einem Mausmodell testete. Alle Impfstoffe lösten bei den Versuchstieren die Bildung neutralisierender Antikörper aus. Alle Versuchstiere zeigten jedoch nach Exposition mit dem SARS-Virus eine Autoimmunreaktion der Lungen, welche von den Forschern auf eine durch die Impfung hervorgerufene, überschießende Immunreaktion auf das Virus zurückgeführt wurde.[45]
Wirtsspektrum
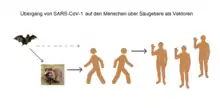
Da der ACE2-Rezeptor bei Katzenartigen und Menschen sehr ähnlich ist, ist es dem Virus auch möglich, außer Larvenrollern auch Hauskatzen und Frettchen zu infizieren.[46][7] Der Übergang von Fledermäusen auf den Menschen erfolgte wahrscheinlich über den Marderhund als Überträger.[47]
Forschungsgeschichte
Ende März 2003 wurde SARS-CoV erstmals im Rahmen der Forschung zur SARS-Pandemie, in mehreren Labors in verschiedenen Ländern, isoliert.[48] Mitte Mai erfolgte per Tierexperiment der endgültige Beweis, dass SARS-CoV die Erkrankung auslöst.[49]
Meldepflicht
In der Schweiz ist der positive und negative laboranalytische Befund zu einem SARS-Erreger für Laboratorien meldepflichtig und zwar nach dem Epidemiengesetz (EpG) in Verbindung mit der Epidemienverordnung und Anhang 3 der Verordnung des EDI über die Meldung von Beobachtungen übertragbarer Krankheiten des Menschen. In Deutschland ist der direkte und indirekte Nachweis des „Severe-Acute-Respiratory-Syndrome-Coronavirus (SARS-CoV)“ seit dem 23. Mai 2020 gemäß § 7 Abs. 1 Nr. 44a des Infektionsschutzgesetzes (IfSG) für Labore namentlich meldepflichtig, sofern der Nachweis auf eine akute Infektion hindeutet.
Weblinks
Anmerkungen
- Siehe Abschnitt #Einordnung und Benennung; die Einordnung und Benennung des Virus SARS-CoV wird dort genauer erläutert und belegt.
- Die Benennung als „SARS-CoV-2“ wurde als Vorabdruck (Preprint) von Gorbalenya et al. (7. Feb. 2020; biorxiv.org vorgeschlagen und in einer Veröffentlichung durch die „Coronaviridae Study Group of the ICTV“ (2. März 2020; PMID 32123347; doi:10.1038/s41564-020-0695-z) bestätigt.
- Von einer Website des ICNV (talk.ictvonline.org konnte eine Excel-Datei mit dem Namen „VMR 010820 MSL35.xlsx“ heruntergeladen werden, die Angaben zur taxonomischen Einteilung und zu Benennungen von Viren enthält. Angaben auf der Website: „Virus Metadata Repository: version August 1, 2020; MSL35“; „August 1, 2020 release of the Virus Metadata Repository (VMR) file containing updated data on new species ratified in March, 2020 and appearing in taxonomy release 2019, MSL35.“ Download der Datei: 6. April 2021.
- Ein Beispiel für eine frühe, gleichzeitige Anwendung der Ausdrücke „SARS-CoV-2“ und „SARS-CoV-1“ ist eine Publikation von Cordes & Heim (2020; PMID 32143123, doi:10.1016/j.jcv.2020.104305), die am 4. Februar eingereicht und am 28. Februar 2020 erstmals (online) veröffentlicht wurde.
- In van Doremalen et al. (2020; PMID 32182409): „SARS-CoV-2 nCoV-WA1-2020 (MN985325.1) and SARS-CoV-1 Tor2 (AY274119.3) were the strains used.“; Übersetzung: „SARS-CoV-2 nCoV-WA1-2020 (MN985325.1) und SARS-CoV-1 Tor2 (AY274119.3) waren die verwendeten Stämme.“
- Man kann die beiden Seuchen, die SARS-Pandemie 2002/2003 und die COVID-19-Pandemie nur schwer vergleichen, da sie nicht gleichzeitig laufen.
Einzelnachweise
- Thomas E Novotny, Emilio Mordini, Ruth Chadwick, J. Martin Pedersen, Fabrizio Fabbri: Bioethical Implications of Globalization: An International Consortium Project of the European Commission. In: PLoS Medicine. Band 3, Nr. 2, 24. Januar 2006, ISSN 1549-1676, S. e43, doi:10.1371/journal.pmed.0030043, PMID 16420098, PMC 1351155 (freier Volltext).
- Shengli Bi, E’de Qin, Zuyuan Xu, Wei Li, Jing Wang: Complete Genome Sequences of the SARS-CoV: the BJ Group (Isolates BJ01-BJ04). In: Genomics, Proteomics & Bioinformatics. Band 1, Nr. 3, August 2003, S. 180–192, doi:10.1016/S1672-0229(03)01023-4, PMID 15629030, PMC 5172409 (freier Volltext).
- Coronaviridae Study Group of the International Committee on Taxonomy of Viruses, A.E. Gorbalenya, S.C. Baker et al.: The species Severe acute respiratory syndrome-related coronavirus: classifying 2019-nCoV and naming it SARS-CoV-2. In: Nature Microbiology. Band 5, Nr. 4, April 2020, ISSN 2058-5276, S. 536–544, doi:10.1038/s41564-020-0695-z, PMID 32123347, PMC 7095448 (freier Volltext) – (nature.com).
- Neeltje van Doremalen, Trenton Bushmaker, Dylan H. Morris, Myndi G. Holbrook, Amandine Gamble: Aerosol and Surface Stability of SARS-CoV-2 as Compared with SARS-CoV-1. In: New England Journal of Medicine. Band 382, Nr. 16, 16. April 2020, S. 1564–1567, doi:10.1056/NEJMc2004973, PMID 32182409, PMC 7121658 (freier Volltext).
- Li W1, Shi Z, Yu M, Ren W, Smith C, Epstein JH, Wang H, Crameri G, Hu Z, Zhang H, Zhang J, McEachern J, Field H, Daszak P, Eaton BT, Zhang S, Wang LF: Bats are natural reservoirs of SARS-like coronaviruses. In: Science. 28. Oktober 2005, Zweiter Satz, doi:10.1126/science.1118391, PMID 16195424 (englisch): “SARS coronavirus (SARS-CoV)”
- Byron E. E. Martina, Bart L. Haagmans, Thijs Kuiken, Ron A. M. Fouchier, Guus F. Rimmelzwaan, Geert van Amerongen, J. S. Malik Peiris, Wilina Lim, Albert D. M. E. Osterhaus: SARS virus infection of cats and ferrets, in: Nature Band 425, S. 915, 30. Oktober 2003, doi:10.1038/425915a, PMID 14586458, PMC 7094990 (freier Volltext)
- ICTV Taxonomy history: Severe acute respiratory syndrome-related coronavirus. In: ICTV-Homepage. International Committee on Taxonomy of Viruses (ICTV), abgerufen am 7. Mai 2020 (englisch).
Und zugehöriges Proposal: 2008.085-126V. (PDF; 175 kB) In: ICTV-Homepage. International Committee on Taxonomy of Viruses (ICTV), S. 23 [2008.105V], 34 [2008.119V] und 36 [2008.121V], abgerufen am 7. Mai 2020 (englisch). - Susanna K. P. Lau, Patrick C. Y. Woo, Kenneth S. M. Li, Yi Huang, Hoi-Wah Tsoi, Beatrice H. L. Wong, Samson S. Y. Wong, Suet-Yi Leung, Kwok-Hung Chan, and Kwok-Yung Yuen: Severe acute respiratory syndrome coronavirus-like virus in Chinese horseshoe bats. In: Proc Natl Acad Sci U S A. 27. September 2005, Erster Satz, doi:10.1073/pnas.0506735102, PMID 16169905, PMC 1236580 (freier Volltext) – (englisch): “severe acute respiratory syndrome coronavirus (SARS-CoV)”
- ICTV Master Species List 2018b.v2. MSL #34, März 2019
- Ben Hu, Lei-Ping Zeng, Xing-Lou Yang, Xing-Yi Ge, Wei Zhang, Bei Li, Jia-Zheng Xie, Xu-Rui Shen, Yun-Zhi Zhang, Ning Wang, Dong-Sheng Luo, Xiao-Shuang Zheng, Mei-Niang Wang, Peter Daszak, Lin-Fa Wang, Jie Cui, Zheng-Li Shi: Discovery of a rich gene pool of bat SARS-related coronaviruses provides new insights into the origin of SARS coronavirus. In: PLOS Pathogens. Band 13, Nr. 11, 30. November 2017, ISSN 1553-7374, S. e1006698, doi:10.1371/journal.ppat.1006698, PMID 29190287, PMC 5708621 (freier Volltext).
- Novel Coronavirus (2019-nCoV). (PDF; 1,0 MB) Situation Report – 22. WHO, 11. Februar 2020, abgerufen am 13. Februar 2020.
- Alexander E. Gorbalenya, Susan C. Baker, Ralph S. Baric, Raoul J. de Groot, Christian Drosten, Anastasia A. Gulyaeva, Bart L. Haagmans, Chris Lauber, Andrey M. Leontovich, Benjamin W. Neuman, Dmitry Penzar, Stanley Perlman, Leo L. M. Poon, Dmitry Samborskiy, Igor A. Sidorov, Isabel Sola, John Ziebuhr: Severe acute respiratory syndrome-related coronavirus: The species and its viruses – a statement of the Coronavirus Study Group. In: bioRxiv. 11. Februar 2020, bioRxiv: 10.1101/2020.02.07.937862v1 (Preprint-Volltext), S. 1–20, doi:10.1101/2020.02.07.937862 (englisch).
- Kristian G. Andersen, Andrew Rambaut, W. Ian Lipkin, Edward C. Holmes, Robert F. Garry: The proximal origin of SARS-CoV-2. In: Nature Medicine. Band 26, Nr. 4, April 2020, ISSN 1546-170X, S. 450–452, doi:10.1038/s41591-020-0820-9, PMID 32284615, PMC 7095063 (freier Volltext) – (englisch, Published: 17 March 2020 (online)).
- Virus Metadata Repository: version August 1, 2020; MSL35. Abgerufen am 6. April 2021 (englisch).
- ICTV: ICTV Taxonomy history: Severe acute respiratory syndrome-related coronavirus, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- Anne K. Cordes, Albert Heim: Rapid random access detection of the novel SARS-coronavirus-2 (SARS-CoV-2, previously 2019-nCoV) using an open access protocol for the Panther Fusion. In: Journal of Clinical Virology. Band 125, April 2020, ISSN 1873-5967, doi:10.1016/j.jcv.2020.104305, PMID 32143123, PMC 7129486 (freier Volltext).
- Suma Radhakrishnan, Hafees Abdullah Perumbally, Sai Surya, Mohammed Shareef Ponneth: Guidelines for Surgical Tracheostomy and Tracheostomy Tube Change During the COVID-19 Pandemic: A Review Article. In: Indian Journal of Otolaryngology and Head and Neck Surgery: Official Publication of the Association of Otolaryngologists of India. Band 72, Nr. 3, September 2020, ISSN 2231-3796, S. 398–401, doi:10.1007/s12070-020-01893-y, PMID 32719738, PMC 7306934 (freier Volltext).
- Jawaria Rahman, Abilash Muralidharan, Sohail J. Quazi, Hajra Saleem, Safeera Khan: Neurological and Psychological Effects of Coronavirus (COVID-19): An Overview of the Current Era Pandemic. In: Cureus. Band 12, Nr. 6, 5. Juni 2020, ISSN 2168-8184, S. e8460, doi:10.7759/cureus.8460, PMID 32528783, PMC 7282368 (freier Volltext).
- Chih-Yin Lin, Chun-An Yao: Potential Role of Nrf2 Activators with Dual Antiviral and Anti-Inflammatory Properties in the Management of Viral Pneumonia. In: Infection and Drug Resistance. Band 13, 2020, ISSN 1178-6973, S. 1735–1741, doi:10.2147/IDR.S256773, PMID 32606823, PMC 7295331 (freier Volltext).
- Andrea Pozzer, Francesca Dominici, Andy Haines, Christian Witt, Thomas Münzel: Regional and global contributions of air pollution to risk of death from COVID-19. In: Cardiovascular Research. Band 116, Nr. 14, 1. Dezember 2020, ISSN 1755-3245, S. 2247–2253, doi:10.1093/cvr/cvaa288, PMID 33236040, PMC 7797754 (freier Volltext).
- Marco A. Marra, Steven J. M. Jones1, Caroline R. Astell, Robert A. Holt: The Genome Sequence of the SARS-Associated Coronavirus. Science, 30. Mai 2003:Vol. 300, Issue 5624, S. 1399–1404 doi:10.1126/science.1085953
- Gouilh, Puechmaille SJ, Gonzalez JP, Teeling E, Kittayapong P, Manuguerra JC: SARS-Coronavirus ancestor’s foot-prints in South-East Asian bat colonies and the refuge theory. In: Infect Genet Evol., Oktober 2011, 11(7), S. 1690–1702. doi:10.1016/j.meegid.2011.06.021, PMID 21763784
- SK Lau, KS Li, Y Huang, CT Shek, H Tse: Ecoepidemiology and complete genome comparison of different strains of severe acute respiratory syndrome-related Rhinolophus bat coronavirus in China reveal bats as a reservoir for acute, self-limiting infection that allows recombination events. In: J Virol., März 2010, 84(6), S. 2808–2019. doi:10.1128/JVI.02219-09. Epub 13. Januar 2010. PMID 20071579
- Dezhong Xu, Huimin Sun, Haixia Su, Lei Zhang, Jingxia Zhang, Bo Wang, Rui Xu: SARS coronavirus without reservoir originated from an unnatural evolution, experienced the reverse evolution, and finally disappeared in the world. In: Chinese Medical Journal, Band 127, Nr. 13, 5. Juli 2014, S. 2537–2542, doi:10.3760/cma.j.issn.0366-6999.20131328
- Ming Wang et al.: SARS-CoV Infection in a Restaurant from Palm Civet. In: Emerg Infect Dis., 11(12), Dezember 2005, S. 1860–1865, doi:10.3201/eid1112.041293, PMC 3367621 (freier Volltext), PMID 16485471
- Robert F. Breiman, Meirion R. Evans, Wolfgang Preiser, James Maguire, Alan Schnur: Role of China in the Quest To Define and Control Severe Acute Respiratory Syndrome. In: Emerging Infectious Diseases. Band 9, Nr. 9, September 2003, ISSN 1080-6040, S. 1037–1041, doi:10.3201/eid0909.030390, PMID 14519236, PMC 3016762 (freier Volltext).
- World Health Organization (Hrsg.): Consensus document on the epidemiology of severe acute respiratory syndrome (SARS). 2003 (who.int [abgerufen am 14. April 2021]).
- W. Li, M. J. Moore, N. Vasilieva, J. Sui, S. K. Wong, M. A. Berne, M. Somasundaran, J. L. Sullivan, K. Luzuriaga, T. C. Greenough, H. Choe, M. Farzan: Angiotensin-converting enzyme 2 is a functional receptor for the SARS coronavirus. In: Nature, Band 426, Nummer 6965, November 2003, S. 450–454, doi:10.1038/nature02145, PMID 14647384.
- Danuta M. Skowronski, Caroline Astell, Robert C. Brunham, Donald E. Low, Martin Petric: Severe acute respiratory syndrome (SARS): a year in review. In: Annual Review of Medicine. Band 56, 2005, ISSN 0066-4219, S. 357–381, doi:10.1146/annurev.med.56.091103.134135, PMID 15660517.
- Umfassende Daten im Fachmagazin Nature erschienen. Charité Universitätsmedizin Berlin, Pressemitteilung
- Roman Wölfel, Victor M. Corman, Wolfgang Guggemos, Michael Seilmaier, Sabine Zange: Virological assessment of hospitalized patients with COVID-2019. In: Nature. Band 581, Nr. 7809, 28. Mai 2020, ISSN 0028-0836, S. 465–469, doi:10.1038/s41586-020-2196-x.
- Christian Drosten, Stephan Günther, Wolfgang Preiser, Sylvie van der Werf, Hans-Reinhard Brodt: Identification of a Novel Coronavirus in Patients with Severe Acute Respiratory Syndrome. In: New England Journal of Medicine. Band 348, Nr. 20, 15. Mai 2003, ISSN 0028-4793, S. 1967–1976, doi:10.1056/NEJMoa030747.
- Sandrine Belouzard, Victor C. Chu, Gary R. Whittaker: Activation of the SARS coronavirus spike protein via sequential proteolytic cleavage at two distinct sites. In: Proceedings of the National Academy of Sciences. Band 106, Nr. 14, 7. April 2009, ISSN 0027-8424, S. 5871–5876, doi:10.1073/pnas.0809524106, PMID 19321428, PMC 2660061 (freier Volltext).
- Zhuang Shen et al.: Superspreading SARS Events, Beijing, 2003. In: Emerging infectious diseases, 10, Nr. 2 (2004), S. 256–260, doi:10.3201/eid1002.030732. PMC 3322930 (freier Volltext).
- Richard A. Stein: Super-Spreaders in Infectious Diseases. In: International Journal of Infectious Diseases 15, Nr. 8 (2011), doi:10.1016/j.ijid.2010.06.020, S. e510-e513.
- Sonja J. Olsen et al.: Transmission of the Severe Acute Respiratory Syndrome on Aircraft. In: N Engl J Med, 18. Dezember 2003, 349, S. 2416–2422, doi:10.1056/NEJMoa031349, PMID 14681507.
- SM Duan, XS Zhao, RF Wen, JJ Huang, GH Pi: Stability of SARS coronavirus in human specimens and environment and its sensitivity to heating and UV irradiation. In: Biomed Environ Sci., September 2003, 16(3), S. 246–255. PMID 14631830
- H. F. Rabenau, J. Cinatl, B. Morgenstern, G. Bauer, W. Preiser, H. W. Doerr: Stability and inactivation of SARS coronavirus. In: Medical microbiology and immunology, Band 194, Nummer 1–2, Januar 2005, S. 1–6, doi:10.1007/s00430-004-0219-0, PMID 15118911, PMC 7086689 (freier Volltext).
- Garry J. Nabel et al.: Evasion of antibody neutralization in emerging severe acute respiratory syndrome coronaviruses. In: PNAS, 18. Januar 2005, doi:10.1073/pnas.0409065102
- OW Ng, A Chia, AT Tan, RS Jadi, HN Leong, A Bertoletti, YJ Tan: Memory T cell responses targeting the SARS coronavirus persist up to 11 years post-infection. In: Vaccine, 12. April 2016;34(17), S. 2008–2014. doi:10.1016/j.vaccine.2016.02.063. Epub 5. März 2016
- Kanta Subbarao: Immunogenicity and Protective Efficacy in Mice and Hamsters of a β-Propiolactone Inactivated Whole Virus SARS-CoV Vaccine. Viral Immunology.Oct 2010.509-519. doi:10.1089/vim.2010.0028
- RB Couch: Immunization with SARS coronavirus vaccines leads to pulmonary immunopathology on challenge with the SARS virus. In: PLoS One, 2012, 7(4), S. e35421. Epub 2012 Apr 20. doi:10.1371/journal.pone.0035421
- Helen Thompson: A cat appears to have caught the coronavirus, but it’s complicated. ScienceNews, 31, März 2020
- James D. Cherry, Paul Krogstad: SARS: The First Pandemic of the 21st Century. nature.com
- P. A. Rota, M. S. Oberste u. a.: Characterization of a novel coronavirus associated with severe acute respiratory syndrome. In: Science. Band 300, Nummer 5624, Mai 2003, S. 1394–1399, doi:10.1126/science.1085952, PMID 12730500.
- R. A. Fouchier, T. Kuiken u. a.: Aetiology: Koch’s postulates fulfilled for SARS virus. In: Nature. Band 423, Nummer 6937, Mai 2003, S. 240, doi:10.1038/423240a, PMID 12748632
