Hepatitis-C-Virus
Das Hepatitis-C-Virus ist ein behülltes, einzelsträngiges RNA-Virus mit positiver Polarität (ss(+)RNA) und ist der Erreger der Hepatitis C. Es ist das einzige bislang bekannte RNA-Virus, das (ohne ein Retrovirus zu sein) eine chronische Infektionskrankheit verursachen kann. Zu Diagnostik, Erkrankungen und Prävention siehe Hepatitis C.
| Hepatitis-C-Virus | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
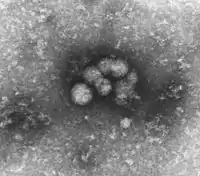
HCV-Virionen aus Serum | ||||||||||||||||||
| Systematik | ||||||||||||||||||
| ||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||
| ||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||
| Hepacivirus C | ||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||
| HCV | ||||||||||||||||||
| Links | ||||||||||||||||||
|
Obwohl man seit den ersten Berichten 1974 über ein infektiöses Agens als Erreger der damals so genannten „Non-A-Non-B-Hepatitis“ wusste, war das Virus lange Zeit nicht identifiziert. Erst nach der erstmaligen Anwendung der Klonierung von Genomfragmenten aus dem Serum eines künstlich mit HCV infizierten Schimpansen gelang 1989 die Entdeckung und 1990 die Sequenzierung des dann Hepatitis-C-Virus genannten Erregers. Es ist auch das erste Virus, dessen Genomsequenz und Virusproteine einem umfassenden Patentschutz unterliegen, was die Entwicklung neuer Testverfahren erheblich erschwerte. Patenthalter ist, nach der Übernahme der Chiron Corporation im Jahr 2006, der Pharmakonzern Novartis.
Das Hepatitis-C-Virus (HCV) gehört zusammen mit dem Hepatitis-B-Virus (HBV), dem Epstein-Barr-Virus (EBV), dem humanen Papillomvirus (HPV), Humanen T-lymphotropen Virus 1 (HTLV-1) und dem Humanen Herpesvirus 8 (HHV-8, auch Kaposi-Sarkom-Herpesvirus, KSHV) zu den humanen Viren, die Krebserkrankungen auslösen können. Es wird geschätzt, dass diese Viren weltweit für 10 bis 15 Prozent aller Krebserkrankungen verantwortlich sind.[3]
Für die Entdeckung des Virus wurde 2020 den Wissenschaftlern Harvey J. Alter, Michael Houghton und Charles M. Rice der Nobelpreis für Physiologie oder Medizin zuerkannt.[4]
Molekularbiologie
Systematik
Durch Sequenzvergleiche und der Homologie zu anderen Virusproteinen wurde das HCV in die Familie der Flaviviridae eingruppiert, wobei zunächst die Einordnung in die Gattungen Flavivirus oder Pestivirus nicht eindeutig war. Phylogenetisch scheint das HCV näher mit den sonst nur tierpathogenen Pestiviren verwandt zu sein, dies legen Hydrophobizitätsprofile der Proteine wie auch die Genomorganisation nahe. Es erschien taxonomisch sinnvoll, das HCV als einzigen Vertreter in ein neues Genus Hepacivirus zu gruppieren. Nahe mit HCV verwandt, jedoch noch keiner Gattung oder anderen Gattungen zugeordnet sind die GB-Viren (GB-Virus A und B bei Neuweltaffen und Tamarinen), besonders das auch beim Menschen vorkommende, jedoch apathogene GB-Virus C.
Variabilität und Subtypen
Die Variabilität des HCV ist auch im Vergleich zu anderen einzelsträngigen RNA-Viren verhältnismäßig hoch. So finden sich auf Genomebene beim Vergleich verschiedener Isolate bis zu 40 % Abweichungen. Daher wird das HCV in sieben Genotypen (1–7) unterteilt, wobei ein einheitlicher Genotyp vereinbarungsgemäß mindestens 72 % Übereinstimmung auf Aminosäureebene besitzen muss. Diese Genotypen werden weiter in 30 Subtypen (1a, 1b, 3a usw.) untergliedert, wobei die Homologie auf Aminosäureebene 73 bis 86 % innerhalb der Subtypen beträgt.
Aufgrund der Leseungenauigkeit der viralen RNA-Polymerase mit einer Mutationsrate von bis pro Nukleotid und Replikation, entwickeln sich in einem infizierten Wirt weitere Abweichungen von der ursprünglichen Sequenz, die man als Quasispezies bezeichnet. Die schnelle Entstehung dieser Quasispezies wird mit der Fähigkeit des HCV in Verbindung gebracht, eine chronische Infektion zu verursachen. Dabei weichen die ständig auftauchenden Varianten (ähnlich der HIV-Infektion) dem Zugriff des Immunsystems aus; dieser Mechanismus wird auch Immunevasion genannt.
Struktur
Das einzelsträngige RNA-Genom (als Säure negativ geladen) liegt im Virion an das Coreprotein (basisch, positiv geladen) angelagert vor. Das Coreprotein bildet höchstwahrscheinlich kein klassisches Kapsid mit einer festen Symmetrie (z. B. ikosaedrisch). Freie Kapside konnten bislang nicht glaubhaft dargestellt oder zusammengelagert werden.
Das Coreprotein ist von innen in der Virushülle verankert und über eine Transmembran-Helix an den Komplex der beiden Hüllproteine E1/E2 (envelope engl. Hülle) gebunden. Der transmembrane Abschnitt des HCV-Coreproteins entspricht wahrscheinlich dem (größeren) M-(Matrix)-Protein bei Viren der Gattung Flavivirus (z. B. Gelbfiebervirus, FSME-Virus). Dieses M-Protein wird vom Core-Protein abgespalten und kleidet die Virushülle von innen aus; beim HCV unterbleibt offenbar diese Abspaltung eines M-Proteins.
Das HCV ist äußerst schwierig im Elektronenmikroskop darzustellen, bisher hat keine Publikation ein endgültig bewiesenes Bild zeigen können. Manche Abbildungen zeigen Partikel einer anzunehmenden Größe von ca. 50 nm, die nach bestimmten Reinigungsverfahren sichtbar werden (siehe oben). Diese schwierige Visualisierung wahrscheinlich aufgrund einer strukturellen Instabilität und Empfindlichkeit gegenüber normalen Präparationsmethoden des HCV ist ein Grund für seine sehr späte Entdeckung trotz intensiver Suche.
Im Genom des HCV findet sich ein großer offener Leserahmen (open reading frame, ORF), von dem ein einziges Polyprotein von 3008 bis 3037 Aminosäuren Länge abgelesen wird. Dieses Protein wird durch zelluläre Proteasen (Signalase) der ER-Membran und virale Proteasen noch während der Translation in die Strukturproteine (Core, E1 und E2) sowie die Nicht-Strukturproteine (NS2 bis NS5) aufgespalten. Dies bedeutet, dass außer dem vorhandenen Virusgenom keine weitere messenger-RNA transkribiert wird. Die Tabelle führt diese Proteine mit ihrer (teilweise noch unklaren) Funktion, der Molmasse und der Aminosäure-Position auf dem Polyprotein auf:
| Protein | Größe (kDa) | Position | Funktion |
|---|---|---|---|
| Core | 21 (19) | 1-190 | Bindung der RNA und des E1/E2-Heterodimers
Oligomerisierung und Lipidbindung, Aktivierung und Hemmung zellulärer Gene (Leucin-Zipper-Motiv) |
| E1 | 31-37 | 191-383 | Glykosyliertes Membranprotein, virales Hüllprotein, E1/E2-Heterodimerisierung |
| E2 | 61-72 | 384-746 | Glykosyliertes Membranprotein, virales Hüllprotein, Bindung an zelluläre Rezeptoren (CD81, SRB1),
Hypervariable Region (HVR1), Antikörper-bindende Region |
| p7 | 7 | 747-809 | Membranprotein, möglicherweise Ionenkanal |
| NS2 | 21-23 | 810-1026 | Zink-Metalloprotease, Autoprotease in Verbindung mit NS3 |
| NS3 | 70-72 | 1027-1657 | Protease, Helikase |
| NS4A | 16-27 | 1658-1710 | Membranassoziation der NS3-NS4A-Komplexe, Kofaktor für NS3 |
| NS4B | 27 | 1711-1971 | membranassoziiert, unbekannte Funktion |
| NS5A | 56-58 | 1972-2419 | Phosphoprotein, früher mit Interferon-Sensitivität assoziiert |
| NS5B | 68-70 | 2420-3010 | RNA-abhängige RNA-Polymerase |
Der offene Leserahmen des etwa 9,5 kb langen HCV-Genoms wird von zwei nichtcodierenden Regionen (NCR, engl. non-coding region) flankiert, denen eine regulatorische Funktion während der Virusreplikation zukommt. Vor dem Startcodon (als Nukleotid-Position 1 definiert) befindet sich die etwa 340 Basen lange 5'-NCR, die innerhalb der HCV-Isolate die größte Sequenz-Übereinstimmung und eine komplexe Faltung des RNA-Stranges zeigt. Am Ende des Genoms findet sich die 250-300 Basen lange 3'-NCR. Im Wesentlichen besteht diese aus einem polymeren Uracil (polyU) bzw. Adenin (polyA) und einer hochkonservierten Nukleotidsequenz von 98 Basen Länge, die als X-Tail bezeichnet wird.
Replikation
Die Erkennung der Leberzelle (Hepatozyt) als Zielzelle und der Eintritt in die Zelle werden mit hoher Wahrscheinlichkeit über einen oder mehrere Rezeptoren vermittelt. Der Rezeptor für das HCV ist noch nicht eindeutig identifiziert; einige Hinweise deuten auf eine Bindung an den beta-Lipoprotein-Rezeptor, den CD81-Rezeptor und den HDL-Rezeptor (SRB1). Durch Hemmung des CD81 und des SRB1 konnte die Aufnahme von E1/E2 in Hepatozyten blockiert werden, doch keiner dieser Rezeptoren alleine reicht zur Infektion einer Zelle aus. Darüber hinaus ist keiner der genannten Rezeptoren leberspezifisch.
In Analogie zu anderen Mitgliedern der Flaviviridae nimmt man an, dass das HCV durch ein sich abschnürendes Bläschen der Zellmembran (Endosom) in die Zelle gelangt, wo es zur Fusion der Endosomenmembran mit der Virushülle und dadurch zur Freisetzung des Coreprotein-RNA-Komplexes in das Zytosol kommt. Das als mRNA lesbare HCV-Genom gelangt zunächst direkt zu den Ribosomen des rauen ER, wo eine erste Synthese von Virusproteinen stattfindet. Dieser erste Schritt ist notwendig, da das Virus zuerst einige Moleküle der viralen RNA-abhängigen RNA-Polymerase NS5B benötigt und dieses Enzym in der Zelle nicht vorhanden ist.
Um die Menge an verfügbarer viraler mRNA zu erhöhen, folgt als weiterer Schritt die Vermehrung der viralen RNA. Dazu synthetisiert die NS5B-Polymerase einen Gegenstrang (Negativstrang) der viralen (+)RNA als Vorlage (Matrize) für die folgende Synthese weiterer Plusstränge. Als Initiationssignal für die NS5B-Polymerase dienen wahrscheinlich die spezifisch gefalteten Abschnitte in den beiden nichtcodierenden Regionen der viralen RNA. Es gibt Hinweise darauf, dass sich beide Enden des RNA-Genoms über die Bindung an zelluläre Proteine zu einem Ring schließen und dadurch ein Replikationskomplex im Sinne eines Rolling-circle-Mechanismus entsteht. In HCV-replizierenden Zellkulturen konnten im Elektronenmikroskop auffällige Membranstrukturen gefunden werden, die als HCV-Replikationskomplexe angesehen werden. Diese als „membranous web“ (membranöses Netz) bezeichneten Strukturen werden durch das hydrophobe Membranprotein NS4B induziert. Ganz ähnliche spezifische Membranveränderungen finden sich als sogenanntes Viroplasma bei allen (+)ss-RNA-Viren, die diesbezüglich untersucht wurden.
Nach ausreichender Synthese der viralen mRNA beginnt nun die Translation der viralen Proteine, vorwiegend der Strukturproteine. Zur Initiation der Translation an den Ribosomen nutzen eukaryotische mRNAs in der Regel eine Modifizierung des 5’-Endes, das sogenannte Cap. Das HCV besitzt durch die Faltung der 5'-NCR jedoch eine besondere Struktur zur CAP-unabhängigen Initiation, die so genannte Interne ribosomale Eintrittstelle (engl. Internal ribosomal entry site, IRES). Dadurch benötigt die HCV-RNA keine zellulären Faktoren zur Bindung an die Ribosomen und zum Start der Proteinsynthese. Die IRES findet sich sonst nur bei einigen Picornaviren wie zum Beispiel dem Poliovirus.
Noch während der Translation werden die Strukturproteine von zellulären Proteasen (Signalase) vom entstehenden Polyproteinstrang abgeschnitten; die Hüllproteine E1 und E2 gelangen in das Lumen des ER, lagern sich in die Membran ein und werden glykosyliert. Das Core-Protein lagert sich von außen in die ER-Membran und bindet aufgrund seiner Ladungseigenschaften die virale mRNA. Es kommt nun zur Verpackung des Core-RNA-Komplexes und zur Abschnürung (Knospung, engl. budding) des HCV-Partikels in das Lumen des ER. Die neuen Virionen verlassen nun durch zelluläre Sekretion über den Golgi-Apparat die Zelle.
Wirte
Der Mensch ist einziger natürlicher Wirt des HCV. Bei der Erforschung des HCV in den 1980er Jahren und den Versuchen zur Herstellung eines Impfstoffes ab 1990 wurden Menschenaffen (vorwiegend Schimpansen) künstlich infiziert. Diese sind ebenso infizierbar, eine chronische Infektion ist jedoch selten. Diese damals vorwiegend in den USA durchgeführten Versuche sind in Europa aus ethischen Gründen untersagt.
Trotz intensiver Experimente steht erst seit 2004 ein Virus-produzierendes Zellkultursystem für HCV zur Verfügung, das die Erforschung der Vermehrung des Virus in der Zelle und die Entwicklung neuer antiviraler Substanzen wesentlich beschleunigen dürfte.
Epidemiologie
Die Prävalenzratio (Zahl der Erkrankten im Verhältnis zur Zahl der Untersuchten) beträgt weltweit 0,031, in Europa und den USA weniger als 0,02. In Japan, der Mongolei und Ägypten liegt sie bei 0,181, in letzterem verursacht durch Fehler bei einer Behandlung gegen Schistosomiasis (kontaminierte Kanülen). Weltweit geht die WHO von 170 Millionen chronischen Virusträgern aus. Die Anzahl der an Hepatitis C Erkrankten ist in manchen Ländern wesentlich geringer.
Die Verteilung der Geno- und Subtypen folgt auch geographischen Mustern: in Europa und Amerika finden sich vorwiegend die sonst weltweit vorkommenden Subtypen 1a, 1b und 3a (vorwiegend bei Drogenabhängigen); in Asien ist der Subtyp 1b der dominierende, in Afrika der Genotyp 4, in Südafrika der Genotyp 5 und in Hongkong und Vietnam der Genotyp 6. Die Genotypen 2 und 3 sind weltweit vertreten, jedoch in wesentlich geringerem Ausmaß.
Der Zeitraum der stammesgeschichtlichen Trennung der Genotypen konnte mittels der Sequenzvariabilität und der berechneten Mutationshäufigkeit auf etwa 500 Jahre geschätzt werden. Daher ist zu vermuten, dass die Diversifizierung der Genotypen mit dem Aufkommen der globalen Seefahrten um das Jahr 1500 einherging.
Übertragung
Das Virus wird parenteral übertragen, d. h. vorwiegend durch Blut und Blutprodukte; eine sexuelle Übertragung ist eher selten. Damit sind folgende Risiken definiert: Dialyse (besonders vor 1991), Bluttransfusion (vor 1991), intravenöser Drogenkonsum, Tätowierung und Piercing. Bei etwa 30 % der Patienten ist der Übertragungsweg unbekannt.
Virostatika
Zugelassene und experimentelle Virostatika gegen das Hepatitis-C-Virus umfassen Ribavirin gegen die RdRP, die NS3/4A-Inhibitoren wie Asunaprevir, Boceprevir, Ciluprevir, Danoprevir, Faldaprevir, Glecaprevir, Grazoprevir, Narlaprevir, Paritaprevir, Simeprevir, Sovaprevir, Telaprevir, Vaniprevir, Vedroprevir, Voxilaprevir und die NS5A-Inhibitoren Daclatasvir, Elbasvir (zugelassen für HCV1 und HCV4), Ledipasvir, MK-8408, Odalasvir, Ombitasvir, Ravidasvir und Velpatasvir.
Meldepflicht
In Deutschland ist jeder direkte oder indirekte Nachweis vom Hepatitis-C-Virus namentlich meldepflichtig nach § 7 des Infektionsschutzgesetzes.
In der Schweiz ist der positive laboranalytische Befund zu einem Hepatitis-C-Virus meldepflichtig und zwar nach dem Epidemiengesetz (EpG) in Verbindung mit der Epidemienverordnung und Anhang 3 der Verordnung des EDI über die Meldung von Beobachtungen übertragbarer Krankheiten des Menschen.
Quellen
- H.-J. Thiel u. a.: Genus Hepacivirus. In: C. M. Fauquet, M. A. Mayo u. a.: Eighth Report of the International Committee on Taxonomy of Viruses. London, San Diego 2005, ISBN 0-12-249951-4, S. 993–998.
- David M. Knipe, Peter M. Howley (eds.-in-chief): Fields’ Virology. 5. Auflage. 2 Bände. Philadelphia 2007, ISBN 978-0-7817-6060-7, S. 1113–1126 und 1253–1291.
Weblinks
- Hepatitis C virus (Taxonomie-Datenbank des NCBI)
- Referenzsequenz NC 004102 des HCV
Einzelnachweise
- ICTV Master Species List 2018b.v2. MSL #34, März 2019
- ICTV: ICTV Taxonomy history: Yellow fever virus, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- D. Martin, J. S. Gutkind: Human tumor-associated viruses and new insights into the molecular mechanisms of cancer. In: Oncogene. Band 27, Nr. 2, 2008, S. 31–42, PMID 19956178.
- The Nobel Prize in Physiology or Medicine 2020. In: nobelprize.org, 5. Oktober 2020.