Virusklassifikation
Die Klassifikation von Viren beruht auf verschiedenen morphologischen, epidemiologischen oder biologischen Merkmalen. Von der rein funktionellen Klassifikation ist jedoch die offiziell gültige Taxonomie von Viren zu unterscheiden, nach der Viren beispielsweise in Familien, Gattungen und Arten nach dem Grad ihrer Verwandtschaft (ermittelt insbesondere durch Genomvergleich) eingeteilt werden. Alle anderen Klassifikationen sind noch zum Teil aus historischen oder praktischen Gründen üblich. Die Entscheidung über Kriterien der Taxonomie und Einteilung von Viren trifft ein internationales Gremium mit dem Namen International Committee on Taxonomy of Viruses (ICTV).
Das klassische System der Virusklassifikation
Im Jahre 1962 wurde von André Lwoff, Robert W. Horne und Paul Tournier entsprechend der von Carl von Linné begründeten binären Klassifikation der Lebewesen eine Taxonomie der Viren („LHT-System“) eingeführt. In ihr werden analog zur Taxonomie anderer Lebewesen, die folgenden Taxa unterteilt:
- Virosphäre (Phylum: Vira)
Die entscheidenden Charakteristika für diese Klassifikation waren:
- die Natur des viralen Genoms (DNA oder RNA)
- die Symmetrie des Kapsids
- Vorhandensein einer Lipidumhüllung
- Größe von Virion und Kapsid
Die drei morphologischen Kriterien bestimmen den Morphotyp (Morphovar)
Frühe Klassifizierungssysteme berücksichtigten anstelle der noch nicht allgemein verfügbaren Genomsequenz folgende Kriterien:
- gemeinsame Organismen, die sie infizieren (Wirte)
- gemeinsame Übertragungswege, z. B. Vektoren
- ähnliche Krankheitsbilder (Symptome) bzw. Infektion des gleichen Organs
- Serotyp (Serovar)
- Habitat (Vorkommen)
- Klassifikation nach Wirten
- Bakteriophagen – infizieren Bakterien oder – nach herkömmlichen Verständnis auch – Archaeen (der veralteten Bezeichnung „Archaebakterien“ folgend)
- Cyanophagen – infizieren Cyanobakterien (alias Blaugrünbakterien, veraltet: „Blaualgen“)
- Coliphagen — infizieren Escherichia coli (Colibakterien)
- Aktinobakteriophagen — infizieren Bakterien der Klasse Actinobacteria (Aktinobakterien)
- Corynephagen — infizieren Aktinobakterien der Gattung Corynebacterium (Corynebakterien)
- Mykobakteriophagen — infizieren Aktinobakterien der Gattung Mycobacterium (Mykobakterien)
- Archaeenviren – infizieren Archaeen
- Mykoviren – infizieren Pilze
- Phytoviren – infizieren Pflanzen
- Tierviren – infizieren Tiere (inkl. Menschen als Primaten)
- Insektenviren
- humanpathogene Viren – infizieren den Menschen
- Klassifikation nach Krankheitsbildern
- Onkoviren – karzinogen
- Hepatitis-Viren
- Influenzaviren (früheres Taxon) — verursachen Influenza (echte Grippe)
- Rhinoviren — verursachen grippale Infekte
- Nekroseviren
- Mosaikvirus
- Klassifikation nach Übertragungsweg
- Arboviren – Arthropoden (Gliederfüßer) als Vektoren
- Klassifikation nach Habitat (Vorkommen)
- Haloviren – salzliebend
- Klassifikation nach Größe (Kapsid/Genom):
- Riesenviren
- Jumbophagen, Megaphagen
In der modernen Taxonomie bleiben diese Kriterien unberücksichtigt, auch wenn sie weiter für die Zusammenfassung unterschiedlicher Viren mit gemeinsamen medizinischen oder epidemiologischen Merkmalen wichtig sind.
Virustaxonomie nach ICTV
Die moderne Virus-Taxonomie nach ICTV orientiert sich ebenfalls nach Linné. Sie umfasst seit 2018 auch die höheren Rangstufen oberhalb der Ordnung, mit Namensendungen, die teilweise vom LHT-System abweichen.[1][2]
- Realm (en. realm) (…viria)
- Subrealm (en. subrealm) (…vira) (Endung wie bei Subphylum im LHC-System, als zweiteoberste Stufe)
- Reich (en. kingdom) (…virae)
- Unterreich (en. subkingdom) (…virites)
- Stamm oder Phylum (…viricota) (in Analogie zu …archaeota - abweichend vom LHC-System sind mehrere Virusphyla möglich)
- Subphylum (…viricotina)
- Klasse (…viricetes)
- Unterklasse (…viricetidae)
- Ordnung (…virales)
- Unterordnung (…virineae)
- Familie (…viridae)
- Unterfamilie (…virinae)
- Gattung oder Genus (…virus)
- Untergattung oder Subgenus (…virus)
- Art oder Species (…virus)
- Untergattung oder Subgenus (…virus)
- Gattung oder Genus (…virus)
- Unterfamilie (…virinae)
- Familie (…viridae)
- Unterordnung (…virineae)
- Ordnung (…virales)
- Unterklasse (…viricetidae)
- Klasse (…viricetes)
- Subphylum (…viricotina)
- Stamm oder Phylum (…viricota) (in Analogie zu …archaeota - abweichend vom LHC-System sind mehrere Virusphyla möglich)
- Unterreich (en. subkingdom) (…virites)
- Reich (en. kingdom) (…virae)
- Subrealm (en. subrealm) (…vira) (Endung wie bei Subphylum im LHC-System, als zweiteoberste Stufe)
Mit Mitte März 2021 gibt es sechs vom ICTV anerkannte Realms, die sich wie folgt unterteilen:[3]
- Realm Adnaviria (Baltimore 1)
- Realm Duplodnaviria (Baltimore 1)
- Realm Monodnaviria (Baltimore 1 und 2)
- Realm Riboviria (Baltimore 3 bis 7)
- Realm Ribozyviria (Gattung Deltavirus und ähnliche; ~ Baltimore 5, aber Satellitenviren mit zirkulärer RNA)
- Realm Varidnaviria (Baltimore 1)
- nicht-zugeordnete Taxa:
- 1 Klasse: Naldaviricetes (Baltimore 1)
- 19 Familien
- 2 Gattungen
Die Baltimore-Klassifikation
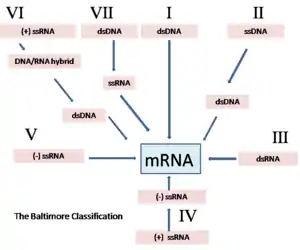
Auf Grundlage des Wissens um die Molekularbiologie der Viren hatte sich seit 1971 eine weitere Klassifikation etabliert, welche auf einen Vorschlag des Nobelpreisträgers David Baltimore zurückgeht.[4][5]
Wichtige Kriterien dieser Klassifizierung sind:
- die Genomstruktur
- die Form (Symmetrie) des Kapsids
- das Vorhandensein einer Hülle
- Anordnung der Gene innerhalb des Genoms
- Replikationsstrategie
- Virusgröße
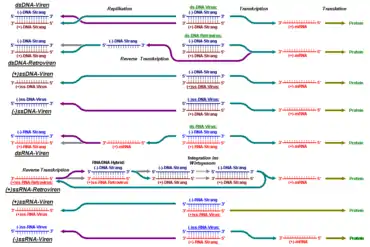
Die verschiedenen Möglichkeiten ergeben sich dadurch, dass ein Strang der doppelsträngigen DNA, so wie sie in allen zellulären Organismen vorliegt, redundant ist und bei Viren daher entfallen kann. Ebenso kann das Virusgenom auch in verschiedenen Formen der RNA vorliegen, die in Zellen als Zwischenstufe bei der Proteinsynthese (mRNA) auftritt. Bei einzelsträngiger DNA oder RNA kommen beide möglichen Kodierungsrichtungen vor: die normale Richtung 5'→3', die als (+) Polarität bezeichnet wird, wie sie in der mRNA vorliegt, und die entgegengesetzte (komplementäre) Richtung (-), in der die RNA quasi als Negativ vorliegt.[6]
Diese Baltimore-Klassifikation nach der Replikationsstrategie wird heute zunehmend ungebräuchlich, denn
- seit 2018 stehen durch das ICTV definierte hohe Rangstufen (oderhalb der Ordnung) zur Verfügung
- viele aufgrund von Sequenzvergleichen identifizierte Verwandtschaftsgruppen (Kladen) erstrecken sich über mehrere Baltimore-Gruppen. Unter den ersten solchen vom ICTV bestätigten Taxa waren
- die Ortervirales (Baltimore 6 und 7) – revers transkribierende RNA- und DNA-Viren: Retroviren und Pararetroviren. Es handelt sich um RNA-Viren mit DNA-Zwischenstadium bzw. umgekehrt.
- die Pleolipoviridae (Baltimore 1 und 2) – Vertreter mit dsDNA und Vertreter mit ssDNA
- bei den Bacilladnaviridae (ssDNA) gibt es kleine, lineare ssDNA-Fragmente, die komplementär sind zu Abschnitten des großen, zirkulären ssDNA-Genoms. Durch deren Anlagerung entsteht ein zirkuläres DNA-Genom, das in kurzen Abschnitten dsDNA, ansonsten ssDNA ist.
- die Endornaviridae sind (+)ssRNA-Viren (nach ICTV, das ist Baltimore 4), aber mit dsRNA-Zwischenstadium (Baltimore 3), meist wird letzteres isoliert. Daher werden sie verschiedentlich auch als dsRNA-Viren klassifiziert.[7]
- Im Übrigen wird schon nach Baltimore bei Einzelstrang-DNA-Viren nicht zwischen den beiden Polaritäten unterschieden, nur bei Einzelstrang-RNA-Viren. Grund ist, dass es bei ersteren keine klare Unterscheidbarkeit gibt.
DNA-Viren
DNA-Viren bilden keine taxonomische Verwandtschaftsgruppe (Klade), stattdessen ist dieser Begriff lediglich eine Sammelbezeichnung der Virusklassifikation. Unter den DNA-Viren konnten jedoch eine Reihe von Taxa (Ordnungen und höher) als Verwandtschaftsgruppen ausgemacht und vom ICTV bestätigt werden.
Baltimore-Gruppe I
Doppelstrang-DNA – dsDNA (englisch double strand), normale Genom-Form allen Lebens.[8]
- Realm Adnaviria
- Reich Zilligvirae
- Phylum Taleaviricota
- Klasse Tokiviricetes
- Ordnung Ligamenvirales
- Ordnung Primavirales mit Familie Tristromaviridae
- Realm Varidnaviria (ursprünglich vorgeschlagen als „Divdnaviria“, „Vertical jelly roll major capsid protein DNA viruses“, „DJR-MCP-Viren“)[9][10]
- zum Reich Bamfordvirae
- Phylum Nucleocytoviricota (veraltet „Nucleocytoplasmaviricota“, „Nucleocytoplasmic large DNA viruses“, NCLDV)
- Klasse Megaviricetes
- Zweig 1:
- Ordnung Algavirales
- Ordnung Imitervirales (veraltet „Megavirales“ s. s.[11]: die erweiterte Familie Mimiviridae syn. „Megaviridae“[12] – mit den Mimiviren)
- Zweig 2:
- Ordnung Pimascovirales (früher „MAPI-Superklade“[11] oder „PMI-Gruppe“)
- Familien Marseilleviridae, Ascoviridae, Iridoviridae, evtl. „Mininucleoviridae“, „Pithoviridae“, sowie Gattung Ichnovirus aus Polydnaviridae
- Klasse Pokkesviricetes
- Zweig 3:
- Ordnung Asfuvirales, evtl. mit Gattung Dinodnavirus
- Ordnung Chitovirales (mit den Pockenviren)
- Phylum Preplasmiviricota
- Klasse Tectiliviricetes
- Ordnung Rowavirales (mit Adenoviren)
- Ordnung Kalamavirales
- Ordnung Vinavirales
- Ordnung Belfryvirales (mit Turriviridae)[13] (mit Gattung Alphaturrivirus,[14] dazu Spezies Sulfolobus turreted icosahedral virus 1 und 2, STIV1 und STIV2)
- ohne Ordnungszuweisung
- Familie Autolykiviridae
- Klasse Maveriviricetes
- Ordnung Priklausovirales (mit Virophagen der Lavidaviridae)
- Reich Helvetiavirae[10]
- Phylum Dividoviricota
- Klasse Laserviricetes
- Ordnung Halopanivirales (mit Sphaerolipoviridae)
- ohne Zuordnung zu einer Ordnung, Klasse, Phylum
- Familie Portogloboviridae (gemäß Vorschlag)
- Realm Duplodnaviria (Vorschlag)[10]
- Reich Heunggongvirae
- Phylum Uroviricota
- Klasse Caudoviricetes
- Ordnung Caudovirales (Bakteriophagen etc. mit Kopf-Schwanz-Struktur)
- Phylum Peploviricota
- Klasse Herviviricetes
- Ordnung Herpesvirales
- Reich Shotokuvirae (hier nur Vertreter mit dsDNA: Papovaviricetes)
- Phylum Cossaviricota (dito)
- Klasse Papovaviricetes
- weitere mögliche Kandidaten für diese Gruppe sind die folgenden dsDNA-Virusfamilen:
- in keinen höheren Rang klassifizierte Klasse:
- Klasse Naldaviricetes (ehemals „Modul 6“) mit folgenden Familien nach Vorschlag von Koonin et al. (2015, 2019)[19][20][21]
- Ordnung Lefavirales
- Familie Baculoviridae
- Familie Hytrosaviridae
- Familie Nudiviridae
- Ordnung nicht zugewiesen
- Familie Nimaviridae
- bisher nicht vom ICTV bestätigter Vorschlag
- Familie Polydnaviridae (vermutlich polyphyletisch,[22] hier nur Gattung Bracovirus)
- in keinen höheren Rang klassifizierte Familien:
- Familie Ampullaviridae
- Familie Bicaudaviridae
- Familie Clavaviridae
- Familie Fuselloviridae
- Familie Globuloviridae
- Familie Guttaviridae
- Familie Ovaliviridae
- Familie Plasmaviridae
- Familie Polydnaviridae (möglicherweise polyphyletisch, siehe Bracovirus, Ichnovirus (siehe NCLDV, vermutlich eine gemeinsame Klade mit Ascoviridae und Iridoviridae zu Pimascovirales in Nucleocytoviricota))
- Familie Portogloboviridae
- Familie Thaspiviridae
- Familie Halspiviridae
- in keinen höheren Rang klassifizierte Gattungen:
- Gattung Dinodnavirus (siehe NCLDV, möglicherweise nahe zu Asfarviridae und der vorgeschlagenen Familie Orpheoviridae)[23]
- Gattung Rhizidiovirus[24] (mit einziger Spezies Rhizidiomyces virus)
- in keinen höheren Rang klassifizierte Spezies:
- Spezies „Tetraselmis viridis virus N1“ (alias „Tetraselmis striata virus N1“ (TsV-N1))[25][26][27]
- Spezies „Heterosigma akashiwo nuclear inclusion virus“ (HaNIV)[28][26][29]
Baltimore-Gruppe II
Einzelstrang-DNA – ssDNA (englisch single strand). Virionen enthalten DNA positiver oder negativer Polarität.[30] Die Virusgruppe „CRESS“ (circular Rep-encoding single-strand DNA viruses) (im weiteren Sinne) ist keine Verwandtschaftsgruppe (Taxon), sondern polyphyletisch.[31][32][33][34][35] Das neue Phylum Cressdnaviricota fasst jedoch die hauptsächlichen Vertreter dieser in einer Verwandtschaftsgruppe zusammen.
- Realm Monodnaviria (hier bis auf Sonderfall Pleolipoviridae)
- Reich Loebvirae
- Phylum Hofneiviricota
- Klasse Faserviricetes (filamentös)
- Ordnung Tubulavirales (zu „CRESS“)
- Familie Inoviridae
- Familie Paulinoviridae
- Familie Plectroviridae
- Reich Sangervirae
- Phylum Phixviricota
- Klasse Malgrandaviricetes
- Ordnung Petitvirales (mit Microviridae, zu „CRESS“ – circular Rep-encoding single-strand DNA viruses)
- Reich Shotokuvirae
- Phylum Cossaviricota (ohne Papovaviricetes da jene mit zirkulärer dsDNA)
- Klasse Quintoviricetes (mit Parvoviridae, zu „CRESS“)
- Klasse Mouviricetes (mit Bidnaviridae)
- Phylum Cressdnaviricota
- Klasse Repensiviricetes
- Ordnung Geplafuvirales (mit Geminiviridae und Genomoviridae)
- Klasse Arfiviricetes
- Ordnung Baphyvirales (mit Bacilladnaviridae)
- Ordnung Recrevirales (mit Redondoviridae)
- Ordnung Cirlivirales (mit Circoviridae)
- Ordnung Cremevirales (mit Smacoviridae)
- Ordnung Mulpavirales (mit Metaxyviridae und Nanoviridae)
- Ordnung Recrevirales(mit Redondoviridae, ssDNA)
- ohne Zuordnung zu einer Klasse oder Ordnung:
- Familie „Cruciviridae“ (Vorschlag)
- keinem höheren Rang zugeordnete Familien:
- Familie Spiraviridae ssDNA(+)
- Familie Anelloviridae ssDNA(-)
- Familie Finnlakeviridae ssDNA
- keinem höheren Rang zugeordnete vorgeschlagene Kladen (Gattungen?):
- Klade „Circularisvirus“[36]
- Spezies: „Dragonfly circularisvirus“ (DfCirV)[37] (zu „CRESS2“[38])
- Spezies: „Cybaeus spider associated circular virus 1“ (CySACV-1), in Spinnen der Gattung Cybaeus[39]
- Spezies: „Golden silk orbweaver associated circular virus 1“ (GoSOrbACV-1), in Spinnen der Gattung Nephila[39]
- Spezies: „Longjawed orbweaver circular virus 1“ (LjOrbCV-1), in Spinnen der Gattung Leucauge[39]
- Spezies: „Spinybacked orbweaver circular virus 1“ (SpOrbCV-1), in Spinnen der Gattung Gasteracantha[39]
- Klade „Volvovirus“[40]
- Spezies: „Acheta domesticus volvovirus“ (AdVVV), in Heimchen[41][42]
- Spezies: „Cricket associated circular virus 1“ (CrACV-1), in Feldgrillen[39] (zu „nicht-klassifizierten CRESS“[38])
- keinem höheren Rang zugeordnete vorgeschlagene Spezies:
Pleolipoviridae
Die Familie Pleolipoviridae war (mit der Ordnung Ortervirales) eines der ersten vom ICTV anerkannten Taxa (Verwandtschaftsgruppe, ermittelt nach der Genom-Sequenz), dessen Mitglieder in verschiedenen Baltimore-Gruppen (nämlich 1 und 2) fallen.
- zum Realm Monodnaviria
RNA-Viren
Im Wesentlichen identisch mit dem Realm Riboviria. Dazu gehören auch die vorgeschlagenen Supergruppen
- „Picornavirus-like superfamily“
- „Alphavirus-like superfamily“
nach Koonin et al. (2015), die Kladen verschiedener Baltimore-Gruppen zusammenfassen.
Baltimore-Gruppe III
Doppelstrang-RNA – dsRNA[48]
- zum Realm Riboviria (hier nur dsRNA)
- zum Reich Orthornavirae
- zum Phylum Pisuviricota
- zur Klasse Duplopiviricetes
- zur Ordnung Durnavirales (hier nur dsRNA-Viren)
- Familie Amalgaviridae
- Familie Partitiviridae (mit den Gattungen Alphapartitivirus, Betapartitivirus, Gammapartitivirus, Deltapartitivirus, Cryspovirus)
- Familie Picobirnaviridae (mit Gattung Picobirnavirus)
- zum Phylum Duplornaviricota
- zur Klasse Chrymotiviricetes
- zur Ordnung Ghabrivirales
- Familie Chrysoviridae (mit Gattung Alphachrysovirus, sowie Betachrysovirus,[49] mit Spezies Colletotrichum fructicola chrysovirus 1)
- Familie Megabirnaviridae (mit Gattung Megabirnavirus)
- Familie Quadriviridae (mit Gattung Quadrivirus)
- Familie Totiviridae
- zur Klasse Vidaverviricetes
- Ordnung Mindivirales (mit Familie Cystoviridae)
- zur Klasse Resentoviricetes
- Ordnung Reovirales (mit Familie Reoviridae, nach Vorschlag Koonin et al. (2015) von den Cystoviridae abstammend)
- zum Phylum Kitrinoviricota
- zur Klasse Alsuviricetes
- zur Ordnung Martellivirales
- Familie Endornaviridae
- innerhalb der Orthornavirae keinem höheren Rang zugeordnet ist die Familie:
- Familie Birnaviridae
- innerhalb der Orthornavirae keinem höheren Rang zugeordnet ist die Gattung:
- Gattung Botybirnavirus
Baltimore-Gruppe IV
Positive Einzelstrang-RNA – ss(+)RNA. Sie wirkt direkt als mRNA.[56]
- zum Realm Riboviria (hier nur ss(+)RNA)
- zum Reich Orthornavirae − hier nur ss(+)RNA
- Phylum Pisuviricota
- Klasse Pisoniviricetes
- Ordnung Nidovirales
- Ordnung Picornavirales
- Ordnung Sobelivirales (mit den Familien Barnaviridae, Alvernaviridae, Solemoviridae)
- Klasse Stelpaviricetes
- Ordnung Stellavirales (mit Familie Astroviridae, diese mit Gattung Mamastrovirus)
- Ordnung Patatavirales(mit Familie Potyviridae)
- Klasse Duplopiviricetes
- Ordnung Durnavirales
- Phylum Kitrinoviricota
- Klasse Alsuviricetes
- Ordnung Tymovirales
- Ordnung Hepelivirales (mit den Familie Hepeviridae, Alphatetraviridae, Benyviridae, Matonaviridae – letztere mit Rötelnvirus)
- Ordnung Martellivirales
- Klasse Flasuviricetes
- Ordnung Amarillovirales (mit Familie Flaviviridae)
- Klasse Tolucaviricetes
- Ordnung Tolivirales (mit den Familien Luteoviridae, Carmotetraviridae, Tombusviridae)
- Klasse Magsaviricetes
- Ordnung Nodamuvirales
- Phylum Lenarviricota
- Klasse Allassoviricetes
- Ordnung Levivirales (mit Familie Leviviridae)
- keinem höheren Rang unter Orthornaviae zugeteilt:
- Familie Permutotetraviridae
- keinem höheren Rang unter Riboviria zugeteilt:
Baltimore-Gruppe V
Negative Einzelstrang-RNA – ss(-)RNA. Sie wirkt als Matrize zur mRNA Synthese.[59]
- zum Realm Riboviria (hier nur ss(-)RNA)
- Phylum Negarnaviricota (veraltet: Flavivirus-like superfamily – im Sinn von Supergruppe)[20]
- Realm Riboviria – ss(-)RNA.
- Phylum, Subphylum, Klasse, Ordnung nicht bestimmt bei:
- Familie: Kolmioviridae (mit Gattung Deltavirus)
Revers transkribierende Viren
Dazu gehören Viren mit positiver Einzelstrang-RNA, die per Reverse Transkriptase (RT) in DNA zurückgeschrieben und ins Zellgenom eingebaut wird (Retroviren ssRNA-RT, Baltimore-Gruppe 6), sowie Viren mit Doppelstrang-DNA, die umgekehrt zur Replikation einen RNA-Zwischenschritt benutzen und daher ebenfalls revers transkribieren (Pararetroviren dsDNA-RT, Baltimore-Gruppe 7). Die meisten dieser Vertreter gehören unabhängig davon der Ordnung Ortervirales an.[60]
Baltimore-Gruppe VI
Positive Einzelstrang-RNA, die per Reverse Transkriptase (RT) in DNA zurückgeschrieben und ins Zellgenom eingebaut wird (Retroviren).
- zum Realm Riboviria
- zur Ordnung Ortervirales (hier bis auf Caulimoviridae - revers transkribierende RNA-Viren ssRNA-RT)
- Familie Belpaoviridae (ssRNA-RT)
- Familie Metaviridae (ssRNA-RT)
- Familie Pseudoviridae (ssRNA-RT)
- Familie Retroviridae (ssRNA-RT, mit Gattung Lentivirus und darin den Spezies HIV-1 und -2, SIV, BIV, FIV)
Baltimore-Gruppe VII
Doppelstrang-DNA, die zur Replikation einen RNA-Zwischenschritt benutzt (revers transkribierende DNA-Viren dsDNA-RT, Pararetroviren).
- zum Realm Riboviria
- zur Ordnung Ortervirales (hier nur Sonderfall Caulimoviridae ssDNA-RT)
- Familie Caulimoviridae (dsDNA-RT: Revers transkribierende DNA-Viren)
- Ordnung Blubervirales (dsDNA-RT: Revers transkribierende DNA-Viren, mit Hepadnaviridae)
Tabellarische Übersicht
| Nukleinsäure | Kapsidsymmetrie | Hülle | Genom | Baltimore-Klasse | Familie | Genus | Arten |
|---|---|---|---|---|---|---|---|
| DNA | ikosaedrisch | nackt | ss(+/-) | II | Parvoviridae | Erythroparvovirus | Parvovirus B19 |
| DNA | ikosaedrisch | nackt | ds zirkulär | I | Polyomaviridae | Betapapillomavirus | Humanes Papillomvirus 1 und 2 |
| DNA | ikosaedrisch | nackt | ds zirkulär | I | Polyomaviridae | Polyomavirus | SV40, BK-Virus, JC-Virus |
| DNA | ikosaedrisch | nackt | ds | I | Adenoviridae | Mastadenovirus | Humanes AdV A–F |
| DNA | ikosaedrisch | umhüllt | ds | VII | Hepadnaviridae | Orthohepadnavirus | Hepatitis-B-Virus |
| DNA | ikosaedrisch | umhüllt | ds | I | Herpesviridae | Simplexvirus | Herpes-simplex-Virus 1 und 2 |
| DNA | ikosaedrisch | umhüllt | ds | I | Herpesviridae | Varicellovirus | Varizella-Zoster-Virus |
| DNA | ikosaedrisch | umhüllt | ds | I | Herpesviridae | Cytomegalovirus | Cytomegalievirus |
| DNA | ikosaedrisch | umhüllt | ds | I | Herpesviridae | Roseolovirus | Humanes Herpesvirus 6A, 6B und 7 |
| DNA | ikosaedrisch | umhüllt | ds | I | Herpesviridae | Lymphocryptovirus | Epstein-Barr-Virus |
| DNA | komplex | umhüllt | ds | I | Poxviridae | Orthopoxvirus | Variolavirus, Vacciniavirus |
| DNA | komplex | umhüllt | ds | I | Poxviridae | Parapoxvirus | Orf-Virus |
| RNA | ikosaedrisch | nackt | ss(+) | IV | Picornaviridae | Enterovirus | Poliovirus 1–3, Rhinovirus A–C, Coxsackievirus, Echovirus |
| RNA | ikosaedrisch | nackt | ss(+) | IV | Picornaviridae | Parechovirus | Parechovirus A |
| RNA | ikosaedrisch | nackt | ss(+) | IV | Picornaviridae | Hepatovirus | Hepatitis-A-Virus |
| RNA | ikosaedrisch | nackt | ss(+) | IV | Picornaviridae | Cardiovirus | Cardiovirus A (Mengovirus, EMC-Virus) |
| RNA | ikosaedrisch | nackt | ss(+) | IV | Astroviridae | Mamastrovirus | Mamastrovirus 1, 6, 8, 9 |
| RNA | ikosaedrisch | nackt | ss(+) | IV | Caliciviridae | Norovirus | Norwalk-Virus |
| RNA | ikosaedrisch | nackt | ss(+) | IV | Hepeviridae | Orthohepevirus | Hepatitis-E-Virus |
| RNA | ikosaedrisch | nackt | ds(10–18 Segmente) | III | Reoviridae | Coltivirus | Colorado-Zeckenfieber-Virus |
| RNA | ikosaedrisch | nackt | ds(10–18 Segmente) | III | Reoviridae | Orthoreovirus | Mammalian orthoreovirus |
| RNA | ikosaedrisch | nackt | ds(10–18 Segmente) | III | Reoviridae | Rotavirus | Rotavirus |
| RNA | ikosaedrisch | umhüllt | ss(+) | IV | Togaviridae | Alphavirus | Sindbisvirus |
| RNA | ikosaedrisch | umhüllt | ss(+) | IV | Togaviridae | Rubivirus | Rötelnvirus |
| RNA | ikosaedrisch | umhüllt | ss(+) | IV | Flaviviridae | Flavivirus | Gelbfieber-Virus, Hepatitis-C-Virus |
| RNA | komplex | umhüllt | ss(+) | VI | Retroviridae | Deltaretrovirus | HTLV-1 HTLV-2 |
| RNA | komplex | umhüllt | ss(+) | VI | Retroviridae | Spumavirus | Spumavirus |
| RNA | komplex | umhüllt | ss(+) | VI | Retroviridae | Lentivirus | HIV-1,-2, SIV |
| RNA | helikal | umhüllt | ss(+) | IV | Coronaviridae | Alphacoronavirus | Felines CoV |
| RNA | helikal | umhüllt | ss(+) | IV | Coronaviridae | Betacoronavirus | SARS-CoV, MERS-CoV |
| RNA | helikal | umhüllt | ss(-) | V | Orthomyxoviridae | Alphainfluenzavirus | Influenzavirus A |
| RNA | helikal | umhüllt | ss(-) | V | Orthomyxoviridae | Betainfluenzavirus | Influenzavirus B |
| RNA | helikal | umhüllt | ss(-) | V | Orthomyxoviridae | Gammainfluenzavirus | Influenzavirus C |
| RNA | helikal | umhüllt | ss(-) | V | Orthomyxoviridae | Deltainfluenzavirus | Influenzavirus D |
| RNA | helikal | umhüllt | ss(-) | V | Paramyxoviridae | Pneumovirus | Respiratorisches Synzytialvirus |
| RNA | helikal | umhüllt | ss(-) | V | Paramyxoviridae | Respirovirus | Parainfluenzavirus HPIV-1, HPIV-3 |
| RNA | helikal | umhüllt | ss(-) | V | Paramyxoviridae | Rubulavirus | Parainfluenzavirus HPIV-2, HPIV-4 |
| RNA | helikal | umhüllt | ss(-) | V | Paramyxoviridae | Rubulavirus | Mumpsvirus |
| RNA | helikal | umhüllt | ss(-) | V | Paramyxoviridae | Morbillivirus | Masernvirus |
| RNA | helikal | umhüllt | ss(-) | V | Rhabdoviridae | Lyssavirus | Tollwutvirus |
| RNA | helikal | umhüllt | ss(-) | V | Filoviridae | Filovirus | Marburgvirus, Ebolavirus |
| RNA | helikal | umhüllt | ss(-) | V | Peribunyaviridae | Orthobunyavirus | Bunyamweravirus |
| RNA | helikal | umhüllt | ss(-) | V | Nairoviridae | Orthonairovirus | Krim-Kongo-Virus |
| RNA | helikal | umhüllt | ss(-) | V | Phenuiviridae | Phlebovirus | Phlebotomus-Fieber-Virus |
| RNA | helikal | umhüllt | ss(-) | V | Hantaviridae | Orthohantavirus | Hantaan-Virus |
| RNA | helikal | umhüllt | ss(-) | V | Arenaviridae | Mammarenavirus | LCM-Virus, Lassa-Virus |
Weblinks
Einzelnachweise
- International Committee on Taxonomy of Viruses Executive Committe, Virus Taxonomy: 2018 Release, How to write virus and species names
- International Committee on Taxonomy of Viruses Executive Committee: The new scope of virus taxonomy: partitioning the virosphere into 15 hierarchical ranks, in: Nature Microbiology Band 5, S. 668–674 vom 27. April 2020, doi:10.1038/s41564-020-0709-x; sowie Nadja Podbregar: Ein Stammbaum für die Virosphäre, auf: scinexx.de vom 29. April 2020. Beide Artikel haben inhaltlich den Stand von Januar 2020, d. h. Einzelheiten der Master Species List (MSL) Nr. 35 des ICTV vom März 2020 sind noch nicht alle berücksichtigt. Für die grundsätzliche Intention des ICTV hat das aber keine Bedeutung, die Entwicklung ist mit der MSL#35 lediglich in der vorgegebenen Richtung schon wieder weitergegangen.
- MSL #36, März 2020
- Lars Gerdes, Ulrich Busch, Sven Pecoraro: Parallele Quantifizierung von gentechnisch veränderten Organismen (GVO), in: Schriftenreihe Gentechnik für Umwelt und Verbraucherschutz, Band 8, 5. Fachtagung "Gentechnik für Umwelt- und Verbraucherschutz", Oberschleißheim, November 2013: Conference Paper, hier: Abb. 6.1: Baltimore-Klassifikation und Virusfamilien
- D. Sander: Family Groups - The Baltimore Method, The Big Picture Book of Viruses, Garry Lab, 1995–2007
- SIB: Baltimore classification, auf: ViralZone
- SIB: Endornaviridae
- SIB: Double Strand DNA Viruses, auf: ViralZone
- Koonin EV, Dolja VV, Krupovic M, Varsani A, Wolf YI, Yutin N, Zerbini M, Kuhn JH: Create a megataxonomic framework for DNA viruses encoding vertical jelly roll-type major capsid proteins filling all principal taxonomic ranks. ICTV Proposal 2019.003G, April–Juli 2019
- ICTV Master Species List 2019 v1 MSL #35, März 2019
- Julien Guglielmini, Anthony C. Woo, Mart Krupovic, Patrick Forterre, Morgan Gaia: Diversification of giant and large eukaryotic dsDNnA viruses predated the origin of modern eukaryotes, in: PNAS, Band 116, Nr. 39, 10./24. September 2019, S. 19585–19592, doi:10.1073/pnas.1912006116, PMID 31506349, Fig. 2.
Julien Guglielmini, Anthony Woo, Mart Krupovic, Patrick Forterre, Morgan Gaia: Diversification of giant and large eukaryotic dsDNA viruses predated the origin of modern eukaryotes, auf: bioRxiv vom 29. Oktober 2018, doi:10.1101/455816. bioRxiv: 10.1101/455816v1 (Preprint-Volltext) (PrePrint). - Sailen Barik: A Family of Novel Cyclophilins, Conserved in the Mimivirus Genus of the Giant DNA Viruses, in: Computational and Structural Biotechnology Journal, Band 16, Juli 2018, S. 231–236, doi:10.1016/j.csbj.2018.07.001
- SIB: Turriviridae, auf: ViralZone
- SIB: Alphaturrivirus, auf: ViralZone
- Nicole L. Welch, Natalya Yutin et al.: Adomaviruses: an emerging virus family provides insights into DNA virus evolution, in: bioRxiv, 7. Juni 2018 bioRxiv: 2018/06/07/341131 (Preprint-Volltext), doi:10.1101/341131, insbes. Fig. 7
- Nicole L. Welch, Michael J. Tisza et al.: Identification of “Missing Link” Families of Small DNA Tumor Viruses, in: BioRxiv; Cold Spring Harbor, 11. Juli 2019, bioRxiv: 10.1101/697771v3 (Preprint-Volltext)
- NCBI: Adomaviridae (family)
- NCBI: Adintoviridae (family)
- Eugene V. Koonin, Natalya Yutin: Evolution of the Large Nucleocytoplasmatic DNA Viruses of Eukaryotes and Convergent Origins of Viral Gigantism, in: Advances in Virus research, Band 103, AP 21. Januar 2019, doi:10.1016/bs.aivir.2018.09.002, S. 167–202.
- Eugene V. Koonin, Valerian V. Dolja, Mart Krupovic: Origins and evolution of viruses of eukaryotes: The ultimate modularity, in: Virology vom Mai 2015; 479–480. 2–25, Epub 12. März 2015, PMC 5898234 (freier Volltext), PMID 25771806
- Jaime Iranzo, Mart Krupovic, Eugene V. Koonin: The Double-Stranded DNA Virosphere as a Modular Hierarchical Network of Gene Sharing, in: mBio 7(4), Juli–August 2016, e00978-16, doi:10.1128/mBio.00978-16, PMC 4981718 (freier Volltext), PMID 27486193
- Dupuy C, Huguet E, Drezen JM: Unfolding the evolutionary story of polydnaviruses. In: Virus Res. 117, Nr. 1, 2006, S. 81–89. doi:10.1016/j.virusres.2006.01.001. PMID 16460826.
- H. Ogata, K. Toyoda, Y. Tomaru, N. Nakayama, Y. Shirai, J. M. Claverie, K. Nagasaki: Remarkable sequence similarity between the dinoflagellate-infecting marine girus and the terrestrial pathogen African swine fever virus. In: Virol J. 6, 2009, S. 178.
- SIB: Rhizidiovirus, auf: ViralZone.
- NCBI: Tetraselmis viridis virus N1 (species)
- António Pagarete, Théophile Grébert, Olga Stepanova, Ruth-Anne Sandaa, Gunnar Bratbak: TsV-N1: A Novel DNA Algal Virus that Infects Tetraselmis striata, in: MDPI Viruses, Band 7, Nr. 7, Section Viruses of Plants, Fungi and Protozoa, 17. Juli 2015, S. 3937–3953, doi:10.3390/v7072806
- Christopher M. Bellas, Ruben Sommaruga: Polinton-like viruses and virophages are widespread in aquatic ecosystems, auf: CSH bioRxiv vom 13. Dezember 2019, doi:10.1186/s40168-020-00956-0
- Janice E. Lawrence, Amy M. Chan, Curtis A. Suttle: A novel virus (HaNIV) causes lysis of the toxic bloom-forming alga Heterosigma akashiwo (Raphidophyceae), in: Journal of Phycology, Band 37, Nr. 2, April 2001, Epub 1. Mai 2002, S. 216–222, doi:10.1046/j.1529-8817.2001.037002216.x
- Janice E. Lawrence, Corina P. D. Brussaard, Curtis A. Suttle: Virus-Specific Responses of Heterosigma akashiwo to Infection, in: Appl Environ Microbiol., Dezember 2006 Dec, Band 72, Nr. 12, 7S. 829–7834, Epub 23. Oktober 2006, doi:10.1128/AEM.01207-06, PMC 1694243 (freier Volltext), PMID 17041155
- SIB: Single Strand DNA Viruses, auf: ViralZone
- Mart Krupovic: Networks of evolutionary interactions underlying the polyphyletic origin of ssDNA viruses, in: Current Opinion in Virology Band 3, Nr. 5, Oktober 2013, S. 578–586, doi:10.1016/j.coviro.2013.06.010, PMID 23850154
- Karyna Rosario, Siobain Duffy, Mya Breitbart: A field guide to eukaryotic circular single-stranded DNA viruses: insights gained from metagenomics. In: Archives of Virology. 157, Nr. 10, Oktober 2012, S. 1851–1871. doi:10.1007/s00705-012-1391-y. PMID 22760663.
- Darius Kazlauskas, Arvind Varsani, Eugene V. Koonin, Mart Krupovic: Multiple origins of prokaryotic and eukaryotic single-stranded DNA viruses from bacterial and archaeal plasmids. In: Nature Communications. 10, Nr. 1, 2019, S. 3425. doi:10.1038/s41467-019-11433-0.
- NCBI: CRESS viruses
- Richard Harth: Major class of viruses reveals complex origins, auf: ScienceDaily: Science News, 31. Juli 2019, Arizona State University
- NCBI: Circularisvirus (clade)
- Karyna Rosario, Anisha Dayaram, Milen Marinov, Jessica Ware, Simona Kraberger, Daisy Stainton, Mya Breitbart, Arvind Varsani: Diverse circular ssDNA viruses discovered in dragonflies (Odonata: Epiprocta), in: Journal of General Virology Band 93, Nr. 12, 1. Dezember 2012, doi:10.1099/vir.0.045948-0. Insbes. Tbl. 2
- Darius Kazlauskas, Arvind Varsani, Mart Krupovic: Pervasive Chimerism in the Replication-Associated Proteins of Uncultured Single-Stranded DNA Viruses, in: MDPI Viruses, Band 10, Nr. 4, Special Issue Viral Recombination: Ecology, Evolution and Pathogenesis, 10. April 2018, 187, doi:10.3390/v10040187; siehe Excel-Datei in Supplement S1 (ZIP: pdf, xlsx)
- Karyna Rosario, Kaitlin Mettel, Bayleigh Benner, Ryan Johnson, Catherine Scott, Sohath Yusseff-Vanegas, Christopher Baker, Deby Cassill, Caroline Storer, Arvind Varsani, Mya Breitbart: Virus Discovery in All Three Major Lineages of Terrestrial Arthropods Highlights the Diversity of Single-stranded DNA Viruses Associated with Invertebrates, University of South Florida, College of Marine Science: Scholar Commons — Marine Science Faculty Publications, 2017, 652. Part of the Life Sciences Commons. 11. Oktober 2018 PeerJ, PMID 30324030, PMC 6186406 (freier Volltext), doi:10.7717/peerj.5761
- NCBI: Volvovirus (clade)
- Hanh T. Pham, Max Bergoin, Peter Tijssen: Acheta domesticus Volvovirus, a Novel Single-Stranded Circular DNA Virus of the House Cricket, in: Genome Announc. 1(2), März-April 2013, e00079-13. Epub 14. März 2013, doi: 10.1128/genomeA.00079-13, PMC 3623006 (freier Volltext), PMID 23516206
- Hanh T. Pham, Hajime Iwao, Max Bergoin, Peter Tijssen: New Volvovirus Isolates from Acheta domesticus (Japan) and Gryllus assimilis (United States), in: ASM Journals: Genome Announcements, Band 1, Nr. 3, 6. Juni 2013, doi:10.1128/genomeA.00328-13
- NCBI: Dragonfly orbiculatusvirus (species)
- NCBI: Dragonfly cyclicusvirus (species)
- Sencilo A, Paulin L, Kellner S, Helm M, Roine E: Related haloarchaeal pleomorphic viruses contain different genome types. In: Nucleic Acids Research. 40, Nr. 12, Juli 2012, S. 5523–34. doi:10.1093/nar/gks215. PMID 22396526. PMC 3384331 (freier Volltext).
- Pietilä MK, Roine E, Sencilo A, Bamford DH, Oksanen HM: Pleolipoviridae, a newly proposed family comprising archaeal pleomorphic viruses with single-stranded or double-stranded DNA genomes. In: Archives of Virology. 161, Nr. 1, Januar 2016, S. 249–56. doi:10.1007/s00705-015-2613-x. PMID 26459284.
- Pietilä MK, Atanasova NS, Manole V, Liljeroos L, Butcher SJ, Oksanen HM, Bamford DH: Virion architecture unifies globally distributed pleolipoviruses infecting halophilic archaea. In: Journal of Virology. 86, Nr. 9, Mai 2012, S. 5067–79. doi:10.1128/JVI.06915-11. PMID 22357279. PMC 3347350 (freier Volltext).
- SIB: Double Strand RNA Viruses, auf: ViralZone
- SIB: Double Stranded RNA Viruses, auf: ViralZone
- Circulifer tenellus virus 1, auf: Virus-Host DB
- NCBI: Circulifer tenellus virus 1 (species)
- Henxia Xia et al.: A dsRNA virus with filamentous viral particled, in: Nature Communicationsvolume 8, Nr. 168 (2017), doi:10.1038/s41467-017-00237-9
- NCBI: camelliae filamentous virus 1 (species)
- Spissistilus festinus virus 1, auf: Virus-Host DB
- NCBI: Spissistilus festinus virus 1 (species)
- SIB: Positive Strand RNA Viruses, auf: ViralZone
- ICTV: Sarthroviridae, Virus Taxonomy: 2019 Release EC 51, Berlin, Germany, July 2019 (MSL #35)
- SIB: Macronovirus, auf: ExPASy: ViralZone
- SIB: Negative Strand RNA Viruses, auf: ViralZone
- SIB: Reverse Transcribing Viruses, auf: ViralZone