Totiviridae
Totiviridae ist eine Familie von RNA-Viren mit doppelsträngigem RNA-Genom.
| Totiviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Totiviridae | ||||||||||||||
| Links | ||||||||||||||
|
Eigenschaften

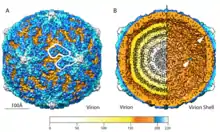
Die Viruspartikel (Virionen) der Totiviridae besitzen ein ikosaedrisches Kapsid ohne Virushülle von etwa 36 bis 40 nm Durchmesser mit einer Triangulationszahl von eins an der inneren Kapsidschicht und dreizehn an der äußeren.[3]

Die Genome der Totiviren lassen sich in zwei Typen einteilen: mit überlappenden Genen oder ohne Überlappung.[4] Das Genom besteht aus einer linearen doppelsträngigen RNA von 4,6 bis 6,7 kbp (Kilobasenpaaren) mit zwei offenen Leserastern (gag und pol), die unter der Kontrolle eines RNA-Pseudoknotens hergestellt werden.[4] Im ersten Typ werden die Proteine als Fusionsprotein aus beiden etwa 210 Basenpaare überlappenden Genen über eine + 1 oder - 1 Leserasterverschiebung hergestellt, während im zweiten Typ die Gene nicht überlappen und einzeln translatiert werden.[4] Das Gag-Protein ist das Kapsidprotein und das Pol-Protein ist eine RNA-Polymerase von etwa 190 Kilodalton.[3] Das Kapsid besteht aus dem Gag-Protein von etwa 100 Kilodalton, welches nach Acetylierung des N-Terminus zunächst asymmetrische Dimere bildet.[3][4] Die zusammengelagerten Kapsidproteine binden die RNA-abhängige RNA-Polymerase Pol, welche wiederum die RNA bindet, wodurch sich ein neugebildetes Virion zusammenfügt.[4] Manche Totiviren besitzen ein drittes offenes Leseraster. Totiviren sind vermutlich unabhängig von anderen RNA-Viren mit doppelsträngigem Genom aus RNA-Viren mit einzelsträngigem Genom positiver Polarität entstanden (s. u.).[3]
Systematik
Innere Systematik
Die folgende Gliederung in fünf Gattungen (Genera) und 28 Spezies der Totiviridae folgt den Vorgaben des International Committee on Taxonomy of Viruses (ICTV) mit Stand März 2020:[2]
- Familie Totiviridae
- Genus Giardiavirus
- Spezies Giardia lamblia virus (Typus)
- Genus Leishmaniavirus
- Spezies Leishmania RNA virus 1 (Typus)
- Spezies Leishmania RNA virus 2

- Genus Totivirus
- Spezies Saccharomyces cerevisiae virus L-A (Typus)
- Spezies Saccharomyces cerevisiae virus LBCLa
- Spezies Scheffersomyces segobiensis virus L
- Spezies Tuber aestivum virus 1
- Spezies Ustilago maydis virus H1
- Spezies Xanthophyllomyces dendrorhous virus L1A
- Spezies Xanthophyllomyces dendrorhous virus L1B
- Genus Trichomonasvirus
- Spezies Trichomonas vaginalis virus 1 (Typus)
- Spezies Trichomonas vaginalis virus 2
- Spezies Trichomonas vaginalis virus 3
- Spezies Trichomonas vaginalis virus 4
- Genus Victorivirus
- Spezies Aspergillus foetidus slow virus 1
- Spezies Beauveria bassiana victorivirus 1
- Spezies Chalara elegans RNA Virus 1
- Spezies Coniothyrium minitans RNA virus
- Spezies Epichloe festucae virus 1
- Spezies Gremmeniella abietina RNA virus L1
- Spezies Helicobasidium mompa totivirus 1-17
- Spezies Helminthosporium victoriae virus 190S (HvV190S, Typus)
- Spezies Magnaporthe oryzae virus 1
- Spezies Magnaporthe oryzae virus 2
- Spezies Rosellinia necatrix victorivirus 1
- Spezies Sphaeropsis sapinea RNA virus 1
- Spezies Sphaeropsis sapinea RNA virus 2
- Spezies Tolypocladium cylindrosporum virus 1
Äußere Systematik
Koonin et al haben 2015 die Totiviridae taxonomisch (aufgrund ihrer Verwandtschaft) einer von ihnen postulierten Supergruppe‚„Picornavirus-like superfamily“ zugeordnet.[5] Die Mitglieder dieser vorgeschlagenen Supergruppe gehören verschiedenen Gruppen der Baltimore-Klassifikation an, in der Regel handelt es sich um einzelsträngige RNA-Viren positiver Polarität ((+)ssRNA, Baltimore-Gruppe 4), es sind aber auch – wie die Birnaviridae – doppelsträngige Vertreter (mit dsRNA gekennzeichnet, Baltimore-Gruppe 3) zu finden. Dieser Vorschlag ist inzwischen abgelöst durch die Master species List #35 des ICTV vom März 2020[2] Eine Gegenüberstellung der Kladogramme findet sich bei Picornavirales §ICTV Master Species List #35.
Weblinks
- MeSH Totiviridae
- Viralzone: Totiviridae
- ICTV: Index of Viruses - Totiviridae. In: ICTVdB - The Universal Virus Database, version 4. Büchen-Osmond, C. (Ed), Columbia University, New York 2009. .
Einzelnachweise
- ICTV Master Species List 2018b v1 MSL #34, Feb. 2019
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- D. M. Knipe, Peter M. Howley, D. E. Griffin, (Hrsg.): Fields Virology. 5. Auflage, Lippincott Williams & Wilkins, Philadelphia 2007, ISBN 978-0-7817-6060-7.
- M. A. Hartley, C. Ronet, H. Zangger, S. M. Beverley, N. Fasel: Leishmania RNA virus: when the host pays the toll. In: Frontiers in cellular and infection microbiology. Band 2, 2012, S. 99, ISSN 2235-2988. doi:10.3389/fcimb.2012.00099. PMID 22919688. PMC 3417650 (freier Volltext).
- Eugene V. Koonin, Valerian V. Dolja, Mart Krupovic: Origins and evolution of viruses of eukaryotes: The ultimate modularity, in: Virology vom Mai 2015; 479-480. 2–25, Epub 12. März 2015, PMC 5898234 (freier Volltext), PMID 25771806