Lysozym
Lysozym (auch Muramidase) ist ein Enzym, das β-1,4-Glycosidische Bindungen zwischen N-Acetylmuraminsäure- (NAM) und N-Acetylglucosaminresten (NAG) in Peptidoglycanen, aus Zuckerderivaten und Peptiden aufgebauten Makromolekülen, hydrolysiert. Lysozyme kommen als Teil des angeborenen Immunsystems bei Tieren vor und können außerdem in Pflanzen, Pilzen, Bakterien und bei Bakteriophagen gefunden werden. Beim Menschen führen Mutationen im LYZ-Gen zu einer seltenen erblichen Form der Amyloidose.[1]
| Lysozym C (Gallus gallus) | ||
|---|---|---|
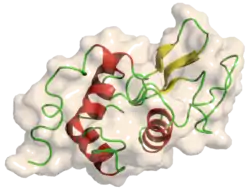
| ||
| Struktur von Lysozym nach PDB 132L | ||
|
Vorhandene Strukturdaten: siehe UniProt | ||
| Masse/Länge Primärstruktur | 14,3 kDa / 129 Aminosäuren | |
| Sekundär- bis Quartärstruktur | (β)αα(β)βββαα, Lysozym-ähnlich | |
| Bezeichner | ||
| Gen-Name(n) | LYZ | |
| Externe IDs |
| |
| Arzneistoffangaben | ||
| ATC-Code | D06BB07 J05AX02 | |
| Enzymklassifikation | ||
| EC, Kategorie | 3.2.1.17, Glycosidase | |
| Reaktionsart | Hydrolyse β-1,4-glycosidischer Bindungen zwischen NAM und NAG | |
| Substrat | Peptidoglycan | |
| Produkte | Spaltprodukte | |
| Vorkommen | ||
| Homologie-Familie | Lactalbumin/Lysozym | |
| Übergeordnetes Taxon | Schleimpilze, Schwämme, Bilateria, Pilze, Bakterien, Phagen | |
| Orthologe | ||
| Mensch | Hausmaus | |
| Entrez | 4069 | 17105 |
| Ensembl | ENSG00000090382 | ENSMUSG00000069516 |
| UniProt | P61626 | P08905 |
| Refseq (mRNA) | NM_000239 | NM_017372 |
| Refseq (Protein) | NP_000230 | NP_059068 |
| Genlocus | Chr 12: 69.35 – 69.35 Mb | Chr 10: 117.28 – 117.28 Mb |
| PubMed-Suche | 4069 | 17105 |
Geschichte
Zu Lysozymen gibt es mehrere tausend wissenschaftliche Publikationen. Hier wird auf einige der ersten Veröffentlichungen und auf solche, die über den Forschungsbereich Lysozyme hinaus von Bedeutung sind, kurz eingegangen.

Die antibakterielle Eigenschaft von Hühnereiklar, die auf Lysozym zurückzuführen ist, wurde erstmals 1909 von Laschtschenko beschrieben.[2] Der Begriff „Lysozym“ wurde allerdings erst 1922 durch Alexander Fleming (1881–1955) eingeführt, der damit dem Enzym den Namen gab.[3] Er beobachtete die antibakterielle Wirkung von Lysozym im Nasenschleim auf das Bakterium Micrococcus lysodeikticus und konnte diese Wirkung auch bei weiteren humanen Sekreten und Geweben feststellen.[3]
Das Lysozym aus Hühnereiklar war das erste vollständig sequenzierte Enzym, das alle kanonischen Aminosäurereste enthielt.[4] Die mit Röntgenstrukturanalyse bestimmte dreidimensionale Struktur von Lysozym aus Hühnereiklar (HEWL) wurde 1965 von David Chilton Phillips (1924–1999) erstmals beschrieben.[5][6] Es war die zweite Struktur eines Proteins, sowie die erste eines Enzyms, die durch Röntgenstreuung bestimmt werden konnte. Lysozym war auch das erste Enzym, anhand dessen Struktur[7][8] ein für das Enzym spezifischer und detaillierter Katalysemechanismus vorgeschlagen wurde.[9] Diese Arbeit lieferte eine Erklärung, wie Enzyme durch ihre Struktur die Geschwindigkeit einer chemischen Reaktion erhöhen. Der von Phillips vorgeschlagene Mechanismus wurde lange Zeit durch experimentelle Befunde gestützt und wurde erst 2001 auf Grund neuer Erkenntnisse überarbeitet.[10]
Klassifizierung
Die Zuordnung der Lysozyme zu den verschiedenen Typen erfolgt anhand ihrer Aminosäuresequenz, sowie aufgrund ihrer biochemischen und enzymatischen Eigenschaften. In der Klassifikation nach Henrissat[11][12][13][14][15] gehören Lysozyme zu den der Glycosidase Familien 22 bis 25 und lytische Transglycosidasen zu den Glycosidase-Familien 23, 102, 103 und 104.
Neben Lysozymen und lytischen Transglycosidasen gibt es weitere Peptidoglycan abbauende Enzyme, wie N-Acetyl-β-D-glucosaminidasen, N-Acetylmuramyl-L-alaninamidasen und verschiedene Endopeptidasen.[16]
Bei Tieren wurden drei sich voneinander unterscheidende Haupttypen von Lysozymen identifiziert, die gewöhnlich als c-Typ (chicken- oder conventional-type), g-Typ (goose-type) und als i-Typ (invertebrate-type) bezeichnet werden.[17] Daneben gibt es noch Lysozyme, die sich keinem der zuvor genannten Typen zuordnen lassen, wie beispielsweise die von Dictyostelium discoideum (UniProt Q8T1G4)[18] oder des Schwamms Suberites domuncula (UniProt Q50JA0).[19] Virale Lysozyme werde in v-Typ, λ-Typ, g-Typ und CH-Typ eingeteilt.[20] Einige virale Enzyme werden aus historischen Gründen noch als Lysozyme bezeichnet, obwohl diese keine β-1,4-N-Acetylmuramidasen sind. Die bei Pflanzen entdeckten Lysozyme haben alle eine Chitinase-Aktivität, aber nicht alle Chitinasen können Peptidoglycane hydrolysieren. Sie werden in h-Typ und g-Typ Chitinasen/Lysozyme eingeteilt. Bei vielen pflanzlichen Chitinasen wurde die Lysozym-Aktivität noch nicht untersucht und einige können aufgrund fehlender Sequenzinformationen nicht zugeordnet werden.[21] Lysozyme bei Bakterien werden funktionell in β-1,4-N-Acetylmuramidasen und in β-1,4-N,6-O-Diacetylmuramidasen unterschieden.[22]
Vorkommen
c-Typ Lysozyme
Der Mensch (UniProt P61626, PDB 1LZS), Säugetiere, Vögel (z. B. Huhn: UniProt P00698, PDB 1LZC), Reptilien, Amphibien und Fische produzieren Lysozyme vom c-Typ oder haben zumindest entsprechende Gene in ihrem Genom. Zu Reptilien und Amphibien liegen bisher aber kaum Informationen vor. Unter den bis 2009 vollständig sequenzierten Wirbellosen konnten nur bei einem Lanzettfischchen (Branchiostoma belcheri tsingtauense) sowie bei Gliederfüßern c-Typ Lysozym kodierende Gene identifiziert werden. Bei Schmetterlingen, Zweiflüglern, Termiten und Schnabelkerfen aus der Klasse der Insekten sowie bei Spinnentieren und Krebstieren wurden c-Typ Lysozyme gefunden und in allen bisher vollständig sequenzierten Insektengenomen konnte zumindest ein Gen gefunden werden, das homolog zu c-Typ Lysozymen ist.[17] Eine Untergruppe bilden die Calcium-Ionen-bindenden Lysozyme, zu denen beispielsweise das Lysozym aus Stutenmilch (UniProt P11376, PDB 2EQL) gehört. Diese Untergruppe ist vor allem bei der Diskussion um die evolutionäre Entstehung von α-Lactalbuminen von Interesse.[23] Bei einer Suche nach c-Typ Lyszym-ähnlichen Genen im humanen Genom können neun Gene gefunden werden, von denen eins für Lysozym C und eins für α-Lactalbumin kodiert. Vier andere werden überwiegend im Hoden exprimiert und scheinen bei der Fortpflanzung eine Rolle zu spielen. Für zwei dieser Gene konnte dies bei Mäusen gezeigt werden. Die anderen vier Gene wurden noch nicht charakterisiert.[24] Auch bei anderen Säugetieren können mehrere c-Typ Lyszym-ähnlichen Gene gefunden werden. Für die unterschiedlichen humanen Gene konnten mögliche orthologe Gene in den meisten Säugetiergenomen gefunden werden.[24]
g-Typ Lysozyme
Lysozyme vom g-Typ verdanken ihren Namen der erstmaligen Entdeckung im Eiklar der Emder Gans. Seitdem wurde es für mehrere Vogelarten wie Huhn, Trauerschwan, afrikanischer Strauß, Nandu und Kasuar charakterisiert. (Struktur eines g-Typ Lysozyms einer Graugans: UniProt P00718, PDB 154L). Bei Hühnervögeln dominiert das c-Typ Lysozym im Eiklar. Bei Gänsevögeln können abhängig von der Art im Eiklar c-Typ, g-Typ oder beide Typen von Lysozym vorkommen. Erst 2001 wurde gezeigt, dass g-Typ Lysozyme außerhalb der Klasse der Vögel vorkommen. Bei einer Datenbanksuche[25] konnten homologe Proteine beim Menschen, Säugetieren, Fischen (z. B. Kabeljau: UniProt B9TU22, PDB 3gxr) und Amphibien gefunden werden. Auch bei Wirbellosen, wie Jakobsmuschel, Auster und einigen Manteltieren wurden funktionelle Proteine gefunden. In den Genomen von Anopheles gambiae, Apis mellifera, Drosophila melanogaster, Drosophila pseudoobscura, Caenorhabditis briggsae und Caenorhabditis elegans existieren keine g-Typ Lysozyme.[17] Eins der beiden zu g-Typ Lysozym homologen Proteine des Menschen wird in der Niere exprimiert. Das andere wird in der Tränendrüse und in geringen Maß im Hoden exprimiert und ist aktiv.[26]
i-Typ Lysozyme
Bisher konnten i-Typ Lysozyme in den Stämmen Weichtiere (z. B. bei einer Venusmuschel: UniProt Q8IU26, PDB 2DQA), Ringelwürmer, Stachelhäuter, Fadenwürmer und Gliederfüßer gefunden werden. In allen verfügbaren Genomen von Wirbeltieren fehlen sie.[17]
v-Typ Lysozyme
Bis 1996 wurden 13 Bakteriophagen mit Lysozymen vom v-Typ beschrieben. Sieben der Bakteriophagen infizierten gramnegative Bakterien, die sechs anderen grampositive Bakterien. Im T4-Phagen sind beispielsweise drei Lysozyme codiert, von denen das Produkt des Gens e, das am besten untersuchte Phagen-Lysozym ist (UniProt P00720, PDB 148L). Das etwa 18 kDa große Lysozym zerstört die Zellwand von Escherichia coli am Ende des Infektionszyklus, nachdem die innere Membran durch ein anderes Phagenprotein modifiziert wurde. Das Lysozym hydrolysiert die glycosidische Bindung hinter N-Acetylmuraminsäureresten, die mit einer Peptid-Seitenkette modifiziert sind. Die N-Acetamido-Gruppe ist für die Aktivität des Lysozyms ebenfalls erforderlich.[20] Eine Struktur von T4-Lysozym mit Substrat wurde von R. Kuroki veröffentlicht.[27] Ein Überblick über die Studien mit T4-Lysozym wird im Review von W. A. Baase u. a.[28] gegeben. Die zahlreichen Studien an T4-Lysozym geben Einblicke in Faltung und Stabilität von Proteinen im Allgemeinen und T4-Lysozym im Speziellen.[28]
λ-Typ Lysozyme
Die λ-Typ Lysozyme sind nach dem Lysozym des λ-Phagen benannt (UniProt P03706, PDB 3D3D). Das Lysozym des λ-Phagen ist keine Hydrolase, sondern eine lytische Transglycosidase.[29]
g-Typ Lysozyme
Virale g-Typ Lysozyme, wie beispielsweise das des Enterobacteria Phagen PRD1 (UniProt P13559) sind in ihrer Sequenz teilweise ähnlich den tierischen g-Typ Lysozymen.[20]
h-Typ Chitinasen/Lysozyme
Die Bezeichnung dieses Typs geht auf zwei als Hevamine bezeichnete Enzyme zurück (UniProt P23472, PDB 1HVQ), die in speziellen Vakuolen des Kautschukbaums Hevea brasiliensis und im Naturkautschuk vorkommen und bereits 1976 isoliert wurden. Für diese Enzyme wurde 1983 eine Lysozymaktivität und 1990 eine Chitinaseaktivität nachgewiesen. Homologe Chitinasen wurden inzwischen bei z. B. bei Acker-Schmalwand Arabidopsis thaliana, Gurke, Tabak, sowie bei dem Hefepilz Saccharomyces cerevisiae und anderen Pilzen im Genom nachgewiesen. Für die Chitinase aus der Gurke konnte jedoch keine Lysozymaktivität festgestellt werden.[21] Die h-Typ Chitinasen werden anhand der Sequenz in Klasse III und Klasse V Chitinasen unterschieden. In der Klassifikation nach Henrissat gehören h-Typ Chitinasen zur Glycosidase-Familie 18 (GH18), die weit verbreitet bei Archaeen, Prokaryoten und Eukaryoten vorkommt.[30]
b-Typ Chitinasen/Lysozyme
Diese Chitinasen weisen in ihrer Aminosäuresequenz keine Ähnlichkeiten zu h-Typ Chitinasen auf. Sie können anhand ihrer Aminosäuresequenz in drei Klassen eingeteilt werden (I, II und IV). Die Bezeichnung geht auf eine Klasse I Chitinase aus Gerste (englisch barley) zurück (UniProt P23951, PDB 2BAA). Für diese und einige andere Klasse I Chitinasen wurde eine Lysozymaktivität nachgewiesen. Die spezifische Lysozymaktivität ist jedoch deutlich geringer als bei aktiven h-Typ Chitinasen oder HEWL. Bei anderen Klasse I und Klasse II Chitinasen war die spezifische Lysozymaktivität etwa vier Größenordnungen geringer als bei HEWL oder konnte nicht festgestellt werden. Zur Lysozymaktivität von Klasse IV Chitinasen wurden noch keine Daten veröffentlicht.[21] Das "Lysozym" aus Papaya (PDB 3CQL) hat spezifische Lysozymaktivität von 35 % im Vergleich zu HEWL.[21] In der Klassifikation nach Henrissat gehören b-Typ Chitinasen zur Glycosidase-Familie 19 (GH19), die außer bei Pflanzen nur bei Actinobacteria, grünen Nichtschwefelbakterien, Purpurbakterien, einigen Gliederfüßern und Fadenwürmern zu finden ist.[31]
Weitere Chitinasen/Lysozyme aus Pflanzen
Für das Lysozym des Amate-Feigenbaums Ficus glabrata (spezifische Lysozymaktivität 85 % im Vergleich zu HEWL) liegt zwar eine detaillierte Beschreibung der enzymatischen Eigenschaften vor, aufgrund der fehlenden Aminosäuresequenz ist eine Zuordnung jedoch noch nicht möglich.[21]
CH-Typ Lysozyme
Das Lysozym des Pilzes Chalaropsis sp. (UniProt P00721) ist eine β-1,4-N,6-O-Diacetylmuramidase, die auch O-acetyliertes Peptidoglycan hydrolysieren kann. Verwandte Lysozyme wurden bei einigen Streptomyces-Arten (UniProt P25310, PDB 1JFX), bei Lactobacillus acidophilus und bei Clostridium acetobutylicum (UniProt P34020) und mehreren Bakteriophagen (z. B. Lactobacillus Phage MV1: UniProt P33486 und Streptococcus Phage Cp-1: UniProt P15057) gefunden.[20][22]
Biologische Bedeutung
Funktion der Lysozyme bei Tieren und Menschen
Bei Säugetieren wie dem Menschen kommt Lysozym in vielen Sekreten, wie Tränenflüssigkeit, Speichel, den Sekreten des Atemtrakts, im Blutserum, in der Zerebrospinalflüssigkeit, im Fruchtwasser, im Zervixschleim und in der Milch vor. Es wird in den Geweben des Atemtrakts, in den Nieren und in der Darmschleimhaut sowie von neutrophilen Granulozyten und Makrophagen produziert.[17][32] Unter physiologischen Bedingungen sind etwa 80 % des Lysozyms im Blutplasma auf den Abbau von neutrophilen Granulozyten zurückzuführen.[33] Monozyten und Makrophagen können Lysozym aktiv sezernieren. Lysozym ist ein Bestandteil des angeborenen Immunsystems und dient der Abwehr von Bakterien. Die Peptidoglycanzellwand grampositiver Bakterien kann durch Lysozym direkt angegriffen werden. Bei gramnegativen Bakterien kann die äußere Membran durch weitere Komponenten des angeborenen Immunsystems, wie Lactoferrin, Defensine und Cathelicidine durchlässig gemacht werden, so dass diese auch durch Lysozym angegriffen werden können. Neben der direkten antibakteriellen Wirkung führt die Freisetzung von Peptidoglycanfragmenten zu einer Modulation des Immunsystems über Peptidoglycan-erkennende-Rezeptoren. Über die Funktion von g-Typ-Lysozymen bei Säugetieren liegen bisher keine experimentellen Studien vor.[17] Lysozyme bei Vögeln werden überwiegend in den Eiern gefunden. Für Hühner konnte zwar die Expression verschiedener Lysozyme in Darmepithelzellen nachgewiesen werden, so dass eine Schutzwirkung gegen pathogene Bakterien im Darm vermutet werden kann. Ein direkter Nachweis dieser Funktion liegt jedoch noch nicht vor.[17] Bei Fischen konnte Lysozym-Aktivität in der Kopfniere, dem Schlüsselorgan des Fischimmunsystems, der Milz, den Kiemen, im Blut, der Haut, dem Verdauungstrakt und in Fischeiern nachgewiesen werden. Für mehrere c- und g-Typ-Lysozyme konnte eine antibakterielle Wirkung gegen grampositive und gramnegative Bakterien gezeigt werden. Vor allem die hohe Expression von g-Typ-Lysozymen in der Haut, den Kiemen und dem Darmepithel sprechen für eine Abwehrfunktion.[17] Lysozym konnte zwar aus dem Eiklar von Reptilieneiern isoliert werden. Darüber hinaus liegen aber keine Informationen über Vorkommen und Funktion vor.[17] Insekten haben eine effektive induzierbare Immunabwehr. Sind sie Bakterien ausgesetzt, produzieren sie in der Hämolymphe eine Reihe antibakterieller Peptide und Proteine, zu denen auch Lysozyme gehören.[17]
Einige Tiere verwenden Lysozyme als Verdauungsenzyme, um Bakterien als Nahrungsquelle zu nutzen. Diese Lysozymarten zeigen zum Teil eine erhöhte Resistenz gegenüber Proteasen und ein pH-Optimum im sauren Bereich.
Die Struktur und Eigenschaften des im Kuhmagen produzierten Lysozyms wurde von Nonaka u. a. untersucht.[34]
Funktion der Lysozyme bei Pflanzen
Da bisher alle pflanzlichen Lysozyme auch eine Chitinase-Aktivität aufweisen, diese ein breiteres pH-Optimum aufweist und die Lysozym-Aktivität zum Teil deutlich geringer ist oder ganz fehlt, sind diese Proteine möglicherweise zur Abwehr von Pilzen gedacht.
Funktion der Lysozyme bei Bakteriophagen
Für die Infektion von Bakterien hat sich bei Bakteriophagen eine Vielzahl von Methoden entwickelt. In einigen Fällen sind daran auch Lysozyme beteiligt, die durch einen lokal begrenzen Abbau des Peptidoglycans eine Infektion ermöglichen. Mit einem großen Überschuss an Phagenpartikeln konnte in diesen Fällen auch eine "Lyse von außen" beobachtet werden. Für die Freisetzung von Phagenpartikeln am Ende eines Infektionszyklus sind zwei Strategien bekannt. Eine ohne Lysozyme, bei denen durch ein Porin Löcher in der Membran gebildet werden, über die die Phagenpartikel austreten können. Dies ist beispielsweise bei den E. coli Phagen MS2 und фX174 der Fall. Bei der zweiten Strategie, die bei größeren Phagen wie beispielsweise T4-, T7- oder λ-Phagen zum Einsatz kommt, werden neben dem Porin noch Lysozyme und/oder Amidasen produziert, die entweder die Polysaccharide oder die quervernetzenden Peptide im Peptidoglycan hydrolysieren. Die Endolysine können im Cytoplasma gelöst vorliegen, wie beim T4- oder λ-Phagen. Es gibt auch Phagen, wie den Enterobakteria-Phagen P1, bei denen das Endolysin (UniProt Q37875) über das Sec-System des Wirts in das Periplasma transportiert wird und in inaktivierter Form (PDB 1XJU) an die Membran gebunden vorliegt. Eine Depolarisation der Membran durch ein Holin führt zu einer Aktivierung des Endolysins (PDB 1XJT) und zur Zelllyse.[35]
Funktion der Lysozyme bei Bakterien
Für Wachstum und Teilung von Bakterienzellen sind nicht nur Enzyme erforderlich, die das Peptidoglycan aufbauen, sondern auch welche, die dieses lokal wieder abbauen können. Diese müssen gut kontrollierbar und in ihrer Aktivität regulierbar sein. Aufgrund ihrer potentiellen Gefährlichkeit für die bakterielle Zelle werden sie als Autolysine bezeichnet. Dazu gehören auch einige bakterielle Lysozyme, beispielsweise das Streptomyces-Lysozym.[36] Arbeiten an der lytischen Transglycosylase "Slt35" (UniProt P41052, PDB 1d0k) von E. coli[37] und den N-Acetylglucosaminidasen von Enterococcus hirae (UniProt P39046) und Listeria monocytogenes (UniProt Q8Y842, PDB 3fi7)[38], die strukturell Lysozymen ähneln geben einen Einblick in die Regulation und Funktion bakterieller Peptidoglycan-Hydrolasen. Einen Überblick gibt der Review von S. Layec.[39] Lytische Transglycosylasen scheinen eine bedeutende Gruppe bakterieller Autolysine zu sein. Sie kommen ubiquitär in Eubakterien, mit Ausnahme der Mykoplasmen, vor. Anhand von Sequenz und Konsensusmotiven werden vier Familien unterschieden, die für unterschiedliche Funktionen benötigt werden. Soweit bekannt ist haben bakterielle lytische Transglycosylasen eine exo-Aktivität, d. h., sie spalten Disaccharidanhydromuropeptide von reduzierenden oder nichtreduzierenden Enden des Peptidoglycans ab. Sie sind am Zellwachstum und an der Zellteilung beteiligt. Sie ermöglichen den Einbau von Proteinkomplexen wie Flagellen und Pili, die in die Peptidoglycanzellwand integriert werden.[40]
Funktion der Lysozyme bei Pilzen
Da das Lysozym des Pilzes Chalaropsis sp. als extrazelluläres bakteriolytisches Enzym produziert wird,[1] ist eine Wirkung als Bakteriocin wahrscheinlich.
Aktivitätsbestimmung
Das natürliche Substrat von Lysozymen ist ein unlösliches Peptidoglycanpolymer mit einer hohen molaren Masse, das meist die Zellwand von Bakterien verstärkt, damit diese dem hohen Zellinnendruck widerstehen. Zur Messung der Lysozymaktivität wird häufig dieses große Biopolymer eingesetzt. Dazu werden beispielsweise getrocknete und UV-inaktivierte Zellen von Micrococcus luteus eingesetzt. Aufgrund der Unlöslichkeit und Komplexität des Substrats können die Reaktionskinetiken nicht als Michaelis-Menten Kinetik beschrieben werden.
Für spezifische Messungen der Lysozymaktivität müssen entweder spezielle Substrate eingesetzt oder die Produkte untersucht werden. Um Probleme mit der Unlöslichkeit und Heterogenität des Substrats bei Zellen oder Zellwandpräparationen zu umgehen, werden auch lösliche Oligosaccharide und Muropeptide eingesetzt.[41]
Gewöhnlich wird eine der folgenden drei unspezifische Methoden eingesetzt.[41]
Trübungsmessung (Lichtstreuung)
Die Abnahme der optischen Dichte einer trüben Zellsuspension wird photometrisch bei 650, 520 oder 450 nm verfolgt.[41]
Viskositätsmessung
Die Zunahme der Viskosität infolge der Depolymerisation der unlöslichen Peptidoglycane in lösliche Glycopeptide wird verfolgt.[41]
Lysoplate Assay
Abgetötete Bakterienzellen werden zu einer einprozentigen Agarlösung gegeben und diese in Petrischalen ausplattiert. Nach dem Erstarren der Agarlösung werden Vertiefungen in den Agar gedrückt und in diese die Enzymproben gegeben. Infolge der Enzymwirkung bilden sich um die Vertiefung transparente Zonen in dem zuvor trüben Agar. Der Durchmesser dieser Zonen wird bestimmt.[41]
Weitere Methoden
Analog zu den Trübungsmessungen gibt es Messungen mit radioaktiven oder farbmarkierten Substraten.[41] Diese sind auch unspezifisch.
Resistenzmechanismen
N-Deacetylierung und O-Acetylierung sind häufige Modifizierungen von Peptidoglycan bei pathogenen Bakterien, die zumindest bei Listeria monocytogenes und Staphylococcus aureus zur Lysozymresistenz beitragen. Eine weitere Strategie ist die Produktion von Lysozym-Inhibitoren, wie im Periplasma von E. coli (UniProt P0AD59, PDB 1GPQ) oder an die äußere Membran gebunden wie bei Pseudomonas aeruginosa (UniProt Q9I574, PDB 3F6Z).[17]
Eigenschaften
Tierische Lysozyme sind globuläre Proteine, die durch eine große Substratbindungsspalte in zwei Domänen geteilt sind, eine größere α-helikale Domäne, in der sich Amino- und Carboxyterminus des Proteins befinden, und eine kleinere β-Faltblatt-Domäne. Diese Art gemischter α+β-Faltung wird als Lysozym-ähnliche Faltung bezeichnet.[42] In c-Typ-Lysozymen befinden sich acht konservierte Cysteinreste, die vier Disulfidbrücken ausbilden. In g-Typ-Lysozymen bei Säugetieren und Vögeln befinden sich vier bis sieben Cysteinreste, bei einigen Fischen jedoch nur ein bzw. kein Cysteinrest, und bei Wirbellosen sechs bis dreizehn Cysteinreste, von denen jedoch keiner denen bei Säugetieren und Vögeln entspricht. Über die Gegenwart und Lage von Disulfidbrücken bei g-Typ-Lysozymen von Wirbellosen ist noch nichts bekannt. Ein hoher Anteil von Cysteinresten scheint bei i-Typ-Lysozymen typisch zu sein. Im Lysozym der Venusmuschel Venerupis philippinarum bilden beispielsweise alle 14 Cysteinreste Disulfidbrücken aus.[43] Die tierischen c- und i-Typ-Lysozyme sind etwa 11 bis 15 kDa groß, g-Typ-Lysozyme etwa 20 bis 22 kDa. Die anhand der Sequenz berechneten isoelektrischen Punkte der c- und g-Typ-Lysozyme liegen im basischen Bereich, der der i-Typ-Lysozyme sind variabler. An der Verdauung beteiligte c- und i-Typ-Lysozyme haben meist einen pI im neutralen oder sauren Bereich.
CH-Typ-Lysozyme und h-Typ-Chitinasen haben Strukturen eines α/β-Hydrolase-Faltungstyps[44], der der TIM-barrel-Faltung[45] zugeordnet werden kann. Sie sind etwa 22 bis 40 kDa groß, wobei die größeren neben der Lysozym-Domäne auch mehrere unabhängig faltende Domänen besitzen, die eine Substrat-bindende Funktion haben, wie beispielsweise beim Streptococcus-Phagen Cp-1. Modulare Proteine können auch bei Bakterien gefunden werden.
Die viralen v-, λ- und g-Typ-Lysozyme sind häufig etwa 17 bis 19 kDa groß. Es gibt auch größere an Viruspartikel gebundene Lysozyme, die an der Infektion beteiligt sind.
Reaktionsmechanismen
Muramidase-Aktivität am Beispiel von HEWL
HEWL ist eine Endoglycosidase, die in Peptidoglycan β-1,4-glycosidische Bindungen zwischen NAM- und NAG-Resten hydrolysiert, wobei die anomere Konfiguration am C1 des NAM-Restes erhalten bleibt. Peptidoglycan wird in der Bindungsspalte zwischen α-helikaler und β-Faltblatt-Domäne gebunden, die sechs Saccharideinheiten lang ist. Die Bindungsstellen der einzelnen Zuckerreste werden mit A(-4), B(-3), C(-2), D(-1), E(+1) und F(+2) bezeichnet. Aus sterischen Gründen passt in die Bindungsstelle C(-2) kein N-Acetylmuraminsäurerest, so dass sich die NAM-Reste bei der Bindung an Peptidoglycan in den Positionen B(-3), D(-1) und F(+2) befinden müssen. Infolge der Bindung entsteht ein Knick in der Polysaccharidkette, wodurch wahrscheinlich die Konformation des NAM-Restes in Position D(-1) in eine Halbsessel-Konformation gezwungen wird. Das Enzym hydrolysiert die β-1,4-glycosidische Bindung zwischen Position D(-1) und E(+1). Im ersten Schritt wird durch den Glutaminsäurerest Glu35 des HEWL der Sauerstoff der glycosidischen Bindung protoniert und der in den Positionen E(+1) und F(+2) gebundene Teil des Substrates könnte sich lösen und weg diffundieren.
Nach dem von David C. Phillips postulierten Mechanismus würde dabei als Intermediat aus dem NAM-Rest ein Glycosyloxocarbenium-Ion entstehen, dessen α-Seite durch den Aspartatrest Asp52 des HEWL abgeschirmt und stabilisiert wird. In einem zweiten Schritt würde einem Wassermolekül, durch den jetzt vorliegenden Glutamatrest Glu35 ein Proton entzogen und das entstehende Hydroxidion das C1-Atom des NAM-Restes von der β-Seite nukleophil angreifen. Das zweite Hydrolyseprodukt würde entstehen, wobei die Konfiguration des anomeren Zentrums am C1 des NAM-Restes erhalten blieb.[9]
Durch neuere Arbeiten konnte gezeigt werden, dass das Oxocarbenium-Ion kein Intermediat, sondern den Übergangszustand hin zu einem kovalent über den Aspartatrest Asp52 gebundenen Intermediat darstellt. Dieses kovalent gebundene Intermediat wird analog zum ursprünglich formulierten Reaktionsmechanismus von einem Hydroxidion von der β-Seite am C1-Atom nukleophil angegriffen. Die ursprüngliche Konfiguration des anomeren Zentrums am C1 des NAM-Restes wird durch Bildung des zweiten Hydrolyseproduktes wiederhergestellt.[10]
Muramidase-Aktivität bei g-Typ Lysozymen und T4-Lysozym
Im Gegensatz zur Hydrolysereaktion durch HEWL geht die Hydrolyse bei g-Typ-Lysozymen von Gans (GEWL) und Kabeljau (gLYS) mit einer Inversion des anomeren Zentrums am C1-Atom des N-Acetylmuraminsäurerest einher. Analog zum Glu35 (HEWL) ist der Glutaminsäurerest Glu73 (GEWL&gLYS) essentiell für die Hydrolyse, ein zweiter saurer Aminosäurerest wie Asp52 (HEWL) in der Nähe des C1-Atoms des NAM-Restes in Position D(-1) fehlt jedoch bei den beiden g-Typ Lysozymen. Stattdessen wird wahrscheinlich durch zwei Aspartatreste(Asp86/97 in GEWL; Asp90/101 in gLYS) ein Wassermolekül in der Nähe des C1-Atoms positioniert, das die Funktion des Asp52 (HEWL) übernimmt.[46] Bei T4L ist Glu11 der essentielle Glutaminsäurerest, der Aspartatrest Asp20 und der Threoninrest Thr26 binden ein Wassermolekül in der Nähe des C1-Atoms, das dieses angreifen kann.[47]
Transglycosilierungsaktivität
Bei HEWL kann das kovalent gebundene Intermediat anstatt mit Wasser auch mit Sacchariden reagieren, so dass es an Stelle einer Hydrolyse zu einer Transglycosylierung kommt.[10] Durch einen Austausch des Threoninrestes Thr26 zu einem Histidinrest im T4-Lysozym wird dieses von einer invertierenden Muramidase in eine Transglycosidase umgewandelt.[47]
Lytische Transglycosylase
Lytische Transglycosylasen spalten die gleiche Bindung wie N-Acetylmuramidasen, bilden 1,6-anhydro-N-Acetylmuraminsäure als Produkt.[29] Strukturen, die einen Einblick in den Reaktionsmechanismus geben, liegen zum Beispiel für gp144[48] und Slt35[37] vor.
Chitinase-Aktivität von HEWL
HEWL kann, wie eine Reihe anderer Lysozyme auch, die β-1,4-glycosidischen Bindungen in Chitin und in den aus Chitin hergestellten wasserlöslichen Oligosacchariden (Chitodextrinen) hydrolysieren.
Weitere Funktionen
Neben der Lysozym-Aktivität konnte bei einigen i-Typ Lysozymen eine Isopeptidase-Aktivität festgestellt werden. Sie hydrolysieren die Isopeptidbindungen, die zwischen der γ-Carboxamidgruppe eines Glutaminrestes und der ε-Aminogruppe eines Lysinrestes gebildet wurde. Diese werden zum Beispiel zwischen Fibrin-Molekülen durch den Fibrinstabilisierender Faktor gebildet. Diese Aktivität wurde beispielsweise beim Blutegel nachgewiesen und verhindert, dass das Blut koaguliert. Ob diese Funktion auch bei anderen Wirbellosen von Bedeutung ist oder sogar die Isopeptidbindungen einiger Peptidoglycane gespaltet wird, ist noch unklar.[17]
Gewinnung

HEWL wird aus dem Eiklar von Hühnereiern gewonnen und gefriergetrocknet gehandelt. Mehr als 100 t werden jährlich produziert. Auch das Lysozym von Streptomyces coelicolor wird durch Submersfermentation in größeren Mengen gewonnen und in den Handel gebracht (Cellosyl ®). Humanes Lysozym wird aus neutrophilen Granulozyten, aus Milch und rekombinant, beispielsweise aus gentechnisch verändertem Reis, gewonnen. Auch die Gewinnung von humanem Lysozym C aus der Milch von Kühen ist inzwischen möglich.[49]
Verwendung
In der Lebensmittelindustrie wird Lysozym zur Konservierung oder beispielsweise in der Weinbereitung zur Kontrolle des biologischen Säureabbaus (Malolaktische Gärung) eingesetzt. Als Konservierungsmittel ist es in der EU als Lebensmittelzusatzstoff der Nummer E 1105 für gereiften Käse und zur Konservierung von Bier, das weder pasteurisiert noch sterilfiltriert wurde, zugelassen. Beim Käse wird die Rissbildung in der Käsekrume (sog. Spätblähung) durch Clostridium tyrobutyricum oder Clostridium botulinum verhindert.[50][51]
Beim Bier soll die Entstehung von Milchsäurebakterien verhindert werden. Die meisten Brauereien unterziehen ihr Bier dem Verfahren der Sterilfiltration oder der Pasteurisierung, um zu verhindern, dass es während der Lagerung vor dem Verzehr zu bakteriellem Verderb kommt. Bei einigen Bierspezialitäten, wie obergärigen, nachgärenden Bieren, z. B. Fass- oder Flaschenbier, können diese Verfahren nicht angewandt werden, weil die vorhandenen lebensfähigen Mikroorganismen bei diesen Bieren Teil des Herstellungsverfahrens sind.[52]
Ein in der Entwicklung befindlicher Anwendungsbereich ist der Einsatz von HEWL in sogenannten „aktiven“ Lebensmittelverpackungen, um die Haltbarkeit bestimmter Lebensmittel zu verlängern.[16] Es gibt transgene Ziegen, Schweine[53] und Rinder,[49] die humanes Lysozym in ihrer Milch produzieren und von denen man sich eine verbesserte Tiergesundheit, eine erhöhte Lebensmittelsicherheit und Haltbarkeit verspricht.[16]
Lysozym wird zum Aufschluss von Bakterienzellen eingesetzt. Zum Aufschluss gramnegativer Bakterien mit HEWL kann EDTA zur Permeabilisierung der Außenmembran hinzugegeben werden.
Außerdem kann die heterologe Expression von humanem Lysozym in Pflanzen zu einer gesteigerten Resistenz gegen Bakterien und Pilzen führen[54].
Klinische Bedeutung
Es wird ein Zusammenhang zwischen reduzierter Lysozymaktivität und bronchopulmonaler Dysplasie bei Neugeborenen vermutet.[55] Kleinkinder, in deren Nahrung Lysozym fehlt, erkranken häufiger an Durchfallerkrankungen.[56] Defensine und Lysozym schützen die Bindehaut des Auges vor Bakterien, so dass Defizite zu Bindehautentzündungen führen können. Mutationen am LYZ-Gen können zur sehr seltenen familiären Amyloidose führen.
Erhöhte Lysozymkonzentrationen im Blutserum treten bei chronischen bakteriellen Infektionen, wie zum Beispiel Tuberkulose[57], bei Sarkoidose[58], Morbus Crohn[59], AIDS[60], rheumatoider Arthritis[61] sowie bei monocytischer und monomyelocytischer Leukämie[62] auf. Bei Harnwegsinfektionen, bestimmten Nierenschäden und bei exzessiver endogener Lysozymproduktion, die die Reabsorptionskapazität des proximalen Tubulus überschreitet, sind erhöhte Lysozymwerte im Harn nachweisbar. Bei bakterieller Meningitis und bei Tumoren des zentralen Nervensystems können erhöhte Werte in der Zerebrospinalflüssigkeit nachgewiesen werden. Bei bakterieller Meningitis sind beispielsweise 5- bis 400-fach erhöhte Werte feststellbar.[59] Bei entzündlichen Erkrankungen des Magen-Darm-Trakts, wie beispielsweise Morbus Crohn, bei Gastroenteritis durch bakterielle oder Rotavirusinfektionen und bei Darmkrebs wurden erhöhte Lysozymmengen in Kot nachgewiesen. Auch im Speichel kann die Lysozymkonzentration erhöht sein.[63] Die Lysozymkonzentration bzw. -menge kann in diesen Fällen als Marker dienen, um den Krankheitsverlauf zu verfolgen, den Therapieerfolg zu beurteilen und Rückfälle festzustellen.
In den Publikationen von Brouwer u. a. (1984)[64] und Porstmann u. a. (1989)[59] wurden für Lysozym folgende Referenzbereiche angegeben.
Serum
- Neugeborene 800–4600 µg/l
- Erwachsene(18–40 Jahre) 450–2950 µg/l
- Erwachsene(41–70 Jahre) 1100–2900 µg/l
- Erwachsene(18–60 Jahre) 950–2450 µg/l
Urin
- Frauen 0,45–20,1 µg/l
- Männer 0,33–6,4 µg/l
- 1,7–123 µg/l
Zerebrospinalflüssigkeit
- 17,6–118 µg/l
Kot
- 0,04–1,5 µg/g
Die therapeutische Wirksamkeit von Lysozym (HEWL) beruht einerseits auf seiner enzymatischen Aktivität, die es erlaubt, das Wachstum empfindlicher Bakterien zu kontrollieren. Darüber hinaus moduliert Lysozym die Immunantwort bei Infektionen, stimuliert das Immunsystem und wirkt entzündungshemmend. Dadurch wirkt es gegen virale Infektionen, verstärkt die Wirkung von Antibiotika und kann bei der Krebsbehandlung und bei einem geschwächten Immunsystem eingesetzt werden. Lysozym wird meist oral verabreicht.[65] Lysozym ist in manchen Halsschmerztabletten enthalten. Allerdings fehlt hier ein echter Wirknachweis.[66] Lysozym aus Hühnereiern kann bei bestehender Hühnereiweißallergie eine Reaktion auslösen.[67]
Literatur
- P. Jollès (Hrsg.): Lysozymes: Model Enzymes in Biochemistry and Biology. (= Experientia Supplementum (EXS). Vol. 75). Birkhäuser, Basel/ Boston/ Berlin 1996, ISBN 978-3-7643-5121-2. (Beinhaltet folgende Reviews: PMID 8765291, PMID 8765292, PMID 8765293, PMID 8765294, PMID 8765295, PMID 8765296, PMID 8765297, PMID 8765298, PMID 8765299, PMID 8765300, PMID 8765301, PMID 8765302, PMID 8765303, PMID 8765304, PMID 8765305, PMID 8765306, PMID 8765307, PMID 8765308, PMID 8765309, PMID 8765310, PMID 8765311, PMID 8765312)
Einzelnachweise
- Pierre Jollès, Jacqueline Jollès: What’s new in lysozyme research? Always a model system, today as yesterday. In: Molecular and Cellular Biochemistry. Band 63, Nr. 2, September 1984, S. 165–189, doi:10.1007/BF00285225, PMID 6387440.
- P. Laschtschenko: Über die keimtötende und entwicklungshemmende Wirkung von Hühnereiweiß. In: Zeitschrift für Hygiene und Infektionskrankheiten. Band 64, Nr. 1, Dezember 1909, S. 419–427, doi:10.1007/BF02216170.
- A. Fleming: On a Remarkable Bacteriolytic Element Found in Tissues and Secretions. In: Proceedings of the Royal Society B: Biological Sciences. Band 93, Nr. 653, 1. Mai 1922, S. 306–317, doi:10.1098/rspb.1922.0023.
- Robert E. Canfield: The Amino Acid Sequence of Egg White Lysozyme. In: The Journal of biological chemistry. Band 238, Nr. 8, August 1963, S. 2698–2707, PMID 14063294 (jbc.org).
- C. C. F. Blake, D. F. Koenig, G. A. Mair, A. C. T. North, D. C. Phillips, V. R. Sarma: Structure of Hen Egg-White Lysozyme: A Three-dimensional Fourier Synthesis at 2 Å Resolution. In: Nature. Band 206, Nr. 4986, 22. Mai 1965, S. 757–761, doi:10.1038/206757a0, PMID 5891407.
- Louise N. Johnson, David C. Phillips: Structure of Some Crystalline Lysozyme-Inhibitor Complexes Determined by X-Ray Analysis At 6 Å Resolution. In: Nature. Band 206, Nr. 4986, 22. Mai 1965, S. 761–763, doi:10.1038/206761a0, PMID 5840126.
- C. C. F. Blake, G. A. Mair, A. C. T. North, D. C. Phillips, V. R. Sarma: On the Conformation of the Hen Egg-White Lysozyme Molecule. In: Proceedings of the Royal Society B: Biological Sciences. Band 167, 1009, A Discussion on the Structure and Function of Lysozyme, 18. April 1967, S. 365–377, doi:10.1098/rspb.1967.0034.
- C. C. F. Blake, G. A. Mair, A. C. T. North, D. C. Phillips, V. R. Sarma: Crystallographic Studies of the Activity of Hen Egg-White Lysozyme. In: Proceedings of the Royal Society B: Biological Sciences. Band 167, 1009, A Discussion on the Structure and Function of Lysozyme, 18. April 1967, S. 378–388, doi:10.1098/rspb.1967.0034.
- David C. Phillips: The hen egg-white lysozyme molecule. In: Proceedings of the National Academy of Sciences. Band 57, Nr. 3, 1. März 1967, S. 484–495 (pnas.org).
- David J. Vocadlo, Gideon J. Davies, Roger Laine, Stephen G. Withers: Catalysis by hen egg-white lysozyme proceeds via a covalent intermediate. In: Nature. Band 412, Nr. 6849, 23. August 2001, S. 835–838, doi:10.1038/35090602, PMID 11518970.
- Bernard Henrissat: A classification of glycosyl hydrolases based on amino-acid sequence similarities. In: Biochemical Journal. Band 280, Nr. 2, 1. Dezember 1991, S. 309–316, PMID 1747104, PMC 1130547 (freier Volltext).
- Bernard Henrissat, Amos Bairoch: New families in the classification of glycosyl hydrolases based on amino- acid sequence similarities. In: The Biochemical journal. Band 293, Nr. 3, 1. August 1993, S. 781–788, PMID 8352747, PMC 1134435 (freier Volltext).
- Gideon Davies, Bernard Henrissat: Structures and mechanisms of glycosyl hydrolases. In: Structure. Band 3, Nr. 9, September 1995, S. 853–859, doi:10.1016/S0969-2126(01)00220-9.
- Bernard Henrissat, Amos Bairoch: Updating the sequence-based classification of glycosyl hydrolases. In: The Biochemical journal. Band 316, Nr. 2, 1. Juni 1996, S. 695–696, PMID 8687420, PMC 1217404 (freier Volltext).
- Bernard Henrissat, Gideon Davies: Structural and sequence-based classification of glycoside hydrolases. In: Current Opinion in Structural Biology. Band 7, Nr. 5, Oktober 1997, S. 637–644, doi:10.1016/S0959-440X(97)80072-3, PMID 9345621.
- Lien Callewaert, Maarten Walmagh, Chris W. Michiels, Rob Lavigne: Food applications of bacterial cell wall hydrolases. In: Current Opinion in Biotechnology. Band 22, Nr. 2, April 2011, S. 164–171, doi:10.1016/j.copbio.2010.10.012, PMID 21093250.
- Lien Callewaert, Chris W. Michiels: Lysozymes in the animal kingdom. In: Journal of Biosciences. Band 35, Nr. 1, März 2010, S. 127–160, doi:10.1007/s12038-010-0015-5, PMID 20413917.
- I. Müller, N. Subert, H. Otto, R. Herbst, H. Rühling, M. Maniak, M. Leippe: A Dictyostelium Mutant with Reduced Lysozyme Levels Compensates by Increased Phagocytic Activity. In: Journal of Biological Chemistry. Band 280, Nr. 11, 11. Januar 2005, S. 10435–10443, doi:10.1074/jbc.M411445200, PMID 15640146.
- N. L. Thakur, S. Perovi -Ottstadt, R. Batel, M. Korzhev, B. Diehl-Seifert, I. M. Mueller, W. E. G. Mueller: Innate immune defense of the sponge Suberites domuncula against gram-positive bacteria: induction of lysozyme and AdaPTin. In: Marine Biology. Band 146, Nr. 2, Januar 2005, S. 271–282, doi:10.1007/s00227-004-1438-z.
- J. Fastrez: Phage lysozymes. In: EXS (Lysozymes: Model Enzymes in Biochemistry and Biology). Band 75, 1996, S. 35–64, PMID 8765293.
- J. J. Beintema und A. C. Terwisscha van Scheltinga: Plant lysozymes. In: EXS (Lysozymes: Model Enzymes in Biochemistry and Biology). Band 75, 1996, S. 75–86, PMID 8765295.
- J.-V. Höltje: Bacterial lysozymes. In: EXS (Lysozymes: Model Enzymes in Biochemistry and Biology). Band 75, 1996, S. 65–74, PMID 8765294.
- H. A. McKenzie: α-Lactalbumins and lysozymes. In: EXS (Lysozymes: Model Enzymes in Biochemistry and Biology). Band 75, 1996, S. 365–409, PMID 8765309.
- David M Irwin, Jason M Biegel, Caro-Beth Stewart: Evolution of the mammalian lysozyme gene family. In: BMC Evolutionary Biology. Band 11, Nr. 1, 2011, S. 166, doi:10.1186/1471-2148-11-166, PMID 21676251, PMC 3141428 (freier Volltext).
- NCBI; Dezember 2009
- P. Huang, W. S. Li, J. Xie, X. M. Yang, D. K. Jiang, S. Jiang, L. Yu: Characterization and expression of HLysG2, a basic goose-type lysozyme from the human eye and testis. In: Molecular immunology. Band 48, Nr. 4, Januar 2011, S. 524–531, doi:10.1016/j.molimm.2010.10.008, PMID 21093056.
- R. Kuroki, L. Weaver, B. Matthews: A covalent enzyme-substrate intermediate with saccharide distortion in a mutant T4 lysozyme. In: Science. Band 262, Nr. 5142, 24. Dezember 1993, S. 2030–2033, doi:10.1126/science.8266098, PMID 8266098.
- Walter A. Baase, Lijun Liu, Dale E. Tronrud, Brian W. Matthews: Lessons from the lysozyme of phage T4. In: Protein Science. Band 19, Nr. 4, April 2010, S. 631–641, doi:10.1002/pro.344.
- J.-V. Höltje: Lytic transglycosidases. In: EXS (Lysozymes: Model Enzymes in Biochemistry and Biology). Band 75, 1996, S. 425–429, PMID 8765311.
- Jane D Funkhouser, Nathan N Aronson: Chitinase family GH18: evolutionary insights from the genomic history of a diverse protein family. In: BMC Evolutionary Biology. Band 7, Nr. 1, 2007, S. 96, doi:10.1186/1471-2148-7-96, PMID 17594485, PMC 1945033 (freier Volltext).
- N. A. Udaya Prakash, M. Jayanthi, R. Sabarinathan, P. Kangueane, Lazar Mathew, K. Sekar: Evolution, Homology Conservation, and Identification of Unique Sequence Signatures in GH19 Family Chitinases. In: Journal of Molecular Evolution. Band 70, Nr. 5, Mai 2010, S. 466–478, doi:10.1007/s00239-010-9345-z, PMID 20480157.
- H. A. McKenzie, F.H. White (Jr.): Lysozyme and α-lactalbumin: structure, function and interrelationships. In: Advances in Protein Chemistry. Band 41, 1991, S. 173–315, doi:10.1016/S0065-3233(08)60198-9, PMID 2069076.
- J. P. van de Merwe, J. Lindemans, G. J. Mol: Plasma lysozyme levels and decay of neutrophilic granulocytes in patients with Crohn’s disease. In: Hepatogastroenterology. Band 27, Nr. 2, April 1980, S. 130–134, PMID 7216126.
- Yasuhiro Nonaka, Daisuke Akieda, Tomoyasu Aizawa, Nobuhisa Watanabe, Masakatsu Kamiya, Yasuhiro Kumaki, Mineyuki Mizuguchi, Takashi Kikukawa, Makoto Demura, Keiichi Kawano: X-ray crystallography and structural stability of digestive lysozyme from cow stomach. In: FEBS Journal. Band 276, Nr. 8, April 2009, S. 2192–2200, doi:10.1111/j.1742-4658.2009.06948.x, PMID 19348005.
- M. Xu, A. Arulandu, D. K. Struck, S. Swanson, J. C. Sacchettini, R. Young: Disulfide Isomerization After Membrane Release of Its SAR Domain Activates P1 Lysozyme. In: Science. Band 307, Nr. 5706, 7. Januar 2005, S. 113–117, doi:10.1126/science.1105143, PMID 15637279.
- A. Rau, T. Hogg, R. Marquardt, R. Hilgenfeld: A new lysozyme fold. Crystal structure of the muramidase from Streptomyces coelicolor at 1.65 A resolution. In: Journal of Biological Chemistry. Band 276, Nr. 34, August 2001, S. 31994–31999, doi:10.1074/jbc.M102591200, PMID 11427528.
- Erik J. van Asselt, Kor H. Kalk, Bauke W. Dijkstra: Crystallographic Studies of the Interactions of Lytic Transglycosylase Slt35 with Peptidoglycan. In: Biochemistry. Band 39, Nr. 8, Februar 2000, S. 1924–1934, doi:10.1021/bi992161p.
- Maike Bublitz, Lilia Polle, Christin Holland, Dirk W. Heinz, Manfred Nimtz, Wolf-Dieter Schubert: Structural basis for autoinhibition and activation of Auto, a virulence-associated peptidoglycan hydrolase of Listeria monocytogenes. In: Molecular Microbiology. Band 71, Nr. 6, März 2009, S. 1509–1522, doi:10.1111/j.1365-2958.2009.06619.x, PMID 19210622.
- Severine Layec, Bernard Decaris, Nathalie Leblond-Bourget: Diversity of Firmicutes peptidoglycan hydrolases and specificities of those involved in daughter cell separation. In: Research in Microbiology. Band 159, Nr. 7-8, September 2008, S. 507–515, doi:10.1016/j.resmic.2008.06.008, PMID 18656532.
- Edie Scheurwater, Chris W. Reid, Anthony J. Clarke: Lytic transglycosylases: Bacterial space-making autolysins. In: The International Journal of Biochemistry & Cell Biology. Band 40, Nr. 4, Januar 2008, S. 586–591, doi:10.1016/j.biocel.2007.03.018.
- J.-V. Höltje: Lysozyme substrates. In: EXS (Lysozymes: Model Enzymes in Biochemistry and Biology). Band 75, 1996, S. 105–110, PMID 8765297.
- Structural Classification of Proteins: Fold: Lysozyme-like. (Memento vom 17. Mai 2011 im Internet Archive) Abgerufen am 12. Juli 2011.
- T. Goto, Y. Abe, Y. Kakuta, K. Takeshita, T. Imoto, T. Ueda: Crystal structure of Tapes japonica Lysozyme with substrate analogue: structural basis of the catalytic mechanism and manifestation of its chitinase activity accompanied by quaternary structural change. In: Journal of Biological Chemistry. Band 282, Nr. 37, 10. Juli 2007, S. 27459–27467, doi:10.1074/jbc.M704555200, PMID 17631496.
- David L. Ollis, Eong Cheah, Miroslaw Cygler, Bauke Dijkstra, Felix Frolow, Sybille M. Franken, Michal Harel, S. Jamse Remington, Israel Silman, Joseph Schrag, Joel L. Sussman, Koen H.G. Verschueren, Adrian Goldman: The α/β hydrolase fold. In: Protein Engineering, Design and Selection (PEDS). Band 5, Nr. 3, 1992, S. 197–211, doi:10.1093/protein/5.3.197, PMID 1409539.
- Structural Classification of Proteins: Superfamily: (Trans)glycosidases. (Memento vom 16. Mai 2011 im Internet Archive) Abgerufen am 12. Juli 2011.
- Ronny Helland, Renate L. Larsen, Solrun Finstad, Peter Kyomuhendo, Atle N. Larsen: Crystal structures of g-type lysozyme from Atlantic cod shed new light on substrate binding and the catalytic mechanism. In: Cellular and Molecular Life Sciences. Band 66, Nr. 15, August 2009, S. 2585–2598, doi:10.1007/s00018-009-0063-x, PMID 19543850.
- R. Kuroki, L. H. Weaver, B. W. Matthews: Structural basis of the conversion of T4 lysozyme into a transglycosidase by reengineering the active site. In: Proceedings of the National Academy of Sciences. Band 96, Nr. 16, 3. August 1999, S. 8949–8954, doi:10.1073/pnas.96.16.8949, PMID 10430876, PMC 17713 (freier Volltext).
- A. Fokine, K. A. Miroshnikov, M. M. Shneider, V. V. Mesyanzhinov, M. G. Rossmann: Structure of the Bacteriophage φKZ Lytic Transglycosylase gp144. In: Journal of Biological Chemistry. Band 283, Nr. 11, 14. Januar 2008, S. 7242–7250, doi:10.1074/jbc.M709398200, PMID 18160394.
- Bin Yang, Jianwu Wang, B. o. Tang, Yufang Liu, Chengdong Guo, Penghua Yang, Tian Yu, Rong Li, Jianmin Zhao, Lei Zhang, Yunping Dai, Ning Li, Vladimir Uversky: Characterization of Bioactive Recombinant Human Lysozyme Expressed in Milk of Cloned Transgenic Cattle. In: PLoS ONE. Band 6, Nr. 3, 16. März 2011, S. e17593, doi:10.1371/journal.pone.0017593.
- P. Kuhnert, B. Muermann, U.-J. Salzer (Hrsg.): Handbuch Lebensmittelzusatzstoffe. Band 3 (Loseblattsammlung), Behr’s Verlag
- R. Lodi, V. Mazzanti: Effetto del lisozima sulle microflore casearie ed anticasearie. In: Latte. 10(6), 1985, S. 549.
- Verordnung (EU) Nr. 471/2012 der Kommission vom 4. Juni 2012 zur Änderung von Anhang II der Verordnung (EG) Nr. 1333/2008 des Europäischen Parlaments und des Rates hinsichtlich der Verwendung von Lysozym (E 1105) in Bier In: EUR Der Zugang zum EU-Recht. EUR-Lex - 32012R0471
- Jia Tong, HengXi Wei, XiaoFang Liu, WenPing Hu, MingJun Bi, YuanYuan Wang, QiuYan Li, Ning Li: Production of recombinant human lysozyme in the milk of transgenic pigs. In: Transgenic Research. Band 20, Nr. 2, April 2011, S. 417–419, doi:10.1007/s11248-010-9409-2.
- H. Nakajima, T. Muranaka, F. Ishige, K. Akutsu, K. Oeda: Fungal and bacterial disease resistance in transgenic plants expressing human lysozyme. Hrsg.: Plant Cell Reports. 10. Auflage. Nr. 16. Springer-Verlag, Juli 1994, S. 674–679.
- M. Revenis, M. Kaliner: Lactoferrin and lysozyme deficiency in airway secretions: Association with the development of bronchopulmonary dysplasia. In: The Journal of Pediatrics. Band 121, Nr. 2, August 1992, S. 262–270, doi:10.1016/S0022-3476(05)81201-6, PMID 1640295.
- Bo Lönnerdal: Nutritional and physiologic significance of human milk proteins. In: The American journal of clinical nutrition. Band 77, Nr. 6, Juni 2003, S. 1537S–1543S, PMID 12812151 (ajcn.org).
- P. E. Perillie, K. Khan, S. C. Finch: Serum lysozyme in pulmonary tuberculosis. In: American Journal of Medical Science. Band 265, Nr. 4, April 1973, S. 297–302, PMID 4196282 (lww.com).
- Elli Koivunen, Carola Groenhagen-Riska, Matti Klockars, Olof Selroos: Blood Monocytes and Serum and Bone Marrow Lysozyme in Sarcoidosis. In: Acta Medica Scandinavica. Band 210, Nr. 1-6, 12. Januar 1981, S. 107–110, doi:10.1111/j.0954-6820.1981.tb09784.x.
- Bärbel Porstmann, Kurt Jung, Helmut Schmechta, Ursula Evers, Monika Pergande, Tomas Porstmann, Hans-Joachim Kramm, Heinz Krause: Measurement of lysozyme in human body fluids: Comparison of various enzyme immunoassay techniques and their diagnostic application. In: Clinical Biochemistry. Band 22, Nr. 5, Oktober 1989, S. 349–355, doi:10.1016/S0009-9120(89)80031-1.
- Michael H. Grieco, Mohan M. Reddy, Harish B. Kothari, Michael Lange, Elena Buimovici-Klein, Daniel William: Elevated β2-microglobulin and lysozyme levels in patients with acquired immune deficiency syndrome. In: Clinical Immunology and Immunopathology. Band 32, Nr. 2, August 1984, S. 174–184, doi:10.1016/0090-1229(84)90119-3.
- I. Torsteinsdóttir, L. Håkansson, R. Hällgren, B. Gudbjörnsson, N.-G. Arvidson, P. Venge: Serum lysozyme: a potential marker of monocyte/macrophage activity in rheumatoid arthritis. In: Rheumatology. Band 38, Nr. 12, 1999, S. 1249–1254, doi:10.1093/rheumatology/38.12.1249.
- Per Venge, Tony Foucard, Jörn Henriksen, Lena Håkansson, Anders Kreuger: Serum-levels of lactoferrin, lysozyme and myeloperoxidase in normal, infection-prone and leukemic children. In: Clinica Chimica Acta. Band 136, Nr. 2-3, 31. Januar 1984, S. 121–130, doi:10.1016/0009-8981(84)90283-3.
- J. W. Jenzano, S. L. Hogan, R. L. Lundblad: Factors influencing measurement of human salivary lysozyme in lysoplate and turbidimetric assays. In: Journal of clinical microbiology. Band 24, Nr. 6, Dezember 1986, S. 963–967, PMID 3782460, PMC 269079 (freier Volltext).
- Johan Brouwer, Trudi van Leeuwen-Herberts, Marjo Otting-van de Ruit: Determination of lysozyme in serum, urine, cerebrospinal fluid and feces by enzyme immunoassay. In: Clinica Chimica Acta. Band 142, Nr. 1, 15. September 1984, S. 21–30, doi:10.1016/0009-8981(84)90097-4.
- G. Sava: Pharmacological aspects and therapeutic applications of lysozyme. In: EXS (Lysozymes: Model Enzymes in Biochemistry and Biology). Band 75, 1996, S. 434–449, PMID 8765312.
- fehlender Einzelnachweis
- Gutachten des Wissenschaftlichen Gremiums für diätetische Produkte, Ernährung und Allergien (NDA) auf Ersuchen der Kommission bezüglich einer Mitteilung durch AMAFE betreffend Ei-Lysozym als Lebensmittelzusatzstoff gemäß Artikel 6 Absatz 11 der Richtlinie 2000/13/EG. (PDF) In: The EFSA Journal. 186, 2005, S. 1–5.