Sekundärstruktur
Als Sekundärstrukturen werden in der Biochemie regelmäßige lokale Strukturelemente von Makromolekülen bezeichnet. Der Fokus liegt dabei auf dem Polymerrückgrat, auch Backbone genannt. Die Konformation der Seitenketten und ihr Verhältnis zu anderen Elementen werden außer Acht gelassen.[1]
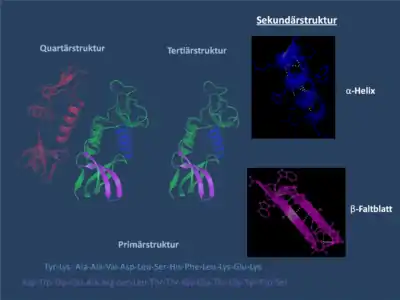
Die Darstellung der Sekundärstruktur eines Polymers bietet einen besseren Überblick als die Darstellung seiner vollständigen Molekülstruktur und gibt zugleich einen wesentlich genaueren Einblick in die tatsächliche Struktur als die Darstellung in der Pauling-Schreibweise oder der Fischer-Projektion.
Eine Sekundärstruktur geht aus der jeweiligen Primärstruktur hervor. Die topologische Anordnung der Atome im Raum wird durch die Bildung von Wasserstoffbrücken zwischen den Atomen festgelegt.[2] Auch die Form der Polymerisierung kann, etwa bei Polysacchariden, Einfluss auf die Sekundärstruktur nehmen.
Übergeordnete Strukturebenen sind die Tertiärstruktur und die Quartärstruktur, mit denen die Konformation des gesamten Makromoleküls beziehungsweise dessen Assoziation in einem makromolekularen Komplex beschrieben wird. Die Unterscheidung hierarchisch geordneter Strukturebenen und ihre Einteilung in Primärstruktur, Sekundärstruktur, Tertiärstruktur und Quartärstruktur wurde 1952 von Kaj Ulrik Linderstrøm-Lang vorgeschlagen.
Sekundärstruktur von Proteinen
Grundsätzlich ergeben sich für die Polypeptidkette eines Proteins unüberschaubar viele Konformationsmöglichkeiten. Der Vergleich von Proteinfaltungen zeigt jedoch, dass sich einige Motive immer wieder finden. Solche lokalen Abschnitte eines Proteins mit einer bestimmten dreidimensionalen Form werden als Sekundärstrukturelemente bezeichnet.
Die räumliche Anordnung des Peptidrückgrats wird wesentlich durch Wasserstoffbrückenbindungen bestimmt, die sich intramolekular zwischen verschiedenen Atomen am Backbone ausbilden. Dabei kann eine Brücke vom Wasserstoff-Atom einer Aminohydrogen-Gruppe (N-H) ausgehend zum Sauerstoff-Atom einer Carbonyl-Gruppe (C=O) in unterschiedlicher Entfernung geschlagen werden. Das jeweils wiederkehrende Muster seiner Wasserstoffbrücken definiert ein Sekundärstrukturelement. Die hierdurch in bestimmten Abständen zueinander festgelegten Atome lassen sich auf verschiedene Weise kennzeichnen, beispielsweise als Helix nach Anzahl von Atomen pro Drehung oder durch Angabe typischer Torsionswinkel in der Peptidkette. Die am häufigsten gefundenen Typen von Sekundärstrukturelementen sind α-Helices und β-Faltblätter.
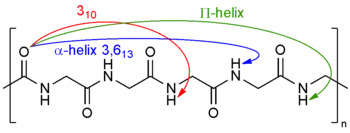
* 310-Helix (oben)
* α-Helix
* π-Helix (unten)
Hinsichtlich der Sekundärstruktur werden unterschieden:
- α-Helix
- π-Helix
- 310-Helix
- linksgängige α-Kette der Kollagene
- β-Faltblatt
- β-Schleife
- β-Helix
- Random Coil: Bereiche, die keine definierte Sekundärstruktur aufweisen
Mit Ausnahme der Beta-Schleifen und Random coils zeichnen sich diese Bereiche dadurch aus, dass in ihnen die beiden einzig möglichen Drehwinkel ψ und φ des Peptidrückgrates festgelegt sind und sich über die Länge des Sekundärstrukturelementes hindurch periodisch wiederholen. Im Ramachandran-Plot sind die möglichen Sekundärstrukturen als Funktion der zugehörigen ψ/φ Winkelpaare dargestellt, im Janin-Plot die Diederwinkel der Aminosäureseitenketten (χ1 und χ2). Durch die Wasserstoffbrücken innerhalb des Peptidrückgrates werden die Sekundärstrukturelemente energetisch stabilisiert. Je nach Art des Sekundärstrukturelements können bestimmte Aminosäureseitenketten auf dessen Struktur destabilisierend wirken.
Daneben resultiert eine weitere Wechselwirkung durch das π*-Orbital des C=O Kohlenstoffes mit einem der beiden Lonepairs des darauffolgenden C=O Sauerstoffes. Dies führt zu einer Pyramidalisierung der sonst planaren Amide.
Die Sekundärstruktur eines Proteins besteht aus verschiedenen Sekundärstrukturelementen. Die nächsthöhere Einteilungsstufe sind Strukturmotive, darüber hinaus Proteindomänen. Die vollständige Proteinstruktur (d. h. die Abfolge bzw. Anordnung der Sekundärstrukturelemente) wird als Tertiärstruktur bezeichnet. Sie ist für jedes Protein charakteristisch und für die biologische Funktion unbedingt notwendig. Während bzw. nach der Translation eines RNA-Moleküls werden die Sekundärstruktur und die höheren Strukturebenen des Proteins ausgebildet. Dieser Proteinfaltung genannte Vorgang wird bei den meisten Proteinen u. a. durch Chaperone unterstützt, geschieht bei kleinen Proteinen jedoch teilweise auch spontan.[3]
Sekundärstruktur von Nukleinsäuren
Auch Nukleinsäuren, also DNA und RNA, können Sekundärstrukturen bilden. Voraussetzung hierfür ist, dass das Nukleinsäuremolekül zunächst als Einzelstrang vorliegt. Komplementäre Abschnitte des Strangs können dann Wasserstoffbrücken ausbilden, was zur Bildung von intramolekularen Basenpaarungen mit doppelsträngiger Struktur führt. Nicht komplementäre Abschnitte bleiben einzelsträngig.
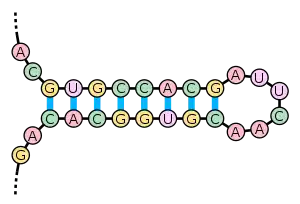
Die gepaarten Abschnitte des doppelsträngig vorliegenden Bereichs, Stamm (stem) genannt, können einen längeren einzelsträngigen Abschnitt dazwischen als Schleife (loop) einschließen und so eine sekundäre Stamm-Schleife-Struktur (stem-loop) bilden. Sekundärstrukturen von Nukleinsäuren mit kleiner Schleife, also kurzem einzelsträngigen Zwischenabschnitt, bezeichnet man in Anlehnung an ihre Form auch als Haarnadelstruktur (hairpin). Die gegenläufige Paarung von komplementären Basensequenzen wird in besonderer Weise durch palindromische Sequenzen in der Nukleotidabfolge eines Nukleinsäurestrangs ermöglicht. Durch Paarung sich umgekehrt wiederholender Basenfolgen an den beiden Enden eines Nukeinsäurestranges entsteht eine Pfannenstielstruktur (panhandle).
Die Sekundärstrukturen haben wichtige Funktionen bei der Regulation der Transkription. Sie können als Primer dienen (siehe Telomerase) oder Voraussetzung für die enzymatische Aktivität der Ribosomen sein (siehe rRNA).
Algorithmen zur RNA-Strukturvorhersage
Einzelnachweise
- Eintrag zu secondary structure. In: IUPAC (Hrsg.): Compendium of Chemical Terminology. The “Gold Book”. doi:10.1351/goldbook.S05530.
- Hermann J. Roth, Christa E. Müller, Gerd Folkers: Stereochemie und Arzneistoffe, Wissenschaftliche Verlagsgesellschaft Stuttgart, 1998, S. 240–244, ISBN 3-8047-1485-4.
- Horton, Robert et al.: Biochemie, 4. Auflage, Pearson Studium, München (2008), S. 152, ISBN 978-3-8273-7312-0.