Escherichia-Virus T7
Der Bakteriophage T7 (oder der T7-Phage, offiziell Escherichia virus T7) ist eine Virus-Spezies der Gattung Teseptimavirus (früher T7virus), die anfällige Bakterienzellen infiziert. T7-Phagen besitzen ein doppelsträngiges DNA-Genom und infizieren die meisten Stämme (englisch: strains) der Colibakterien (Escherichia coli). Der Bakteriophage T7 hat einen lytischen Vermehrungszyklus und eine Reihe von Eigenschaften, die ihn zu einem idealen Phagen für Experimente machen.[2][3]
| Escherichia-Virus T7 | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|

Escherichia-Virus T7 | ||||||||||||||||||||
| Systematik | ||||||||||||||||||||
| ||||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||||
| ||||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||||
| Escherichia virus T7 | ||||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||||
| T7 | ||||||||||||||||||||
| Links | ||||||||||||||||||||
|
Entdeckung
In einer Studie von Milislav Demerec und Ugo Fano aus dem Jahr 1945 war T7 als einer von sieben Phagentypen (T1 bis T7) beschrieben worden, die Kolibacterien infizieren.[4][5] Wahrscheinlich ist es identisch mit dem zuvor von Max Delbrück verwendeten Phagen δ. Zudem hat wahrscheinlich Félix H. d’Hérelle in den 1920er Jahren bereits einen engen Verwandten von T7 untersucht.[6][5]
Wirte
T7 wächst auf ‚rauen‘ Stämmen von Kolibakterien (d. h. solchen ohne O-Antigen Polysaccharid voller Länge auf ihrer Oberfläche) und einigen anderen Darmbakterien, aber nahe Verwandte infizieren auch glatte und sogar gekapselte Stämme.[7]
Aufbau
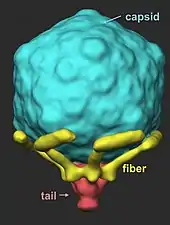

T7-Viruspartikel (Virionen) haben eine komplexe strukturelle Symmetrie. Das Kapsid des Phagen hat einen ikosaedrischen Kopf mit einem Innendurchmesser von 55 nm, daran haftet einem Schwanz von 19 nm Durchmesser und Länge von 28,5 nm.
Genom
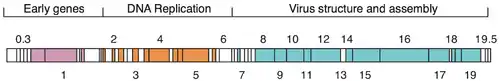
Das Genom des Phagen T7 gehört zu den ersten vollständig sequenzierten Genomen (1983 veröffentlicht).[8][9] Der Kopf des Viruspartikels enthält das etwa 40 kbp große doppelsträngige DNA-Genom, das für 55 Proteine kodiert.[10]
Vermehrungszyklus
T7 hat einen kurzen Replikationszyklus von 17 Minuten (bei 37 °C), das ist die Zeit von der Infektion bis zur Lyse (Auflösung) der Wirtszelle, wenn die neuen Phagen-Virionen freigesetzt werden. Aufgrund der kurzen Latenzzeit werden die meisten physiologischen Studien bei geringen 30 °C durchgeführt, wobei infizierte Zellen sich dann nach 30 Minuten auflösen. Es wurden jedoch hohe Fitnessstämme von T7 mit einer Latenzzeit von nur ca. 11 min bei 37 °C isoliert (unter optimalen Bedingungen in angereicherten Nährmedien).[12]
Infektion der Wirtszellen
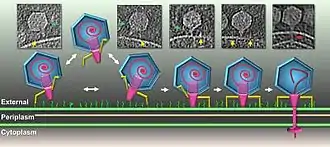
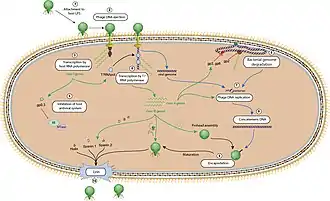
Der T7-Phage erkennt bestimmte Rezeptoren auf der Oberfläche der Colibakterien und haftet an der Zelloberfläche vermöge seiner Schwanzfasern; bei einigen T7-Stämmen sind die Schwanzfasern jedoch durch Schwanzspitzen (‚Spikes‘) mit enzymatischer Aktivität ersetzt, die die O- bzw. K-Antigene auf der Zelloberfläche abbauen. Beim Adsorbtions- und Penetrationsprozess werden Lysozyme verwendet, um eine Öffnung in der Peptidoglycanschicht der Bakterienzellwand zu schaffen, was dann den Transfer der viralen DNA in das Bakterium ermöglicht.
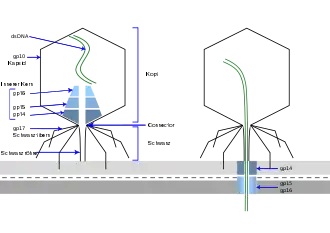
Wegen des kurzen, verkümmerten Schwanzes der T7-ähnlichen Phagen müssen zuerst Proteine des Virions einen Kanal von der Schwanzspitze aus in das Zellzytoplasma bilden, damit das Phagengenom zu Beginn der Infektion in die Zelle injiziert werden kann.[13] Der Phage injiziert dabei auch Proteine, die benötigt werden, um die Replikation des viralen Genoms zu starten und das Wirtsgenom zu zerstückeln.[14] Der T7-Bakteriophage überwindet verschiedene Verteidigungsstrategien der Wirtsbakterien, neben der Peptidoglycan-Zellwand insbesondere auch das CRISPR-System.[14] Sobald der T7-Phage sein virales Genom in das Bakterium eingeschleust hat, wird der DNA-Replikationsprozess des Wirtsgenoms gestoppt und die Replikation des viralen Genoms beginnt.
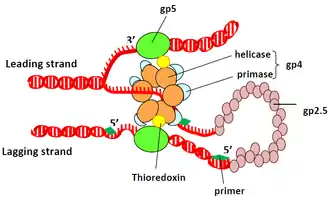
Die Helikase gp4 wickelt die doppelsträngige DNA in zwei einzelsträngige DNA-Matrizen auf (siehe Abbildung „Replikation des Phagen T7“). Eine Primase fügt einen Oligoribonukleotid-Primer hinzu. Das Protein gp5/Thioredoxin katalysiert dann die Synthese von Vorderstrang (en. leadingstrand) und Hinterstrang (en. lagging strand). Dabei umhüllt gp2.5 die während der Replikation entstehende einzelsträngige DNA.
Unter optimalen Bedingungen kann der T7-Phage den gesamten Prozess innerhalb von 25 Minuten mit der Auflösung (Lyse) des Bakteriums (d. h. dem Tod der E. coli-Wirtszelle) abschließen, wobei das Virus über 100 Nachkommen produzieren kann.[14]
Anwendungen in der Molekularbiologie
Die T7-Promotorsequenz wird aufgrund ihrer extrem hohen Affinität für T7-RNA-Polymerase und des damit verbundenen hohen Expressionsniveaus in der Molekularbiologie vielfach verwendet.
T7 wurde als Modellorganismus in der synthetischen Biologie verwendet. Chan et al. haben 2005 das Genom von T7 ‚refaktorisiert‘ und ersetzten dabei ungefähr 12 kbp seines Genoms durch synthetisierte DNA.[15] Das Design dieser konstruierten DNA wurde so ausgelegt, dass diese in verschiedener Hinsicht einfacher zu handhaben ist: Einzelne funktionelle Elemente wurden zwecks vereinfachter Modifikation (durch eine Restriktionsendonuklease) voneinander getrennt. Insbesondere wurden überlappende Protein-kodierende Domänen (englisch coding sequences, d. h. ‚Gene‘ im eigentlichen Sinne) getrennt. Es wurde auch untersucht, ob T7 oder Teile davon, ggf. auch zusammen mit anderen Komponenten, zur Behandlung menschlicher Tumorzellen geeignet sein könnte.[16]
Siehe auch
Einzelnachweise
- ICTV: ICTV Taxonomy history: Enterobacteria phage T4, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- T7virus: Enterobacteria phage T7, auf: ViralZone, Swiss Institute of Bioinformatics (SIB)
- Master Species List 2018a v1, auf: ICTV online
- Milislav Demerec, Ugo Fano: Bacteriophage-Resistant Mutants in Escherichia Coli. In: Genetics. 30, Nr. 2, März 1945, S. 119–136. PMID 17247150. PMC 1209279 (freier Volltext).
- Lobocka M, Szybalski (Hrsg.): Bacteriophages. Academic Press, 31. Dezember 2012, ISBN 978-0-12-394788-8, S. 226 ff.
- Félix d’Hérelle: The Bacteriophage and Its Behavior. Baltimore, MD: Williams & Wilkins, 1926
- I. J. Molineux: Chapter 20: The T7 group. In: The Bacteriophages (R. Calendar, Hrsg.), S. 277 ff, Oxford University Press, Oxford, 2006
- Genome of bacteriophage T7.
- J. J. Dunn, F. W. Studier: Complete nucleotide sequence of bacteriophage T7 DNA and the locations of T7 genetic elements. In: Journal of Molecular Biology. 166, Nr. 4, 1983, S. 477–535. doi:10.1016/S0022-2836(83)80282-4. PMID 6864790.
- Uniprot: reference proteome of bacteriophage T7.
- R. Häuser, S. Blasche, T. Dokland, E. Haggård-Ljungquist, A. von Brunn, M. Salas, S. Casjens, I. Molineux, P. Uetz: Bacteriophage protein-protein interactions (= Advances in Virus Research. Vol. 83). 2012, ISBN 978-0-12-394438-2, Bacteriophage Protein–Protein Interactions, S. 219–298, doi:10.1016/B978-0-12-394438-2.00006-2, PMID 22748812, PMC 3461333 (freier Volltext).
- R. H. Heineman, J. J. Bull: Testing Optimality with Experimental Evolution: Lysis Time in a Bacteriophage. In: Evolution. 61, Nr. 7, 2007, S. 1695–1709. doi:10.1111/j.1558-5646.2007.00132.x. PMID 17598749. PMC 1974807 (freier Volltext).
- C. Y. Chang, P. Kemp, I. J. Molineux: Gp15 and gp16 cooperate in translocating bacteriophage T7 DNA into the infected cell. In: Virology. 398, Nr. 2, 2010, S. 176–186. doi:10.1016/j.virol.2009.12.002. PMID 20036409. PMC 2825023 (freier Volltext).
- New Details about Bacteriophage T7-Host Interactions. Archiviert vom Original am 17. August 2011.
- Chan LY, Kosuri S, Endy D: Refactoring bacteriophage T7. In: Molecular Systems Biology. 1, 2005, S. 2005.0018. doi:10.1038/msb4100025. PMID 16729053. PMC 1681472 (freier Volltext).
- Chen X, Li Y, Xiong K, Aizicovici S, Xie Y, Zhu Q, Sturtz F, Shulok J, Snodgrass R, Wagner TE, Platika D: Cancer gene therapy by direct tumor injections of a nonviral T7 vector encoding a thymidine kinase gene. In: Human Gene Therapy. 9, Nr. 5, März 1998, S. 729–736. doi:10.1089/hum.1998.9.5-729. PMID 9551620.
Weblinks
- T7Virus, auf: ViralZone, Swiss Institute of Bioinformatics (SIB)
- New Details about T7-host interactions. Microbe Magazine
- T7 reference proteome in Uniprot
- Skoltech research shows how a 'Swiss Army knife' protein helps phages disarm their victims, auf: EurekAlert! vom 27. April 2020