Disulfidbrücke
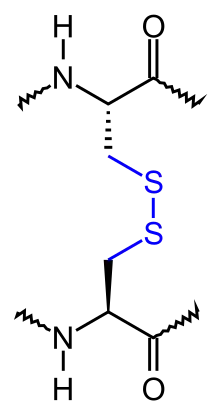
Eine Disulfidbrücke, Disulfidbindung oder Disulfidbrückenbindung bezeichnet in der Chemie eine kovalente Bindung zwischen zwei Schwefelatomen, deren jeweils einzige freie Valenz mit einem Organylrest abgesättigt ist. In der Biochemie ist die Disulfidbindung die kovalente Bindung (eine Atombindung) zwischen den Schwefel-Atomen zweier Cystein-Moleküle, die in der Aminosäureseitenkette eines Proteins vorkommen.
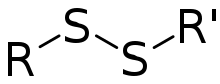
_V.1.svg.png.webp)
_V3.svg.png.webp)
Zwei mittels Disulfidbindung verknüpfte Cystein-Reste in Proteinen bezeichnet man dabei auch als Cystin-Brücke.
Funktion
Disulfidbrücken formen und stabilisieren die dreidimensionale Proteinstruktur (Tertiärstruktur) durch die Bildung von Schlaufen innerhalb der Aminosäureketten oder verknüpfen mehrere Aminosäureketten zu einem funktionstüchtigen Protein. Disulfidbrücken sind als kovalente Bindungen dabei deutlich fester als beispielsweise die auch im Molekül vorkommenden Wasserstoffbrückenbindungen, die zu den Nebenvalenzbindungen zählen.
Die Bildung von Disulfidbrücken kann ein separater Schritt bei der Faltung eines Proteins sein, ohne dass nachfolgende Faltungsschritte hinausgezögert werden.[2]
Beispiele
Disulfidbindungen sind typisch für sekretorische Proteine, da sie sich im Cytosol nicht ausbilden können.
Insulin
So besteht das Proteohormon Insulin aus zwei verschiedenen Aminosäureketten, die als A- und B-Kette bezeichnet werden, wobei die A-Kette intrachenar durch eine und interchenar mit der B-Kette durch zwei Disulfid-Bindungen verbunden ist.
Lipase
Eine Lipase, ein fettspaltendes Enzym aus der Bauchspeicheldrüse (Pankreas) des Schweins, besitzt sieben Disulfidbrücken.
GPCRs
G-Protein-gekoppelte Rezeptoren sind membranständige Proteine, die durch eine Disulfidbrücke zwischen der 3. transmembranären Helix und der zweiten extrazellulären Schleife stabilisiert werden. Mutationsstudien belegen, dass der Rezeptor nicht mehr exprimiert wird, wenn eines der beteiligten Cysteine zu Serin mutiert wird.
Bindungsbildung
Reaktion
Die bei der Ausbildung einer Disulfidbrückenbindung beteiligten funktionellen Gruppen nennt man Thiolgruppen (Mercaptogruppen). Vereinfacht lässt sich die Bildung einer solchen S-S-Bindung als Oxidation (Abgabe von Wasserstoff bzw. Elektronen) verstehen:
Oxidation: R-SH + HS-R' → R-S-S-R' + 2 H+ + 2e−
Reduktion: 2 Fe3+ + 2 e− → 2 Fe2+
R und R' bezeichnen dabei in der Biochemie die Cysteine am Peptid/Protein. Die beiden überschüssigen Wasserstoff-Atome werden von einem Wasserstoff-Akzeptor gebunden (die Schreibweise [H] verdeutlicht, dass sie nicht als Wasserstoffgas freigesetzt werden). Sie können letztendlich beispielsweise auf Sauerstoff übertragen werden.
- 4 [H] + O2 → 2 H2O
Zeitpunkt
Disulfidbrücken werden noch während der Translation in die Proteine eingefügt, wenn sich Teile von diesen im Rahmen ihrer Synthese schon im (beim Eukaryoten) Endoplasmatischen Retikulum (ER) befinden, oder danach, wenn sie sich komplett im ER oder einem anderen membranumhüllten Zellorganell aufhalten, was dann eine posttranslationale Modifikation darstellt. Bei Prokaryoten gilt dies analog für die Translation ins Periplasma.
Da die Bildung einer Disulfidbrücke aus zwei Mercaptogruppen eine Oxidation ist, kann die Reaktion nur in einem oxidativen Milieu erfolgen. Das Cytoplasma ist eine reduzierende Umgebung, daher enthalten cytoplasmatische Proteine in der Regel keine Disulfidbrücken.
Enzyme
Die Ausbildung von Disulfidbindungen ist kein spontaner Prozess; sie ist eine Redox-Reaktion, die einen entsprechenden Reaktionspartner zum Elektronenübertrag erfordert. Die Ausbildung erfolgt enzymkatalysiert. Verfügt ein Protein darüber hinaus über mehr als nur zwei Cysteine, so besteht die Möglichkeit, dass sich durch Verknüpfung der „falschen“ Cysteine Disulfidbrücken ergeben, die nicht dem nativen Zustand des Proteins entsprechen. Es muss eine Umknüpfung falscher Disulfidbrücken (engl. reshuffling) erfolgen.
Proteindisulfidisomerasen
Eukaryoten besitzen im Endoplasmatischen Retikulum (ER) Proteindisulfidisomerasen (PDI). Die fortschreitende Faltung führt dabei zusammengehörende Cysteine langsam in räumliche Nähe, was korrekte Verknüpfungen zunehmend wahrscheinlicher macht.
Thio-Disulfid-Oxidoreduktasen
Das prokaryotische Gegenstück zu den Proteindisulfidisomerasen ist das periplasmatische und innere-Membran-ständige Dsb-System (dsb von di-sulfide-bond), das Disulfid-Ausbildung und -Isomerisierung kontrolliert.
GSH/GSSG-System
Glutathion (GSH) ist ein Isopeptid, das im Cytoplasma von sowohl Prokaryoten als auch Eukaryoten vorhanden ist und an der Ausbildung von Disulfidbrücken teilnimmt. Es reagiert in einer Disulfidaustauschreaktion:
R und R' sind wiederum die Cysteine im Proteinrückgrat, GSSG ist das GSH-Dimer mit Disulfidbrücke (ausgedrückt durch die nebeneinander geschriebenen Schwefelatome „SS“).
- R-SH + GSSG → R-S-S-G + GSH
Das linke der beiden Produkte bezeichnet man als gemischtes Disulfid. Es wird weiter umgesetzt:
- R-S-S-G + HS-R' → R-S-S-R' + GSH
Im Cytosol wird es (enzymatisch) in reduzierter Form gehalten (GSH). Man spricht von „reduzierenden Bedingungen“.
Diese Bedingungen können durch die relativen Konzentrationsverhältnisse von GSH und dementsprechend disulfidverbrückten Dimer GSSG veranschaulicht werden:
| Zellkompartiment | GSH | GSSG |
|---|---|---|
| Cytosol | 60 | 1 |
| ER | 1 | 2 |
Die Verhältnisse im ER entsprechen dem extrazellulären Milieu in Anwesenheit von Sauerstoff (das Lumen des ER ist topologisch äquivalent zum Außenraum).
GSH spielt ebenfalls eine Rolle beim oxidativen Stress.
Bedeutung für die rekombinante Proteinexpression
Disulfidbrücken in Proteinen limitieren deren rekombinante Expressionsfähigkeit, d. h. deren biotechnologische Herstellung.
In Eukaryoten werden Disulfidbrücken im Endoplasmatischen Retikulum ausgebildet. Expressionssysteme sind aber häufig Prokaryoten, die kein ER besitzen. Wird das Protein ins Cytosol translatiert, können keine Disulfidbrücken entstehen (siehe GSH/GSSG-System).
Bildung von Einschlusskörpern
Ohne Disulfidbrücken ist die Faltung des Proteins gestört. Neben dem proteolytischen Abbau kann es insbesondere bei der aus Gründen der Ausbeute gewünschten, übermäßigen Produktion (Überexpression) des Proteins zur Bildung von Einschlusskörpern kommen (Proteinaggregation zum sog. inclusion body). Dabei ist der Einschlusskörper in seinem Inneren vor Reduktion geschützt und bildet deshalb willkürlich Disulfidbrücken zu anderen Proteinen aus. Sowohl Fehlfaltung als auch die Bildung von Einschlusskörpern machen weitere Arbeitsschritte bei der Aufreinigung des Proteins erforderlich und liefern zum Teil nur in begrenztem Umfang funktionelles Protein.
Resolubilisierung mit Reduktionsmitteln

DTT (Dithiothreitol), Mercaptoethanol, Tris(2-carboxyethyl)phosphin und DTE (Dithioerythritol) sind Reduktionsmittel für Disulfidbindungen. Sie werden in der Biochemie verwendet, um in Einschlusskörpern die willkürlich ausgebildeten Disulfidbindungen wieder aufzulösen.
Dies geschieht durch Reduktion derselben, wie im Bild gezeigt. Die einzelnen Proteine werden dadurch voneinander getrennt und lösen sich wieder. Man spricht von der Resolubilisierung (engl. soluble = löslich) der Proteine.
Reoxidation mit Glutathion
Die Proteine aus diesem Prozess enthalten lediglich reduzierte Disulfidbindungen. Um funktionelles Protein zu erhalten, muss das Protein korrekt gefaltet sein. Dazu müssen die Disulfidbindungen wieder ausgebildet werden und dies in einer kontrollierten Weise, sodass nur die „gewünschten“ Cystein-Paare eine Bindung miteinander eingehen.
Um das zu erreichen, werden die Proteine mit Glutathion (GSH) versetzt. Es erfolgt eine Reoxidation der Disulfidbindungen in den nativen Zustand (= „Zurück-Oxidation“ zu dem von der Natur vorgesehenen Bindungszustand dieses Proteins). Bei diesem Prozess müssen diverse Bedingungen angepasst und eingehalten werden, um eine erneute Proteinaggregation (siehe Bildung von Einschlusskörpern) zu vermeiden. Dazu werden die Protein- und GSH-Konzentrationen, pH-Wert der Lösung, Temperatur und Reaktionszeiten variiert und optimiert.
Darüber hinaus besteht die Möglichkeit, Faltungs-Additive wie Arginin zuzugeben, die die korrekte Disulfidbindungsausbildung ebenfalls unterstützen. Bei der sog. Pulsrenaturierung wird nicht gleich zu Beginn der Renaturierung das gesamte Protein in die Renaturierungs-Lösung gegeben. Stattdessen wird nach Zugabe kleiner Portionen kurz gewartet, um den bereits in der Lösung befindlichen Proteinen Zeit zur Faltung zu geben. Gefaltete Proteine aggregieren nicht mehr, wodurch das Risiko, dass ungefaltete Proteine aufeinandertreffen und miteinander Einschlusskörper bilden, vermindert wird.
Expression ins Periplasma
Im Periplasma von Prokaryoten herrschen, anders als im Cytosol, oxidierende Bedingungen. Ein GSG/GSSG-System gibt es hier nicht, denn die äußere Membran ist für Proteine, die kleiner sind als 500 Da, permeabel (GSH hat eine molare Masse von nur 307,3 g/mol). Durch die äußere Membran kann auch Sauerstoff diffundieren. Die rekombinante Expression von Proteinen mit Disulfidbindungen wird deshalb auch mit dem Periplasma als Zielort erforscht.
Einzelnachweise
- Hans-Dieter Jakubke, Hans Jeschkeit: Aminosäuren, Peptide, Proteine, Verlag Chemie, Weinheim, S. 101, 1982, ISBN 3-527-25892-2.
- Creighton, TE. Protein folding coupled to disulphide bond formation. Biol Chem. 1997 Aug, 378(8):731–744.
Literatur
- W. Thieman, M. Palladino: Biotechnologie. Pearson Studium, Deutschland, 2007, ISBN 978-3-8273-7236-9