Triosephosphatisomerase
Die Triosephosphatisomerase (TIM, TPI) ist das Enzym, das Dihydroxyacetonphosphat (DHAP) zu Glycerinaldehyd-3-phosphat (GAP) umwandelt. Dies ist ein Teilschritt der Glycolyse. TPI ist damit unverzichtbar für alle Lebewesen, die Glucose oder Fructose nur mittels Glycolyse verwerten können.
| Triosephosphatisomerase | ||
|---|---|---|
 | ||
| Bändermodell nach PDB 2jk2 | ||
| Eigenschaften des menschlichen Proteins | ||
| Masse/Länge Primärstruktur | 248 Aminosäuren | |
| Sekundär- bis Quartärstruktur | Homodimer | |
| Isoformen | 2 | |
| Bezeichner | ||
| Gen-Name | TPI1 | |
| Externe IDs | ||
| Enzymklassifikation | ||
| EC, Kategorie | 5.3.1.1, Isomerase | |
| Substrat | Dihydroxyacetonphosphat (=Glyceronphosphat) | |
| Produkte | D-Glycerinaldehyd-3-phosphat | |
| Vorkommen | ||
| Homologie-Familie | CLU_024251_2_0 | |
| Übergeordnetes Taxon | Chordatiere | |
| Orthologe | ||
| Mensch | Hausmaus | |
| Entrez | 7167 | 21991 |
| Ensembl | ENSG00000111669 | ENSMUSG00000023456 |
| UniProt | P60174 | P17751 |
| Refseq (mRNA) | NM_000365 | NM_009415 |
| Refseq (Protein) | NP_000356 | NP_033441 |
| Genlocus | Chr 12: 6.87 – 6.87 Mb | Chr 6: 124.81 – 124.81 Mb |
| PubMed-Suche | 7167 | 21991 |
Im Menschen kodiert ein Gen (TPI1) auf Chromosom 12, Locus 12p13 das funktionelle Protein, mindestens drei Pseudogene sind bekannt. Mutationen am Gen können Triosephosphat-Isomerase-Defizienz verursachen.
Ein potenter Inhibitor ist 2-Phosphoglycolat, was in Pflanzen im Zuge der Photorespiration abgebaut wird.[1]
Struktur
Die Triosephosphatisomerase ist ein Mitglied der all-α- und all-β-Klasse (α/β) von Proteinen und ein Homodimer, das aus zwei sequenzidentischen Untereinheiten (Ketten) mit jeweils 247 Aminosäuren besteht. Jedes TPI-Monomer (Kette) enthält den vollständigen Satz katalytischer Aminosäurereste, jedoch ist das Enzym nur in der oligomeren Form aktiv.[2] Daher ist die Dimerisierung für die volle Funktion des Enzyms wesentlich, obwohl nicht angenommen wird, dass eine Kooperativität zwischen den beiden aktiven Zentren besteht.[3]
Jede Untereinheit enthält 8 äußere α-Helices, die 8 innere β-Stränge umgeben und eine konservierte Strukturdomäne bilden, die als geschlossenes α/β-Barrel (α/β) oder genauer gesagt als TIM-Fass (engl. TIM barrel) bezeichnet wird. Der TIM-Fass wurde ursprünglich nach dem Enzym benannt und ist schätzungsweise in 10 % aller Enzyme enthalten. Charakteristisch für die meisten TIM-Fass-Domänen ist das Vorhandensein des aktiven Zentrums des Enzyms in den Regionen der unteren Schleife, die durch die acht Schleifen erzeugt werden, die die C-Termini der β-Stränge mit den N-Termini der α-Helices verbinden. TIM-Fassproteine teilen auch ein strukturell konserviertes Phosphatbindungsmotiv mit der Phosphatgruppe, die sich im Substrat oder in den Cofaktoren befindet.[4]
In jeder Kette tragen unpolare Aminosäuren, die ausgehend von den β-Strängen nach innen weisen, zum hydrophoben Kern der Struktur bei. Die α-Helices sind amphipathisch: Ihre äußeren (mit Wasser in Kontakt tretenden) Oberflächen sind polar, während ihre inneren Oberflächen weitgehend hydrophob sind. Die Schleifen sind eine Mischung aus polaren und unpolaren Aminosäureresten.[5]
Katalysiertes Gleichgewicht
![]()
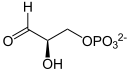
TPI stellt ein Gleichgewicht zwischen den Zwischenprodukten Dihydroxyacetonphosphat (DHAP) und Glycerinaldehyd-3-phosphat (GAP) her. Die Substrate entstehen aus Fructose-1,6-Bisphosphat in der vorgelagerten Aldolase-Reaktion. Das Gleichgewicht liegt stark auf der Seite des DHAP, für den Fortlauf der Glycolyse wird allerdings GAP benötigt, so dass sich das Gleichgewicht durch Produktentnahme verschiebt (Prinzip von Le Chatelier).
Die Katalyse erfolgt über ein Endiol- bzw. Endiolat-Intermediat. Hierbei tritt ein Glutamat-Rest (Glu165) im aktiven Zentrum des Enzyms mit dem ungewöhnlich hohen pKs-Wert von 6,5 als Base auf, ein Histidin-Rest (His95) als Säure.
Die Umsetzung von DHAP zu GAP erfordert einen ausgefallenen Reaktionsmechanismus, in dessen Verlauf Glu165 ein H+-Ion vom C-Atom 2 abstrahiert, während His95 ein H+-Ion ans C1-Atom abgibt. Dieser Mechanismus kann, da die Carboxygruppe des Glu viel azider (saurer) als das C2-Atom ist, unter nicht-enzymatischen Bedingungen keinesfalls ablaufen. Die TIM bildet durch die ideal auf das Substrat angelegte Umgebung des aktiven Zentrums hingegen sog. Low-barrier hydrogen bonds (LBHB) aus, eine spezielle Art von Wasserstoff-Brücken, die mit −40 bis −80 kJ/mol (anstatt etwa −12 bis −30 kJ/mol) deutlich stabiler sind. Diese LBHB werden durch gleichzeitige Protonierung und Deprotonierung an den C-Atomen C1 bzw. C2 erreicht.

Eine 10 Aminosäuren lange Sequenz des Enzyms, ein sogenannter Loop, verdeckt das aktive Zentrum im substratbeladenen Zustand. Einerseits wird damit das Endiol-Zwischenprodukt stabilisiert und die katalytische Aktivität auf diese Weise um den Faktor 105 erhöht, andererseits wird ein Entweichen dieses Zwischenprodukts verhindert – das Endiolphosphat würde spontan dephosphorylieren und sich zum toxischen Methylglyoxal umlagern.
Die TIM gilt als „katalytisch perfektes Enzym“. Dies bedeutet, dass Veränderungen am Enzym, gleich welcher Art, keine Umsatzsteigerung mehr herbeizuführen vermögen: Die Wechselzahl von 4300 Substratmolekülumsätzen pro Sekunde wird nur durch die Diffusionsgeschwindigkeiten von Substrat und Produkt begrenzt.
Weblinks
Einzelnachweise
- Anderson, LE. (1971): Chloroplast and cytoplasmic enzymes. II. Pea leaf triose phosphate isomerases. In: Biochim Biophys Acta. 235(1); 237–244; PMID 5089710; doi:10.1016/0005-2744(71)90051-9.
- C. Rodríguez-Almazán, R. Arreola, D. Rodríguez-Larrea, B. Aguirre-López, M. T. de Gómez-Puyou, R. Pérez-Montfort, M. Costas, A. Gómez-Puyou, A. Torres-Larios: Structural basis of human triosephosphate isomerase deficiency: mutation E104D is related to alterations of a conserved water network at the dimer interface. In: Journal of Biological Chemistry. Band 283, Nummer 34, August 2008, S. 23254–23263, doi:10.1074/jbc.M802145200, PMID 18562316.
- K. D. Schnackerz, R. W. Gracy: Probing the catalytic sites of triosephosphate isomerase by 31P-NMR with reversibly and irreversibly binding substrate analogues. In: European Journal of Biochemistry. Band 199, Nummer 1, Juli 1991, S. 231–238, doi:10.1111/j.1432-1033.1991.tb16114.x, PMID 2065677.
- A. Marchler-Bauer, Y. Bo, L. Han, J. He, C. J. Lanczycki, S. Lu, F. Chitsaz, M. K. Derbyshire, R. C. Geer, N. R. Gonzales, M. Gwadz, D. I. Hurwitz, F. Lu, G. H. Marchler, J. S. Song, N. Thanki, Z. Wang, R. A. Yamashita, D. Zhang, C. Zheng, L. Y. Geer, S. H. Bryant: CDD/SPARCLE: functional classification of proteins via subfamily domain architectures. In: Nucleic acids research. Band 45, D101 2017, S. D200–D203, doi:10.1093/nar/gkw1129, PMID 27899674, PMC 5210587 (freier Volltext).
- E. Lolis, G. A. Petsko: Crystallographic analysis of the complex between triosephosphate isomerase and 2-phosphoglycolate at 2.5-A resolution: implications for catalysis. In: Biochemistry. Band 29, Nummer 28, Juli 1990, S. 6619–6625, doi:10.1021/bi00480a010, PMID 2204418.