Primärstruktur
Unter Primärstruktur versteht man in der Biochemie die unterste Strukturebene eines Biopolymers, d. h. die Abfolge (Sequenz) seiner Grundbausteine. Da Proteine aus Aminosäuren bestehen, wird ihre Primärstruktur Aminosäuresequenz genannt. Entsprechend trägt diese bei Nukleinsäuren (DNA und RNA) den Namen Nukleotidsequenz. In der Chemie und in der Werkstoffkunde bezeichnet der Begriff Primärstruktur zudem die Sequenz synthetischer Polymere (Kunststoffe).
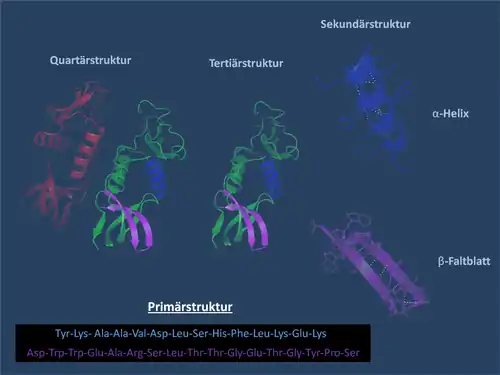
Auswirkungen auf die Gestalt des Proteins
Die Gestalt der höheren Strukturebenen (Sekundärstruktur, Tertiärstruktur, Quartärstruktur) eines Proteins geht aus der Primärstruktur hervor. Sie ist bereits durch die Sequenz der Aminosäuren festgelegt. Bereits während der Translation bildet sich die Sekundärstruktur meist in ihrer endgültigen Form infolge der Wechselwirkungen zwischen den Aminosäuren. In einigen Fällen sind an diesem Prozess Enzyme und andere Umgebungseinflüsse beteiligt (siehe auch: Prion). Aus der Sekundärstruktur geht wiederum die räumliche Strukturerfüllung (Tertiärstruktur) und gegebenenfalls die Komplexierung mit anderen Untereinheiten zu Proteinkomplexen (Quartärstruktur) hervor.
Zurzeit existiert noch keine verlässliche Methode zur Vorhersage der exakten räumlichen Anordnung der Aminosäurekette anhand der Primärstruktur. Aus Erfahrungswerten lassen sich in der Regel jedoch Aussagen über vorhandene Strukturelemente und die Funktion des Proteins treffen.
Zusammenhang zwischen Nukleotidsequenz und Aminosäuresequenz
Die Aminosäuresequenz eines Proteins kann aus der Nukleotidsequenz der Nukleinsäure, in der es codiert ist, abgeleitet werden, da der genetische Code bekannt ist und jedes Codon für nur eine Aminosäure codiert ist. Umgekehrt ist das nicht möglich, da die meisten Aminosäuren mehr als nur ein Codon haben. Sie können also durch unterschiedliche Nukleotidsequenzen codiert sein. Der genetische Code wird daher als degeneriert bezeichnet.[1]
Die Umsetzung der genetischen Information eines Gens in die Aminosäuresequenz eines Proteins ist Teil der Genexpression und der Proteinbiosynthese. Erster Teil dieser gesteuert ablaufenden Prozesse ist die Transkription, der zweite ist die Translation.
Für die Angabe der Primärstruktur von Proteinen und Nukleinsäuren existieren vereinbarte Konventionen.
- Die Aminosäuresequenz von Proteinen wird vom aminoterminalen Ende (N-Terminus) zum carboxyterminalen Ende (C-Terminus) geschrieben.
- Die Nukleotidsequenz von Nukleinsäuren (DNA, RNA) wird vom 5'-Phosphat-Ende zum 3'-Hydroxy-Ende geschrieben.
Analyse der Primärstruktur
Proteine
Der Edman-Abbau ist die klassische Methode zur Sequenzierung von Proteinen und besteht aus drei Schritten:
- Markierung der ersten N-terminalen Aminosäure durch Phenylisothiocyanat.
- Abspaltung der markierten Aminosäure.
- Identifikation der abgespaltenen Aminosäure, z. B. durch HPLC oder durch Ionenaustauschchromatographie.
Zur Sequenzierung der nächsten Aminosäure folgt die Wiederholung der 3 Schritte.
Die Methode wurde weitgehend automatisiert und funktioniert für Peptide bis zu einer Länge von ca. 50 Aminosäuren. Größere Proteine werden vor der Analyse in Fragmente gespalten, die getrennt sequenziert werden. In letzter Zeit gewinnen verstärkt massenspektroskopische Methoden an Bedeutung in diesem Bereich.[2]
DNA
Frederick Sanger entwickelte 1976 eine Methode zur DNA-Sequenzierung. Diese wird Didesoxymethode oder Kettenabbruchmethode genannt, da hierbei durch den Einbau von Didesoxy-Nukleotiden in die DNA-Synthese ein Abbruch der Synthesereaktion bewirkt wird. Durch Zugabe einer kleinen Menge eines bestimmten Didesoxynukleotids zur Synthesereaktion wird das entsprechende Desoxynukleotid teilweise durch das Didesoxynukleotid ersetzt, was den Abbruch der Reaktion an dieser Stelle bewirkt. Es ergeben sich unterschiedlich lange DNA-Fragmente aus deren Länge die Stellung des ersetzten Desoxynukleotids abgeleitet werden kann.[3]
Auch dieses Verfahren wurde weitgehend automatisiert. Die Auftrennung durch Elektrophorese erfolgt meist in einer gemeinsamen Gelspur. Die Fragmente werden durch Fluoreszenzmarkierungen voneinander unterschieden, die durch einen Laser detektiert werden.[4]
RNA
RNA kann durch das Enzym Reverse Transkriptase in cDNA umgeschrieben und diese DNA sequenziert werden (siehe oben).[5]
Einzelnachweise
- Löffler, Petrides, Heinrich: Biochemie und Pathobiochemie, 8. Auflage, Springer Medizin Verlag, Heidelberg (2007), S. 289, ISBN 978-3-540-32680-9.
- Wilson, Walker: Principles and Techniques of Biochemistry and Molecular Biology, 6. Auflage, Cambridge University Press, New York (2005), S. 380 ff., ISBN 978-0-521-53581-6.
- Horton, Robert et al.: Biochemie, 4. Auflage, Pearson Studium, München (2008), S. 840f., ISBN 978-3-8273-7312-0.
- Strachan, Read: Human Molecular Genetics, 4. Auflage, Garland Science, New York (2011), S. 217 ff., ISBN 978-0-8153-4149-9.
- Strachan, Read: Human Molecular Genetics, 4. Auflage, Garland Science, New York (2011), S. 183, ISBN 978-0-8153-4149-9.