Escherichia-Virus T4
Escherichia-Virus T4 (offiziell Escherichia virus T4, früher T-even phages) ist eine Spezies – die Typusspezies – von Viren in der Gattung Tequatrovirus (früher T4virus, T4likevirus, T4-ähnliche Viren), die Escherichia-coli-Bakterien infizieren, und daher auch als Bakteriophagen bezeichnet werden. Der Typus-Stamm (oder Subtyp) dieser Spezies ist der T4-Phage (auch Enterobacteria phage T4, Bakteriophage T4, T4-Bakteriophage). Er gilt mit seiner Morphologie als Prototyp der Bakteriophagen und hatte in der virologischen Forschung große Bedeutung für das Verständnis der Vermehrungsstrategie von Phagen und Viren und der allgemeinen Genetik (siehe auch Hershey-Chase-Experiment, Max Delbrück). Die Spezies umfasst neben T4 unter anderem die Subtypen T2 und T6, woher die im Englischen gebräuchliche Bezeichnung T-even bacteriophages kommt (zu unterscheiden von der Unterfamilie Tevenvirinae, die außer dieser Gattung noch weit mehr Virustaxa umfasst).
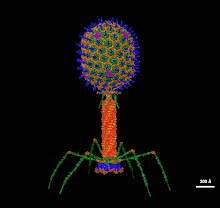
| Escherichia-Virus T4 | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|

Elektronenmikroskopische Aufnahme | ||||||||||||||||||||
| Systematik | ||||||||||||||||||||
| ||||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||||
| ||||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||||
| Escherichia virus T4 | ||||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||||
| T4 | ||||||||||||||||||||
| Links | ||||||||||||||||||||
|
Struktur
Als Mitglied der Ordnung Caudovirales, Familie Myoviridae, Gattung Tequattrovirus (alias T4virus, T4likevirus)[3] besteht das Genom des T4-Phagen aus einem einzigen doppelsträngigen DNA-Molekül mit einer Länge von 168.903 bp. Es umfasst etwa 300 Gene und macht 48 % des Gewichtes des Virions aus. Die DNA enthält neben den normalen Nukleotiden anstelle von Cytosin das 5-Hydroxymethylcytosin, das zusätzlich durch einen Glucose-Rest glykosyliert sein kann. Diese Glykosylierung der DNA ist vermutlich für die Steuerung der Genexpression mit verantwortlich. Die Leserichtungen des Genoms sind zirkulär abwechselnd vertauscht und an den Enden gibt es redundante Abschnitte. Innerhalb des Kapsids ist die DNA nach einem komplexen Packungsschema regelmäßig angeordnet.
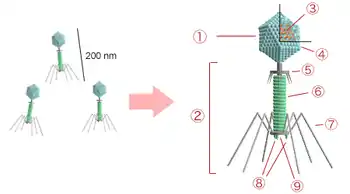
Das Kapsid des T4-Phagen ist grundsätzlich ikosaedrisch, jedoch ist eine von der Grundform abweichende, längliche Streckung besonders typisch für diese Spezies („verlängerte, fünfeckige, bipyramidale Antiprismen“). Es besteht aus 152 Kapsomeren und ist etwa 111 nm lang und 78 nm im Durchmesser groß. Myoviridae besitzen komplexe kontraktile Schwanzstücke, die aus einem Halsstück (Kragen), einer Grundplatte, an der sechs kurze Spikes und sechs lange Schwanzfibern ansetzen. Insgesamt ist das Schwanzstück 113 × 16 nm groß mit einem Molekulargewicht von etwa 2 × 107 Da aus 430 Polypeptideketten von 22 Genen.[4] Bei der Replikation des T4-Phagen entstehen eine ganze Reihe an strukturell instabilen oder nicht DNA-verpackende Fehlbildungen (Mehrfachkapside, nicht gestreckte Ikosaeder) des Kapsids, was aufgrund des hochkomplexen Aufbaus nicht verwundert. Das Virion besteht insgesamt aus 49 verschiedenen Proteinen. Eine detaillierte Schemazeichnung der T4-Virionen mit kontrahiertem und unkontrahiertem Schwanz findet sich bei EurekAlert (Mai 2016).[5]
Es können durch unterschiedliche Eigenschaften der Antigenbindung acht serologische Untergruppen von Escherichia-Virus T4 unterschieden werden. Taxonomische Subtypen der Spezies Escherichia-Virus T4 sind: Enterobacteria phage C16, F10, Fsα, PST, SKII, SKV, SKX, SV3, T2, T4 (Typus-Stamm) und T6. Der T2-Phage (früher eine eigene Spezies) ist damit genauso wie der T6-Phage nur ein Subtyp des Escherichia-Virus T4.
Infektionszyklus
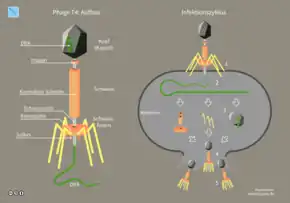
Der Infektionszyklus dauert ca. 30 min. Hierzu dockt der Phage mit den Enden der Schwanzfasern an Porinproteine der äußeren Membran von E. coli an. Der Schwanzstachel mit Lysozymfunktion durchbricht die Zellwand enzymatisch und die Phagen-DNA wird durch den Schwanz hindurch in das Innere der Zelle injiziert. Dort wird die DNA zuerst von bakteriellen Enzymen transkribiert und translatiert. Die neu entstandene Phagen-Polymerase repliziert nun die Phagen-Gene. Schon währenddessen beginnt die Synthese des Phagenkapsids. In dieses wird schließlich die replizierte DNA verpackt. Zuletzt wird Phagen-spezifisches Lysozym synthetisiert, das die Zellwand des Bakteriums zerstört. Dadurch werden die reifen Phagen freigesetzt.
Besonderheiten
Die T4-Phagen verfügen über einen äußerst schnellen DNA-Synthese-Apparat, dank der viralen DNA-Polymerase Typ B mit einer Fehlerrate von nur einem Austausch auf 300 Genom-Kopien. Sie besitzt spezielle DNA-Reparaturmechanismen, die sonst nur in eukaryotischen Zellen vorkommen. Dadurch besitzt der T4-Phage trotz hoher Replikationsrate eine hohe genetische Stabilität.
In der Molekularbiologie und der Gentechnik ist die T4-DNA-Ligase von großer Bedeutung, sie wird auch kurz als T4-Ligase bezeichnet. Auch die T4-Polynucleotidkinase, die T4-RNA-Ligase und die T4-DNA-Polymerase sind wichtige Enzyme in der molekularbiologischen Forschung.
Die Phagen dieser Spezies (T-even phages:T2, T4 und T6) enthalten in ihrer DNA anstelle der Pyrimidinbase Cytosin die Nichtstandard-Base 5-Hydroxymethylcytosin.[6]
Historisches
Bakteriophagen wurden 1915 von dem englischen Wissenschaftler Frederick Twort und 1917 von Félix d'Hérelle entdeckt. Die Benennung der ersten untersuchten Bakteriophagen in
- Typ (englisch Type) 1 (T1), Tunaviridae,
- Typ 2 (T2), Subtyp der Spezies hier,
- Typ 3 (T3), Autographiviridae,
- Typ 4 (T4), Typusstamm der Spezies hier,
- Typ 5 (T5), Demerecviridae,
- Typ 6 (T6), Subtyp der Spezies hier, und
- Typ 7 (T7), Autographiviridae
stammt von Max Delbrück. Diese sieben Phagen werden manchmal unter der Sammelbezeichnung T-Phagen (en. T phages) zusammengefasst,[6][7][8] was aber keine Verwandtschaftsgruppe (Taxon) darstellt. Stattdessen werden diese Bakteriophagen vom ICTV (mit Stand Januar 2021) nach einigen Verschiebungen den oben angegebenen Familien zugeordnet. Lediglich die Phagen mit gerader Typ-Nummer (T-even phages) erwiesen sich zufällig als nahe miteinander verwandt, so dass für vom ICTV die in diesem Artikel beschriebene Spezies eingerichtet wurde. Die Typen mit ungerader Nummer (T-odd/T-uneven phages) bilden jedoch kein Taxon. Allerdings ist allen diesen Phagentypen ein Kopf-Schwanz-Aufbau gemeinsam, was sie als Vertreter der Virusordnung Caudovirales kennzeichnet. Später wurde von anderen Autoren diese Gepflogenheit bei der Benennung anderer Caudoviren teilweise weitergeführt (z. B. „T12“, Vorschlag, ohne Familienzuordnung)[9].
Literatur
- C. M. Fauquet, M. A. Mayo et al.: Eighth Report of the International Committee on Taxonomy of Viruses. Academic Press, Amsterdam u. a. 2005, ISBN 0-12-249951-4.
- J. Cairns, G. S. Stent, J. D. Watson (Hrsg.): Phage and the Origins of Molecular Biology. The Centennial Edition. CSHL Press, Cold Spring Harbor, NY, 2007, ISBN 978-0-87969-800-3.
- G. Fuchs: Allgemeine Mikrobiologie. 9. Auflage. Georg Thieme Verlag, Stuttgart 2014, ISBN 978-3-13-444609-8.
Weblinks
Einzelnachweise
- Victor Padilla-Sanchez: Structural Model of Bacteriophage T4. In: WikiJournal of Science. 4, Nr. 1, 2021, S. 5. doi:10.15347/WJS/2021.005.
- ICTV: ICTV Taxonomy history: Enterobacteria phage T4, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- Myoviridae - dsDNA Viruses - dsDNA Viruses (2011). Abgerufen am 7. August 2019 (englisch).
- Petr G Leiman, Fumio Arisaka, Mark J van Raaij, Victor A Kostyuchenko, Anastasia A Aksyuk: Morphogenesis of the T4 tail and tail fibers. In: Virology Journal. Band 7, Nr. 1, Dezember 2010, ISSN 1743-422X, doi:10.1186/1743-422X-7-355, PMID 21129200, PMC 3004832 (freier Volltext) – (Online [abgerufen am 7. August 2019]).
- How viruses infect bacteria: A tale of a tail, auf: EurekAlert! vom 18. Mai 2016. Quelle: Ecole Polytechnique Fédérale de Lausanne
- T-Phages, auf: Harvard Catalyst Profiles. Abgerufen am 31. Januar 2021.
- NCBI: T-Phages
- Rolf Sauermost, Doris Freudig et al.: T-Phagen, Lexikon der Biologie, auf: Spektrum.de, abgerufen am 31. Januar 2021. Die Familienzuordnungen entsprechen nicht mehr den aktuellen Stand nach ICTV.
- L. McKane, J. J. Ferretti: Phage-host interactions and the production of type A streptococcal exotoxin in group A streptococci. In: Infection and Immunity. Band 34, Nr. 3, Dezember 1981, S. 915–9, PMID 7037644, PMC 350956 (freier Volltext).