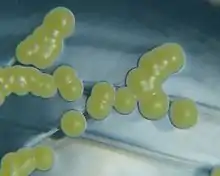
Micrococcus luteus
Micrococcus luteus ist ein grampositives Bakterium aus der Gattung Micrococcus. Der Name der Gattung wird auch eingedeutscht Mikrokokkus geschrieben. Seine Zellen sind aerob, sie können sich also nur vermehren, wenn Sauerstoff vorhanden ist. Als sogenannter „Luftkeim“ ist er in der Luft vorhanden, er ist aber auch Teil der normalen Hautflora des Menschen und gilt als nicht krankheitserregend. Auf Nährböden wächst er als gelb gefärbte Kolonie.[1] Bereits Alexander Fleming untersuchte 1921 die Wirkung des von ihm entdeckten Lysozyms auf das Bakterium.[2]
| Micrococcus luteus | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
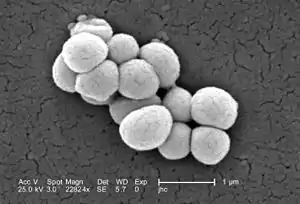
Micrococcus luteus | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Micrococcus luteus | ||||||||||||
| (Schröter 1872) Cohn 1872 emend. Wieser u. a. 2002 |
Die Spezies umfasst zahlreiche Stämme.[3] Das Genom des Stammes Micrococcus luteus NCTC 2665 wurde 2007 vollständig sequenziert,[4] es folgten weitere Genomanalysen anderer Stämme. Die Systematik der Art, wie auch der Gattung, wurde seit Ende der 1990er Jahre mehrfach aktualisiert. Die 2013 der Art Micrococcus luteus zugeordneten Stämme unterscheiden sich in außerordentlich vielen Merkmalen.[5][6] Von Bedeutung ist M. luteus in der Lebensmittelmikrobiologie bei der Bestimmung des Keimgehaltes, sowie als Indikator bei der Überprüfung von Antibiotika.
Merkmale
Erscheinungsbild
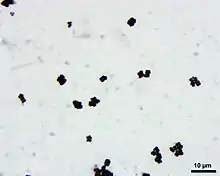
Die Zellform des Bakteriums ist rund bis oval, es handelt sich um Kokken. Ihr typisches Erscheinungsbild im lichtmikroskopischen Bild kommt durch eine Besonderheit bei der Zellteilung zustande: Die Zellen trennen sich nicht nach jeder Teilung vollständig, sondern bleiben mit der Zellwand aneinander hängen. So entstehen Pakete aus vier zusammenhängenden Kokken, sogenannte Tetraden.[2] Darüber hinaus kommen auch Pakete aus zwei Kokken (Diplokokken) vor. Eine einzelne Zelle hat einen Durchmesser von etwa 0,5–3,5 µm.[7] In der Gram-Färbung verhält sich M. luteus grampositiv, er wird also durch die verwendeten Farbstoffe blau angefärbt. Verursacht wird dies durch eine dickere Mureinschicht in der Zellwand. Er besitzt keine Flagellen zur aktiven Bewegung, kann keine Überdauerungsformen wie Endosporen bilden,[6] und die Bakterienzellwand ist nicht von einer Kapsel umgeben.
In Reinkultur bildet er auf festen, Glucose-haltigen Nährböden schwefelgelbe bis goldgelbe Kolonien.[8] Wenn kein Kohlenhydrat, sondern nur Pepton enthalten ist, sind die Kolonien hellgelb bis cremefarben gefärbt.[9] Diese – auch in seinem Namen vermerkte – Pigmentierung ist auf das Vorhandensein von gelb gefärbten Carotinoiden zurückzuführen.[2]
Wachstum und Stoffwechsel
Micrococcus luteus ist strikt aerob,[1] er benötigt also Sauerstoff zum Wachsen, dies dient als Unterscheidungsmerkmal zu Vertretern der Familie der Staphylococcaceae, die Glucose auch anaerob in einer Gärung unter Säurebildung verstoffwechseln. Das Bakterium ist Katalase-positiv und Oxidase-positiv.[6][8] Die zur Kultivierung üblicherweise verwendeten Temperaturen liegen im Bereich von 25–37 °C,[8] das Temperaturoptimum liegt bei 28 °C,[9] somit zählt das Bakterium zu den mesophilen Organismen. Der optimale pH-Wert für das Wachstum liegt bei dem neutralen Wert pH 7,0,[8] wobei auch alkalische pH-Werte bis pH 10,0 toleriert werden.[5] M. luteus ist relativ unempfindlich gegenüber Trockenheit und hohen Salzkonzentrationen,[1] er wächst noch in Nährmedien mit 10 % Natriumchlorid (Kochsalz), während bei einem Anteil von 15 % kein Wachstum mehr erfolgt.[5]
Charakteristisch ist sein aerober Stoffwechsel,[6] er nimmt oxidierbare Substrate auf, die er mit Hilfe von Sauerstoff oxidiert – dies geschieht mit Hilfe der Atmungskette – und nutzt die dabei freiwerdende Energie. Dieser Prozess wird auch bei Bakterien als Atmung bezeichnet. Weiterhin ist sein Stoffwechsel als chemoorganotroph und heterotroph zu kennzeichnen, er benutzt organische Verbindungen als Energiequelle und ebenso zum Aufbau zelleigener Stoffe.[6] So wird beispielsweise Glucose als organisches Substrat unter Oxidation mit Sauerstoff zu Kohlenstoffdioxid und Wasser abgebaut:
- Reaktionsgleichung
- Glucose + Sauerstoff reagieren zu Kohlenstoffdioxid + Wasser
Kohlenhydrate, die auf diesem Weg verstoffwechselt werden, sind unter anderem D-Glucose, D-Mannose und Saccharose.[5] Als Reservestoff kann das Bakterium Glykogen – ein Polysaccharid aus Glucoseeinheiten – in der Zelle speichern,[2] um dieses bei Bedarf wieder zu Glucose zur Energiegewinnung abzubauen.
M. luteus besitzt eine Reihe von Enzymen, die im Stoffwechsel verwendet werden, um bestimmte Substrate abzubauen und deren Nachweise zur Identifizierung im Rahmen einer „Bunten Reihe“ verwendet werden. So besitzt er das Enzym Nitratreduktase (NADH) (EC 1.7.1.1) und kann somit Nitrat (NO3−) zu Nitrit (NO2−) reduzieren. Das Enzym Urease zum Abbau von Harnstoff ist nicht bei allen Stämmen vorhanden. Außerdem verfügt er über proteolytische Enzyme, mit denen er Proteine (Eiweiße) abbauen kann.[8]
Das Bakterium reagiert sehr empfindlich auf Lysozym, ein antibakteriell wirkendes Enzym, das in Hühnereiklar, in Tränenflüssigkeit und im Nasenschleim vorhanden ist. Alexander Fleming untersuchte 1921 die Wirkung von Lysozym auf das Bakterium und beobachtete eine rasche Lyse („Auflösung“) der Zellen in einem flüssigen Medium.[10] Der Grund liegt im Aufbau der Zellwand: Bei grampositiven Bakterien wie M. luteus besteht diese aus vielen Mureinschichten, die Vernetzung der Glucose-ähnlichen Bausteine wird durch Lysozym gespalten und damit die Zellwand zerstört. Bereits 1 µg/ml (Mikrogramm pro Milliliter) Lysozym ist bei M. luteus wirksam, während beim ebenfalls grampositiven Bacillus megaterium eine Konzentration von 50 µg/ml für die Lyse nötig ist.[2] Fleming benannte das untersuchte Bakterium 1929 als Micrococcus lysodeikticus,[11] nach aktueller Klassifikation handelt es sich um den Stamm Micrococcus luteus DSM 20030 (auch als Micrococcus luteus Fleming NCTC 2665 bezeichnet).[12]
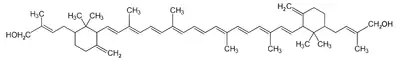
Die meist gelbe Farbe seiner Kolonien ist ein typisches Merkmal und lässt sich auf das Vorhandensein von Sarcinaxanthin zurückführen, einem gelben Farbstoff aus der Gruppe der Xanthophylle, die zu den Carotinoiden gehören.[13] Der Farbstoff ähnelt in seiner Struktur dem Zeaxanthin (gelber Farbstoff im Mais). Die Pigmentierung von „Luftkeimen“, also Mikroorganismen, die in der Luft zu finden sind, lässt sich häufig beobachten. Die Farbstoffe dienen als Schutz gegenüber den UV-Strahlen und den Strahlen des sichtbaren Lichts. Die pigmentierten Bakterien haben an Standorten, die dem Licht stark ausgesetzt sind, einen Vorteil gegenüber farblosen Bakterien, die rascher abgetötet werden. Die Pigmente in der Zellmembran schützen vor der Photooxidation, bei der z. B. die Cytochrome, wichtige Proteine in der Atmungskette, zerstört werden.[2]
Diese antioxidative Wirkung von Sarcinaxanthin und davon abgeleiteten Verbindungen wurde durch eine Untersuchung aus dem Jahr 2010 bestätigt.[14] Sarcinaxanthin gehört zu den C50-Carotinoiden, sie enthalten 50 Kohlenstoffatome im Molekül. Derartige Verbindungen sind als natürliche Farbstoffe selten, bei den meisten bisher aus natürlichen Quellen isolierten Carotinoiden handelt es sich um C40-Carotinoide. Die Biosynthese des C50-Carotinoids Sarcinaxanthin erfolgt aus zwei Molekülen Farnesyldiphosphat (C20) als Vorstufe, und umfasst Lycopin (C40), Nonaflavuxanthin (C45), und Flavuxanthin (C50) als Zwischenstufen.[13]
Genetik
Das Genom des Stammes Micrococcus luteus Fleming NCTC 2665 wurde im Jahr 2007 vollständig sequenziert, 2009 wurde darüber im Journal of Bacteriology berichtet.[15] Der für die Untersuchung verwendete Bakterienstamm lässt sich auf den von Alexander Fleming 1929 als Micrococcus lysodeikticus bezeichneten Bakterienstamm zurückführen. Das Genom weist eine Größe von 2501 Kilobasenpaaren (kb) auf,[4] das ist in etwa die Hälfte der Genomgröße von Escherichia coli. Es sind 2236 Proteine annotiert.[16] Im Jahr 2010 wurde das Genom eines weiteren Stammes – Micrococcus luteus SK58 – sequenziert, dieser Stamm wurde von der menschlichen Haut isoliert. Die Genomgröße fällt mit 2623 kb[17] etwas größer aus als bei dem zuerst untersuchten Stamm, es sind 2489 Proteine annotiert.[16] Ende 2012 folgte die Sequenzierung des Genoms des Stammes Micrococcus luteus modasa, dieser wurde in der indischen Stadt Modasa aus mit Kohlenwasserstoffen kontaminiertem Boden isoliert.[18][19] Die Variabilität der M. luteus Stämme wird zurzeit (2019) in zahlreichen Genomprojekten erforscht.[16]
Die Ergebnisse der Sequenzierungen zeigen einen auffallend hohen GC-Gehalt (den Anteil der Nukleinbasen Guanin und Cytosin) in der Bakterien-DNA, er liegt bei etwa 73 Mol-Prozent.[16][18] Dies beweist, dass M. luteus nicht mit Sarcina-Arten verwandt ist, die sich durch besonders niedrigen GC-Gehalt im Genom auszeichnen.[1] Aufgrund der Ähnlichkeit im mikroskopischen Erscheinungsbild wurde das Bakterium früher als Sarcina lutea bezeichnet.[12] Weitere genetische Untersuchungen an M. luteus beinhalten z. B. den Gen-Cluster, der die Enzyme für die Sarcinaxanthin-Biosynthese codiert. Dabei wurde dieser Gen-Cluster „Stück für Stück“ in einen E. coli als Wirt übertragen und so die einzelnen Schritte der Biosynthese aufgeklärt. Darüber hinaus gelang auch die vollständige Genexpression, so dass das Wirtsbakterium Sarcinaxanthin produzierte.[13]
Auch ein Plasmid des Bakteriums dient als Untersuchungsobjekt. Das als pMEC2 bezeichnete Plasmid weist eine Genomgröße von 4,2 kb auf – ist also im Vergleich zum Bakterienchromosom sehr klein – und verleiht M. luteus eine Resistenz gegenüber Makrolidantibiotika wie Erythromycin, sowie gegen Lincomycin. Dabei ist die Makrolid-Resistenz ein induzierbares Merkmal des Bakteriums: Nur wenn das Nährmedium äußerst geringe Mengen (etwa 0,02–0,05 µg/ml) Erythromycin enthält, die noch nicht für eine Hemmung des Bakterienwachstums ausreichen, wirkt dies als Induktor und das entsprechende Genprodukt wird gebildet, wodurch M. luteus die Resistenz erhält. Eine plasmidgebundene, induzierbare Resistenz in den nicht pathogenen, jedoch zur normalen Hautflora gehörenden Bakterien wirft die Frage auf, inwieweit sie an der Verbreitung von Antibiotikaresistenzen beteiligt sind.[20]
Pathogenität
M. luteus ist normalerweise nicht krankheitserregend, er wird durch die Biostoffverordnung in Verbindung mit der TRBA 466 der Risikogruppe 1 zugeordnet.[21] Allerdings wurden Einzelfälle beobachtet, bei denen er bei Patienten mit geschwächtem Immunsystem (beispielsweise durch eine Infektion mit HIV) Infektionen der Haut hervorrief.[22]
Nachweise

Das Bakterium lässt sich gut in flüssigen bzw. auf festen Nährmedien kultivieren, die Pepton und Fleischextrakt enthalten.[12] Ein Selektivmedium ist nicht vorhanden, allerdings kann eine selektive Anreicherung erfolgen, wenn das Medium einen hohen Salzgehalt (7,5 % Natriumchlorid) aufweist und aerob bei etwa 30 °C inkubiert wird. Auch die Pigmentierung der Kolonien ist ein Hinweis auf M. luteus, ebenso wie die im mikroskopischen Bild typischen Zellaggregate in Form der vier zusammenhängenden Zellen (Tetraden). Von den vermutlich auch auf dem Nährmedium gewachsenen, fakultativ anaeroben Staphylokokken kann er durch einen Oxidations-Fermentations-Test (OF-Test) unterschieden werden, da diese sowohl aerob wie auch anaerob Säure aus Glucose bilden, während Mikrokokken Glucose nur mit Sauerstoff verstoffwechseln können.[1] Weitere biochemische Tests zur Identifizierung beinhalten den Katalase- und Oxidase-Test, sowie typische Tests aus einer „Bunten Reihe“, wobei unter anderem auf die Verwertbarkeit verschiedener Kohlenhydrate und anderer Substrate untersucht wird. Dabei verhält sich M. luteus positiv bei der Nitratreduktion, negativ bei der Indol-Bildung, positiv in der Voges-Proskauer-Reaktion[9] und ist Urease-variabel.[8] Ein darauf basierendes Schnellbestimmungssystem im Miniaturformat (Analytical Profile Index) zur Bestimmung von Bakterien aus der Familie Staphylococcaceae ist kommerziell verfügbar und umfasst auch den Nachweis von Micrococcus-Arten.[23] Die Ergebnisse für M. luteus sind in der frei zugänglichen Datenbank BacDive der DSMZ (Deutsche Sammlung von Mikroorganismen und Zellkulturen) einsehbar.[24]
Vorkommen

Micrococcus luteus wird als sogenannter „Luftkeim“ bezeichnet (umgangssprachlich wird er auch „Gelber Luftkokkus“ genannt), da er häufig auf Nährmedien in Petrischalen wächst, die bei einer Luftkeimsammlung verwendet werden.[8] Er ist ubiquitär verbreitet, also fast überall zu finden, neben der Raumluft z. B. auf Staubpartikeln, Gegenständen und in der oberen Bodenschicht. Außerdem ist er Teil der natürlichen Hautflora beim Menschen.[1] Dort ist er überwiegend auf den eher unbekleideten Körperteilen nachweisbar. Über den Menschen oder die Luft wurde er auf andere Habitate verbreitet, wie See- und Süßwasser, Pflanzen, sowie Fleisch- und Milchprodukte.[8]
Dokumentiert ist beispielsweise die Isolierung von M. luteus Stämmen aus dem Boden,[7] Belebtschlamm aus einer Abwasserbehandlungsanlage,[5] Meerwasser,[13] von einem Korallenstück,[14] der menschlichen Haut,[25] mittelalterlicher Wandmalerei,[5] Innenraumluft,[5] aus Käse[26] und verdorbenem Fleisch.[27]
Systematik
Äußere Systematik
Micrococcus luteus wurde 1872 von Ferdinand Cohn nach Vorarbeiten von Joseph Schröter beschrieben.[28] Cohn hatte die Bakterien auf einem Kartoffelnährboden gezüchtet. In derselben Arbeit hatte Cohn erstmals ein auf Darwin aufbauendes Klassifikationsschema für Bakterien mit Gattungs- und Artnamen entwickelt.[29] Erst 1955 gelang die gesicherte Unterscheidung von Micrococcus- und Staphylococcus-Arten.[8] Als Folge davon wurden zu Beginn des 21. Jahrhunderts Staphylokokken und andere Gattungen in die neu beschriebene Familie der Staphylococcaceae eingeordnet, während sie zuvor mit der Gattung Micrococcus in der Familie der Micrococcaceae zusammengefasst wurden.[1] Ende des 20. Jahrhunderts erfolgte eine Neubeschreibung der Gattung Micrococcus durch Stackebrandt u. a. – mit Zuordnung von bisherigen Micrococcus-Arten zu anderen Gattungen.[6] Untersuchungen zu Beginn des 21. Jahrhunderts führten zu einer weiter verbesserten Beschreibung der Gattung Micrococcus, wie auch der Arten M. luteus und M. lylae durch Wieser u. a.[5] Micrococcus luteus ist Typusart der Gattung.[6]
Innere Systematik
Die in früheren Zeiten morphologisch orientierte Systematik in der Mikrobiologie führt dazu, dass M. luteus unter zahlreichen Synonymen bekannt ist. Dies sind die bereits genannten Bezeichnungen Micrococcus lysodeikticus (Versuche von Fleming mit Lysozym) und Sarcina lutea (wegen des mikroskopischen Erscheinungsbildes), aber auch Sarcina citrea und Sarcina flava.[12] Schröter hatte 1872 das Bakterium ursprünglich als Bacteridium luteum bezeichnet.[28]
2019 zeigten phylogenetische Untersuchungen taiwanesischer Wissenschaftler, dass sich die drei Microcoocus-Spezies M. aloeverae, M. yunnanensis und M. luteus genetisch so ähnlich sind, dass sie zu einer Art zugehörig sind. Sie schlugen daher vor, die beiden zuerst genannten Arten als später beschriebene heterotypische Synonyme von M. luteus zu klassifizieren.[30]
Untersuchungen aus den Jahren 1999 bis 2002 zeigen eine erstaunliche Vielfalt der Art Micrococcus luteus, und zwar hinsichtlich chemotaxonomischer Merkmale und biochemischer bzw. stoffwechselphysiologischer Eigenschaften. Zur Aufklärung der Stammesgeschichte – und der verwandtschaftlichen Beziehungen der Organismen untereinander – untersucht man die DNA-Sequenzen und bei Bakterien zusätzlich die 16S rRNA, ein für Prokaryoten typischer Vertreter der ribosomalen RNA. Genetische Untersuchungen der DNA-Sequenzen mittels DNA-DNA-Hybridisierung und die Analyse der 16S rRNA Sequenzen weisen bei M. luteus auf eine nahe Verwandtschaft der untersuchten Stämme hin, so dass sie alle der Art zugeordnet bleiben oder neu zugeordnet werden.[5] Folgende Stämme werden unterschieden (Stand 2019):
- Micrococcus luteus NCTC 2665 ist der speziestypische Stamm. Synonyme dafür sind Micrococcus luteus NCIB 9278, Micrococcus luteus CCM 169, Micrococcus luteus ATCC 4698 (entspricht ATCC 15307), Micrococcus luteus DSM 20030, Micrococcus luteus CN 3475, Micrococcus luteus Fleming NCTC 2665. Gleichbedeutende Bezeichnungen lauten Micrococcus luteus strain (Stamm) NCTC 2665 und Micrococcus luteus str. NCTC 2665.[3][4] Hierbei handelt es sich um den „Fleming Stamm“, das Genom ist sequenziert (Biovar I).
- Micrococcus luteus SK58. Gleichbedeutende Bezeichnungen lauten Micrococcus luteus strain (Stamm) SK58 und Micrococcus luteus str. SK58.[3] Er wurde von der menschlichen Haut isoliert, das Genom ist sequenziert.
- Micrococcus luteus str. modasa.[3] Er wurde aus kontaminiertem Boden in Indien isoliert, das Genom ist sequenziert.
- Micrococcus luteus MU201.[3] Er wurde aus Käse isoliert,[26] das Genom ist sequenziert.[31]
- Micrococcus luteus CD1_FAA_NB_1.[3] Dieser Stamm wird im Rahmen des Human Microbiome Project (Humanes Mikrobiom Projekt, HMP) untersucht.[32] Das HMP wurde 2008 vom US-amerikanischen National Institutes of Health (Nationale Gesundheitsinstitute) initiiert und hat das Ziel, das menschliche Mikrobiom näher zu charakterisieren. Die Genomsequenzierung dieses Stammes von M. luteus ist unvollständig.[33]
- Micrococcus luteus J28.[3] Das Genom ist sequenziert.[34]
- Micrococcus luteus DSM 14234, Synonym Micrococcus luteus CCM 4959. Er wurde von einer mittelalterlichen Wandmalerei in Österreich isoliert[12] und wurde während der Untersuchung als strain D7 bezeichnet (Biovar II).[5]
- Micrococcus luteus DSM 14235, Synonym Micrococcus luteus CCM 4960. Er wurde aus Belebtschlamm aus einer Abwasserbehandlungsanlage in Ballarat (Australien) isoliert[12] und wurde während der Untersuchung als strain Ballarat bezeichnet (Biovar III).[5]
Die Zuordnung zu drei Biovaren ist das Ergebnis der oben angeführten Untersuchungen. Es wurde auf eine Unterteilung in Subspezies (Unterarten) verzichtet, stattdessen erfolgte eine eher formale Trennung in Biovare. Der Begriff „Biovar“ basiert auf der biologischen Variabilität der untersuchten Organismen.
Im Falle von M. luteus betrifft dies v. a. chemotaxonomische Merkmale, untersucht wurden die in den Bakterien vorhandenen Menachinone, diese Chinone haben eine wichtige Funktion in der Atmungskette, ähnlich wie die Ubichinone in der Atmungskette beim Menschen. Bezüglich des Aufbaus der Bakterienzellwand wurden die polaren Lipide und Diaminosäuren untersucht, um den Typ der Peptidoglycane (Murein) zu charakterisieren. Weitere Untersuchungen umfassen den Abbau von Kohlenhydraten und anderen Substraten. Die folgende Tabelle gibt einen Überblick über die Untersuchungsergebnisse und damit über die Eigenschaften der drei Biovare wieder.[5]
| Merkmale | Biovar I M. luteus NCTC 2665 | Biovar II M. luteus DSM 14234 | Biovar III M. luteus DSM 14235 |
|---|---|---|---|
| Gemeinsamkeiten | Abbau von D-Glucose, D-Mannose und Saccharose; Hydrolyse von p-Nitrophenyl-, α-Glucopyranosid- und L-Alanin-p-Nitroanilid; (schwaches) Wachstum bei 45 °C, bei pH 10,0 und bei einem Anteil von 10 % Natriumchlorid. | ||
| pH-Wert | kein Wachstum bei pH 6,0 | Wachstum bei pH 6,0 variabel | kein Wachstum bei pH 6,0 |
| Urease | Urease-positiv | Urease-variabel | Urease-positiv |
| Verwertung von | Maltitol, L-Aspartat und Propionat | D-Maltose, D-Trehalose, Acetat, Propionat, DL-3-Hydroxybutyrat, DL-Lactat, Pyruvat, L-Histidin, L-Phenylalanin, L-Serin und Phenylacetat | D-Maltose, D-Trehalose, 4-Aminobutyrat, Fumarat, DL-3-Hydroxybutyrat, DL-Lactat, Oxoglutarat, Pyruvat, L-Histidin und L-Prolin |
| Peptidoglycan-Typ | A2 | A2 | A4α |
| Vorherrschendes Menachinon | MK-8 und MK-8(H2) | MK-8(H2) | MK-8(H2) |
Üblicherweise zeigen die Bakterienstämme einer Art größere Übereinstimmungen in phänotypischen Merkmalen. So deutliche Variationen innerhalb einer Art wurden bisher nicht beobachtet. Dabei muss aber bedacht werden, dass eine Organismus-Art keine statische Einheit darstellt, sondern sich – über einen langen Zeitraum betrachtet – verändert. Eine mögliche Deutung dieser Ergebnisse ist, dass sie nur einen „Schnappschuss“ im Laufe der Evolution zeigen.[5]
Etymologie
Der Gattungsname lässt sich auf das Aussehen der Zellen zurückführen (mikros aus dem Altgriechischen bedeutet „klein“, kokkos ist altgriechisch für „Beere“), der Artname verweist auf das Aussehen der Kolonien (luteus aus dem Lateinischen bedeutet „goldgelb“).[8]
Bedeutung

Das von M. luteus produzierte Carotinoid Sarcinaxanthin bietet das Potential wirtschaftlicher Nutzung. Carotinoide wirken als Antioxidantien („Radikalfänger“), die bereits zu diesem Zweck als Lebensmittelzusatzstoffe oder Nahrungsergänzungsmittel vermarktet werden. Auch ein Einsatz als Lebensmittelfarbstoff – ähnlich wie bei Beta-Carotin – ist denkbar.[13]
Von Bedeutung ist das Bakterium bereits als Indikator bei der Überprüfung von Antibiotika. M. luteus ist nicht nur gegenüber Lysozym empfindlich, sondern wird durch mehrere Antibiotika, z. B. Chloramphenicol, Erythromycin, Neomycin und Streptomycin im Wachstum gehemmt.[8] In einem sogenannten Antibiogramm wird bestimmt, ob ein Mikroorganismus (meist ein Krankheitserreger) gegenüber einem bestimmten Antibiotikum empfindlich reagiert oder gegen dieses resistent ist, nach dem Ergebnis richtet sich im Idealfall die Antibiotikatherapie. Umgekehrt kann man auch untersuchen, auf welche Mikroorganismen ein bestimmtes Antibiotikum wirkt, indem man ein Plättchen mit einer definierten Menge eines Antibiotikums in die Mitte des Nährmediums in einer Petrischale legt und verschiedene Bakterienarten radial dazu ausstreicht (siehe Abbildung). Wenn das Bakterium nicht empfindlich auf den Wirkstoff reagiert, wächst es bis an das Plättchen heran, im anderen Fall hemmt das in das Nährmedium diffundierte Antibiotikum sein Wachstum.[2] Bei einer Überprüfung eines Antibiotikums führt man M. luteus als Positivkontrolle mit, wenn bereits vorher bekannt ist, dass er empfindlich reagiert.
Eine in Zukunft ökologisch und ökonomisch interessante Anwendung von bestimmten M. luteus Stämmen liegt in ihrer Fähigkeit, als Komplexbildner Metallionen zu binden. Untersuchungen zeigen, dass dies bei Erzen, die Gold und Strontium in niedrigen Gehalten aufweisen, möglich ist, so dass eine Anreicherung der Metalle mit Hilfe des Bakteriums erfolgen kann.[35] Auch eine mikrobiologische Sanierung von kontaminierten Böden ist denkbar – durch den Stamm M. luteus str. modasa – der in der Lage ist, Kohlenwasserstoffe abzubauen und somit potenziell zur Entfernung von Erdölrückständen im Boden eingesetzt werden kann.[18]
Auch aus Sicht der Lebensmittelmikrobiologie ist M. luteus von Bedeutung. Da er nicht pathogen ist, gibt es keine direkten Grenz- oder Richtwerte. Allerdings kann sein Wachstum auf Lebensmitteln zu deren Verderb führen, da er sich von den organischen Inhaltsstoffen ernährt. Die Anwesenheit der Bakterien führt folglich zu einer Anreicherung von Stoffwechselprodukten im Lebensmittel und ebenso zum Verlust der Inhaltsstoffe. Hierbei sind die proteolytischen Enzyme von M. luteus von Bedeutung, mit denen er Proteine (Eiweiße) abbauen kann.[8] Dies führt bei zahlreichen proteinhaltigen Lebensmitteln (z. B. Fleischprodukten) zum Verderb, ohne dass es zu einer Lebensmittelvergiftung kommt.
Um dem vorzubeugen, sind hygienische Maßnahmen bei der Herstellung und Verpackung von Lebensmitteln anzuwenden, weiterhin werden Grenz- oder Richtwerte für Bakteriengruppen festgelegt. Dies ist insbesondere die „aerobe mesophile Koloniezahl“, darunter werden alle Bakterien aus dem Lebensmittel zusammengefasst, die mit Sauerstoff bei mittleren Temperaturen (20–40 °C) auf einem komplexen Nährmedium heranwachsen, also Kolonien bilden. Da M. luteus aerob und mesophil und außerdem sehr weit verbreitet ist, stellt er einen bedeutenden Vertreter in dieser Gruppe dar. Richtwerte für die aerobe mesophile Koloniezahl werden durch die Deutsche Gesellschaft für Hygiene und Mikrobiologie veröffentlicht, diese betragen beispielsweise:[36]
- 1 • 105 KbE (Koloniebildende Einheiten) pro Gramm bei Speiseeis für die lose Abgabe an den Verbraucher und bei Instantprodukten, die kalt angerührt werden (Mousse, Pudding, Desserts);
- 1 • 106 KbE pro Gramm bei Feinkostsalaten; feuchten, verpackten Teigwaren (z. B. Spätzle); Räucherlachs und Süßwasserfischen;
- 5 • 106 KbE pro Gramm bei Hackfleisch, rohem Schweinefleisch und rohem Geflügelfleisch.
Quellen
Literatur
- Michael T. Madigan, John M. Martinko, Jack Parker: Brock Mikrobiologie. Deutsche Übersetzung herausgegeben von Werner Goebel. 1. Auflage. Spektrum Akademischer Verlag, Heidelberg/ Berlin 2000, ISBN 3-8274-0566-1.
- Hans G. Schlegel, Christiane Zaborosch: Allgemeine Mikrobiologie. 7. Auflage. Thieme Verlag, Stuttgart/ New York 1992, ISBN 3-13-444607-3.
- Mikroorganismen im Unterricht. In: Horst Bayrhuber, Eckhard R. Lucius (Hrsg.): Handbuch der praktischen Mikrobiologie und Biotechnik. 1. Auflage. Band 3. Metzler-Schulbuchverlag, Hannover 1992, ISBN 3-8156-3351-6, S. 63–64.
- M. Wieser, E. B. Denner u. a.: Emended descriptions of the genus Micrococcus, Micrococcus luteus (Cohn 1872) and Micrococcus lylae (Kloos u. a. 1974). In: International journal of systematic and evolutionary microbiology. Band 52, Nr. 2, 2002, S. 629–637, doi:10.1099/ijs.0.01901-0 (PDF, 326kB [abgerufen am 23. März 2013]).
Einzelnachweise
- Michael T. Madigan, John M. Martinko, Jack Parker: Brock Mikrobiologie. Deutsche Übersetzung herausgegeben von Werner Goebel. 1. Auflage. Spektrum Akademischer Verlag GmbH, Heidelberg/Berlin 2000, ISBN 3-8274-0566-1.
- Hans G. Schlegel, Christiane Zaborosch: Allgemeine Mikrobiologie. 7. Auflage. Thieme Verlag, Stuttgart/ New York 1992, ISBN 3-13-444607-3.
- Taxonomy Browser Micrococcus luteus. In: Website National Center for Biotechnology Information (NCBI). Abgerufen am 27. Dezember 2019.
- Micrococcus luteus NCTC 2665. In: Website Genomes OnLine Database (GOLD). Abgerufen am 27. Dezember 2019.
- M. Wieser, E. B. Denner u. a.: Emended descriptions of the genus Micrococcus, Micrococcus luteus (Cohn 1872) and Micrococcus lylae (Kloos u. a. 1974). In: International journal of systematic and evolutionary microbiology. Band 52, Nummer 2, März 2002, S. 629–637, ISSN 1466-5026. PMID 11931177.
- E. Stackebrandt, C. Koch, O. Gvozdiak, P. Schumann: Taxonomic dissection of the genus Micrococcus: Kocuria gen. nov., Nesterenkonia gen. nov., Kytococcus gen. nov., Dermacoccus gen. nov., and Micrococcus Cohn 1872 gen. emend. In: International journal of systematic bacteriology. Band 45, Nummer 4, Oktober 1995, S. 682–692, ISSN 0020-7713. PMID 7547287.
- Micrococcus luteus str. modasa Genome sequencing. In: Website BioProject des National Center for Biotechnology Information (NCBI). Abgerufen am 27. Dezember 2019.
- Mikroorganismen im Unterricht. In: Horst Bayrhuber, Eckhard R. Lucius (Hrsg.): Handbuch der praktischen Mikrobiologie und Biotechnik. 1. Auflage. Band 3. Metzler-Schulbuchverlag, Hannover 1992, ISBN 3-8156-3351-6, S. 63–64.
- Joachim M. Wink: Micrococcus luteus DSM 20030. (PDF; 150 kB) In: Compendium of Actinobacteria from Dr. Joachim M. Wink, University of Braunschweig - abrufbar über die DSMZ. Abgerufen am 26. März 2013.
- A. Fleming: Lysozyme: President’s Address. In: Proceedings of the Royal Society of Medicine. Band 26, Nummer 2, Dezember 1932, S. 71–84, ISSN 0035-9157. PMID 19989057. PMC 2204285 (freier Volltext).
- A. Fleming: Lysozyme: a bacteriolytic ferment found normally in tissues and secretions. In: The Lancet. Band 213, Nummer 5501, 2. Februar 1929, S. 217–220, doi:10.1016/S0140-6736(00)97556-1.
- Catalogue of microorganisms (Katalog der Mikroorganismen). In: Website des Leibniz Institut DSMZ - Deutsche Sammlung von Mikroorganismen und Zellkulturen GmbH. Abgerufen am 27. Dezember 2019.
- R. Netzer, M. H. Stafsnes u. a.: Biosynthetic pathway for γ-cyclic sarcinaxanthin in Micrococcus luteus: heterologous expression and evidence for diverse and multiple catalytic functions of C(50) carotenoid cyclases. In: Journal of bacteriology. Band 192, Nummer 21, November 2010, S. 5688–5699, ISSN 1098-5530. doi:10.1128/JB.00724-10. PMID 20802040. PMC 2953688 (freier Volltext).
- A. Osawa, Y. Ishii u. a.: Characterization and antioxidative activities of rare C(50) carotenoids-sarcinaxanthin, sarcinaxanthin monoglucoside, and sarcinaxanthin diglucoside-obtained from Micrococcus yunnanensis. In: Journal of Oleo Science. Band 59, Nummer 12, Dezember 2010, S. 653–659, ISSN 1347-3352. PMID 21099143.
- M. Young, V. Artsatbanov u. a.: Genome sequence of the Fleming strain of Micrococcus luteus, a simple free-living actinobacterium. In: Journal of Bacteriology. Band 192, Nummer 3, Februar 2010, online-Veröffentlichung November 2009, S. 841–860, doi:10.1128/JB.01254-09, PMID 19948807, PMC 2812450 (freier Volltext).
- Micrococcus luteus. In: Website Genome des National Center for Biotechnology Information (NCBI). Abgerufen am 27. Dezember 2019.
- Micrococcus luteus SK58. In: Website Genomes OnLine Database (GOLD). Abgerufen am 27. Dezember 2019.
- A. Ghosh, S. A. Chaudhary u. a.: Whole-Genome Sequencing of Micrococcus luteus Strain Modasa, of Indian Origin. In: Genome announcements. Band 1, Nummer 2, 2013 Mar-Apr, S. e0007613, ISSN 2169-8287. doi:10.1128/genomeA.00076-13. PMID 23516205. PMC 3593314 (freier Volltext).
- Micrococcus luteus modasa. In: Website Genomes OnLine Database (GOLD). Abgerufen am 27. Dezember 2019.
- W. Liebl, W. E. Kloos, W. Ludwig: Plasmid-borne macrolide resistance in Micrococcus luteus. In: Microbiology. Band 148, Nummer 8, August 2002, S. 2479–2487, ISSN 1350-0872. PMID 12177341.
- TRBA (Technische Regeln für Biologische Arbeitsstoffe) 466: Einstufung von Prokaryonten (Bacteria und Archaea) in Risikogruppen. In: Website der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin (BAuA). 25. August 2015, S. 266, abgerufen am 27. Dezember 2019 (letzte Änderung vom 14. August 2019).
- K. J. Smith, R. Neafie, J. Yeager, H. G. Skelton: Micrococcus folliculitis in HIV-1 disease. In: The British journal of dermatology. Band 141, Nummer 3, September 1999, S. 558–561, ISSN 0007-0963. PMID 10583069.
- API® ID-Teststreifen. In: Website der bioMérieux Deutschland. Abgerufen am 27. Dezember 2019.
- Deutsche Sammlung von Mikroorganismen und Zellkulturen (DSMZ): Micrococcus luteus, Type Strain. In: Website BacDive. Abgerufen am 27. Dezember 2019.
- Micrococcus luteus SK58. In: Website BioProject des National Center for Biotechnology Information (NCBI). Abgerufen am 27. Dezember 2019.
- Micrococcus luteus MU201 genome sequence. In: Website BioProject des National Center for Biotechnology Information (NCBI). Abgerufen am 27. Dezember 2019.
- Daniel V. Lim: Microbiology. 2. Auflage. Mc Graw-Hill, Boston 1998, ISBN 0-697-26186-7, S. 614 f.
- Ferdinand Cohn: Untersuchungen über Bacterien. In: Beiträge zur Biologie der Pflanzen. Bd. 1, Nr. 2, 1872, S. 127–234, hier S. 153.
- Ferdinand Cohn: Untersuchungen über Bacterien. In: Beiträge zur Biologie der Pflanzen. Bd. 1, Nr. 2, 1872, S. 127–234.
- Chien-Hsun Huang, Chun-Lin Wang u. a.: Reclassification of Micrococcus aloeverae and Micrococcus yunnanensis as later heterotypic synonyms of Micrococcus luteus. In: International Journal of Systematic and Evolutionary Microbiology. Band 69, November 2019, S. 3512–3518, doi:10.1099/ijsem.0.003654.
- Micrococcus luteus MU201. In: Website Genomes OnLine Database (GOLD). Abgerufen am 27. Dezember 2019.
- Micrococcus luteus CD1_FAA_NB_1 Human Microbiome Project (HMP) reference genome. In: Website BioProject des National Center for Biotechnology Information (NCBI). Abgerufen am 27. Dezember 2019.
- Micrococcus luteus CD1_FAA_NB_1. In: Website Genomes OnLine Database (GOLD). Abgerufen am 27. Dezember 2019.
- Micrococcus luteus J28. In: Website Genomes OnLine Database (GOLD). Abgerufen am 27. Dezember 2019.
- Micrococcus luteus NCTC 2665 – Bioremediation strain capable of sequestering metals. In: Website BioProject des National Center for Biotechnology Information (NCBI). Abgerufen am 27. Dezember 2019.
- Fachgruppe Lebensmittelmikrobiologie und -hygiene, Arbeitsgruppe Mikrobiologische Richt- und Warnwerte der DGHM e.V.: Mikrobiologische Richt- und Warnwerte zur Beurteilung von Lebensmitteln (Stand Mai 2012). (Nicht mehr online verfügbar.) In: Website der Deutschen Gesellschaft für Hygiene und Mikrobiologie (DGHM). Archiviert vom Original am 11. Februar 2013; abgerufen am 24. März 2013.
Weblinks
- Jean Euzéby, Aidan C. Parte: Genus Micrococcus. In: List of Prokaryotic names with Standing in Nomenclature (LPSN). Abgerufen am 27. Dezember 2019.
- Eintrag zur Gattung Micrococcus. In: MicrobeWiki – Curated pages are reviewed and updated by microbiologists at Kenyon College. 6. August 2010, abgerufen am 26. März 2013.