Podoviridae
Die Virusfamilie Podoviridae umfasst Bakteriophagen der Ordnung Caudovirales mit linearem, doppelsträngigem DNA-Genom von 16 bis 70 kBp Länge, einem 50–70 nm im Durchmesser großen ikosaedrischen Kapsid mit einem kurzen, nicht-kontraktilen Schwanzteil von ca. 20 × 8 nm. Je nach Gattung befinden sich am Schwanzteil sechs kurze Schwanzfibern oder mehrere kurze Fortsätze (spikes). Dieses kurze Schwanzteil ist innerhalb der Ordnung Caudovirales charakteristisch für die Podoviridae, daher leitet sich der Name für die Familie vom griechischen podos (ποδός) für „Fuß“ ab.
| Podoviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|

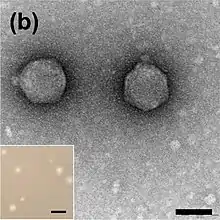
Typisches Aussehen eines Virusteilchens der Podoviridae | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Podoviridae | ||||||||||||||
| Links | ||||||||||||||
|
Die Gattungen innerhalb der Podoviridae unterscheiden sich hinsichtlich der Organisation des Genoms, den Mechanismen der DNA-Verpackung und dem Vorhandensein einer DNA-Polymerase. Die Spezies der Gattung Salasvirus (früher Phi29virus, Phi29likevirus, Φ29-ähnliche Viren) besitzen im Gegensatz zu den anderen ein langgestrecktes, nicht-isometrisches Kapsid. Sie wurde 2021 in die neue Familie Salasmaviridae der Caudoviricetes verschoben.
Im März 2021 wurde vorgeschlagen, die Familie mitsamt der Ordnung Caudovirales wegen fehlender Monophylie aufzulösen und durch neu zu schaffende Familien zu ersetzen, damit neue Ergebnisse aus der Metagenomik in die Taxonomie aufgenommen werden können.[3]
Systematik
Die Familie Podoviridae umfasst mit 11. Juni März 2021 (ICTV MSL #36) drei Unterfamilien sowie etliche weitere Spezies, die keiner Gattung (Genus) zugeordnet werden.[2] Die Klade der „crAss-like viruses“ („crAssphagen“) mit ausnahmsweise zirkulärem Genom[4][5] wird inzwischen vorschlagsmäßig als eigene Ordnung Crassvirales der Klasse Caudoviricetes unterstellt.
Familie Podoviridae
- Genus Flowerpowervirus
- Genus Immanueltrevirus
- Genus Manuelvirus
- Genus Akonivirus
- Genus Tinytimothyvirus
- Genus Diegovirus (veraltet Pocjvirus)
- Genus Oslovirus (veraltet Tl2011virus)
- Genus Traversvirus (veraltet Nona33virus)
- Spezies Escherichia-Virus 24B (en. Escherichia virus 24B)
- Spezies Escherichia-Virus 933W (en. Escherichia virus 933W, mit Enterobacteria-Phage 933W)[6][7]
- Spezies Escherichia-Virus Min27 (en. Escherichia virus Min27)
- Spezies Escherichia-Virus P27 (en. Escherichia virus P27, mit Escherichia-Phage P27)
- Spezies Escherichia-Virus PA28 (en. Escherichia virus PA28)
- Spezies Escherichia-Virus Stx2 II (en. Escherichia virus Stx2 II)
- Genus Anjalivirus
- Genus Astrithrvirus
- Genus Badaztecvirus
- Genus Bjornvirus
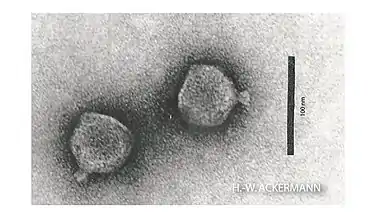
- Genus Bruynoghevirus (veraltet Luz24virus, Luz24likevirus, LUZ24-ähnliche Viren)
- Spezies Pseudomonas-Virus LUZ24 (en. Pseudomonas virus LUZ24, Typus)
- Genus Burrovirus
- Genus Chopinvirus
- Spezies Lactococcus-Virus KSY1 (en. Lactococcus virus KSY1)
- Genus Cimandefvirus
- Genus Delislevirus
- Spezies Mycoplasma-Virus P1 (en. Mycoplasma virus P1)
- Genus Dybvigvirus
- Spezies Actinomyces-Virus Av1 (en. Actinomyces virus Av1)
- Genus Enhodamvirus (veraltet Vp5virus)
- Genus Fipvunavirus
- Genus Firingavirus
- Genus Gervaisevirus
- Genus Giessenvirus
- Spezies Escherichia-Virus C1302 (en. Escherichia virus C1302)
- Genus Hollowayvirus (veraltet F116virus)
- Genus Jasminevirus
- Genus Kafunavirus (veraltet Kf1virus)
- Genus Kelquatrovirus
- Genus Kochitakasuvirus (veraltet Kpp25virus)
- Genus Kozyakovvirus
- Genus Krylovvirus
- Genus Kuravirus (veraltet Phieco32virus, Phieco32-ähnliche Viren)
- Spezies Escherichia-Virus 172-1 (en. Escherichia virus 172-1)
- Spezies Escherichia-Virus ECB2 (en. Escherichia virus ECB2)
- Spezies Escherichia-Virus NJ01 (en. Escherichia virus NJ01)
- Spezies Escherichia-Virus phiEco32 (en. Escherichia virus phiEco32, Typus)
- Spezies Escherichia-Virus eptima11 (en. Escherichia virus Septima11)
- Spezies Escherichia-Virus SU10 (en. Escherichia virus SU10)
- Spezies „Escherichia-Phage ES17“ (en. „Escherichia phage ES17“, vorgeschlagen)[8][9]
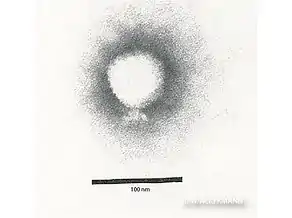
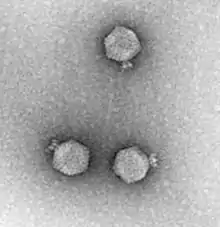
- Genus Lahexavirus
- Genus Lastavirus
- Genus Lederbergvirus (veraltet P22virus, P22likevirus, P22-ähnliche Viren)
- Spezies Escherichia-Virus HK620 (en. Escherichia virus HK620)
- Spezies Salmonella-Virus BTP1 (en. Salmonella virus BTP1)
- Spezies Salmonella-Virus P22 (en. Salmonella virus P22, Typus, mit Bakteriophage P22)
- Spezies Salmonella-Virus SE1Spa (en. Salmonella virus SE1Spa)
- Spezies Salmonella-Virus ST64T (en. Salmonella virus ST64T)
- Spezies Shigella-Virus Sf6 (en. Shigella virus Sf6)
- Spezies „Salmonella-Phage epsilon34“ (en. „Salmonella phage epsilon34“ mit Salmonella-Phage 34[10] alias Bakteriophage ε34[11][12])
- Genus Lessievirus (veraltet Bcep22virus)
- Genus Lightbulbvirus (veraltet Cba41virus)
- Genus Myxoctovirus
- Genus Pagevirus
- Genus Parlovirus
- Genus Perisivirus (veraltet Prtbvirus)
- Genus Privateervirus
- Genus Rauchvirus (veraltet Bpp1virus, Bpp1likevirus, BPP-1-ähnliche Viren)
- Genus Ryyoungvirus
- Genus Schmidvirus (veraltet Una961virus)
- Genus Sendosyvirus
- Spezies Hamiltonella-Virus APSE1 (en. Hamiltonella virus APSE1) – Wirt Hamiltonella defensa, Endosymbiont in Acyrthosiphon pisum (Erbsenlaus)
- Spezies Hamiltonella-Virus APSE2 (en. Hamiltonella virus APSE2) – Wirt Hamiltonella defensa, Endosymbiont in Acyrthosiphon pisum (Erbsenlaus)
- Genus Skarprettervirus
- Genus Sortsnevirus
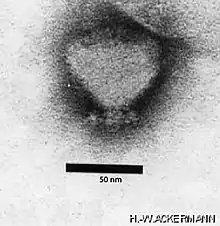
- Genus Uetakevirus (veraltet Epsilon15virus, Epsilon15likevirus, Epsilon15-ähnliche Viren)
- Species Salmonella-Virus Epsilon15 (en. Salmonella virus Epsilon15, Typus)[13]
- Genus Vicosavirus
- Genus Wumpquatrovirus
- Spezies Phormidium-Virus WMP4 (en. Phormidium virus WMP4)
- Genus Wumptrevirus
- Spezies Phormidium-Virus WMP3 (en. Phormidium virus WMP3)
- Genus Xuquatrovirus
- Spezies Escherichia virus PTXU04 (en. Escherichia virus PTXU04, Typus)

- Genus „Cyanopodovirus“ (informell: nicht zugeodnete Cyanophagen mit Podoviren-Morphologie)
- Spezies „Podophage BAC9D04“ (alias „Synechococcus podovirus BAC9D04“)[14][15][16]
- Spezies „Cyanophage Ma-LBP“ (alias „Microcystis-Phage Ma-LBP“)[17]
- Spezies „Cyanophage Ma-LEP“ (alias „Microcystis-Phage Ma-LEP“)[18]
- Spezies „Synechococcus podovirus MPP-A“[19]
- Spezies „Synechococcus podovirus MPP-B“[19]
- Genus nicht bestimmt:
- Spezies Pseudomonas-Virus 119X (en. Pseudomonas virus 119X)
Verschiebungen:
- Die Unterfamilie Autographivirinae wurde in den Rang einer Familie Autographiviridae erhoben,
und die Gattungen Aqualcavirus und Bifseptvirus in diese Familie verschoben.
- Die Gattung Nonanavirus wurde in die Familie Siphoviridae verschoben.
- Die Unterfamilie Picovirinae wurde in die neue Familie Salasmaviridae verschoben.
- Die Unterfamilie Rakietenvirinae wurde in die neue Familie Rountreeviridae verschoben.
- Weitere Gattungen und Spezies ohne nähere Zuordnung wurden in andere (insbes. neue) Familien verschoben.
Andere (vom ICTV bereits registrierte) Phagen aus der Familie Podoviridae wie Roseobacter-Virus SIO1 (mit Phage SIO1) und Vibrio-Virus VpV262 (mit Phage VpV262) sind zwar evolutionär mit den Autographiviridae verwandt, enthalten jedoch keine phagenkodierte RNA-Polymerase und zeigen auch größere Unterschiede auf der Ebene der Genomorganisation.[25][1]
Für die früher aufgrund ihrer Morphologie vorschlagsmäßig den Podoviridae zugeordneten crAssphagen wurde inzwischen eine eigene Ordnung „Crassvirales“ inniehalb der Klasse Caudoviricetes vorgeschlagen.[3]
Neben den crAssphagen wurden in der menschlichen Darmflora noch eine weitere Klade gefunden, die „Gubaphagen“ (englisch gut bacteriophages, „Gubaphage clade)“ (mit zwei Gattungen, „G1“ – infiziert Bacteroides und „G2“ – infiziert Parabacteroides [en]), die nach den crAssphagen dort die zweithäufigsten Viren (d. h. Bakteriophagen) darstellen. Die Merkmale der Gubaphagen erinnern dabei an die von „p-crAssphage“.[26][27] Aufgrund dieser Ähnlichkeiten war zunächst ebenfalls eine Zugehörigkeit zu den Podoviridae anzunehmen, dem Vorschlag der neuen Ordnung „Crassvirales“ wird dagegen eine Nähe oder gar Zugehörigkeit zu dieser wahrscheinlich.
Literatur
- C. M. Fauquet, M. A. Mayo et al.: Eighth Report of the International Committee on Taxonomy of Viruses. London, San Diego, 2004.
- David M. Knipe, Peter M. Howley et al. (Hrsg.): Fields’ Virology. 4. Auflage, Philadelphia 2001.
Einzelnachweise
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- ICTV: ICTV Master Species List 2020.v1, New MSL including all taxa updates since the 2019 release, March 2021 (MSL #36)
- Dann Turner, Andrew M. Kropinski, Evelien M. Adriaenssens: A Roadmap for Genome-Based Phage Taxonomy, in: MDPI Viruses Band 13, Nr. 3, Section Bacterial Viruses, 18. März 2021, 506, doi:10.3390/v13030506
- NCBI: crAss-like viruses/phages (clade)
- SIB: crAss-like phages, auf: Expasy ViralZone – Order: Caudovirales, Estimated about 10 genera
- NCBI: Escherichia virus 933W (species)
- SIB: Viral exotoxin
- NCBI: Escherichia phage ES17 (species)
- Sabrina I. Green, Carmen Gu Liu, Xue Yu, Shelley Gibson, Wilhem Salmen, Anubama Rajan, Hannah E. Carter, Justin R. Clark, Xuezheng Song, Robert F. Ramig, Barbara W. Trautner, Heidi B. Kaplan, Anthony W. Maresso: Targeting of Mammalian Glycans Enhances Phage Predation in the Gastrointestinal Tract, in: mBio vom 9. Februar 2021, doi:10.1128/mBio.03474-20. Dazu:
- Antibiotic Game-Changer: Phages Can Anticipate Bacteria’s Location and Destroy Them Before They Cause an Infection, auf: SciTechDaily vom 9. Februar 2021. Quelle: TAILΦR labs, Baylor College of Medicine
- NCBI: Salmonella phage 34 (no rank)
- Robert Villafane, Milka Zayas, Eddie B. Gilcrease, Andrew M. Kropinski, Sherwood R. Casjens: Genomic analysis of bacteriophage ε34 of Salmonella enterica serovar Anatum (15+), in: BMC Microbiol. 8, S. 227, 17. Dezember 2008, doi:10.1186/1471-2180-8-227, PMC 2629481 (freier Volltext), PMID 19091116
- Harald Brüssow, Carlos Canchaya, Wolf-Dietrich Hard: Phages and the Evolution of Bacterial Pathogens: from Genomic Rearrangements to Lysogenic Conversion, in: Microbiol Mol Biol Rev. 68(3), September 2004, S. 560–602, doi:10.1128/MMBR.68.3.560-602.2004, PMC 515249 (freier Volltext), PMID 15353570, siehe insbes. Table 1
- Preeti Gipson, Matthew L. Baker, Desislava Raytcheva, Cameron Haase-Pettingell, Jacqueline Piret, Jonathan A. King, Wah Chiu: Protruding knob-like proteins violate local symmetries in an icosahedral marine virus. In: Nature Communications. 5, Nr. 4278, 2. Juli 2014. doi:10.1038/ncomms5278., Corrigendum, in: Nature Communications, Band 6, Nr. 6040, 12. Januar 2015, doi:10.1038/ncomms7040.
- Itai Sharon et al.: Viral photosynthetic reaction center genes and transcripts in the marine environment, in: The ISME Journal Band 1, S. 492–501, Oktober 2007, doi:10.1038/ismej.2007.67
- Ruth-Anne Sandaa et al.: Photosynthetic genes in viral populations with a large genomic size range from Norwegian coastal waters, in: FEMS Microbiology Ecology, Band 63, Nr. 1, 1. Januar 2008, S. 2–11, doi:10.1111/j.1574-6941.2007.00400.x
- John H. Paul, Matthew B. Sullivan: Marine phage genomics: what have we learned?, Curr. Op. in Biotechnology Band 16, Nr. 3, Juni 2005, S. 299-307, doi:10.1016/j.copbio.2005.03.007, PMID 15961031, EuroPMC 15961031, siehe Fig. 3 (Genomkarte)
- Stephen Tucker, Peter Pollard: Identification of Cyanophage Ma-LBP and Infection of the Cyanobacterium Microcystis aeruginosa from an Australian Subtropical Lake by the Virus, in: ASM Applied and Environmental Microbiology, Band 71, Nr. 2, 3. Februar 2005, S. 629-635, doi:10.1128/AEM.71.2.629-635.2005
- NCBI: Cyanophage Ma-LEP, equivalent: Microcystis phage Ma-LEP (species)
- Sijun Huang, Si Zhang, Nianzhi Jiao, Feng Chen: Comparative Genomic and Phylogenomic Analyses Reveal a Conserved Core Genome Shared by Estuarine and Oceanic Cyanopodoviruses, in: PLOS ONE, 16. November 2015, doi:10.1371/journal.pone.0142962
- NCBI: Ochrobactrum phage vB_OspP_OH (species)
- Przemyslaw Decewicz, Piotr Golec, Mateusz Szymczak, Monika Radlinska, Lukasz Dziewit: Identification and Characterization of the First Virulent Phages, Including a Novel Jumbo Virus, Infecting Ochrobactrum spp., in: MDPI Int. J. Mol. Sci, Band 21, Nr. 6, Special Issue Bacteriophage—Molecular Studies, 18. März 2020, 2096; doi.org/10.3390/ijms21062096
- NCBI: Puniceispirillum phage HMO-2011 (species)
- Ilnam Kang, Hyun-Myung Oh, Dongmin Kang, Jang-Cheon Cho: Genome of a SAR116 bacteriophage shows the prevalence of this phage type in the oceans, in: PNAS 110 (30), 23. Juli 2013, S. 12343–12348, doi:10.1073/pnas.1219930110
- Hyun-Myung Oh, Kae Kyoung Kwon, Ilnam Kang, Sung Gyun Kang, Jung-Hyun Lee, Sang-Jin Kim, Jang-Cheon Cho: Complete Genome Sequence of “Candidatus Puniceispirillum marinum” IMCC1322, a Representative of the SAR116 Clade in the Alphaproteobacteria, in: Journal of Bacteriology, 26. Mai 2010, doi:10.1128/JB.00347-10, PMID 20382761
- R. Lavigne, D. Seto, P. Mahadevan, H.-W. Ackermann, A. M. Kropinski: Unifying classical and molecular taxonomic classification: analysis of the Podoviridae using BLASTP-based tools. In: Research in Microbiology. Band 159, Nr. 5, 2008, S. 406–414, doi:10.1016/j.resmic.2008.03.005, PMID 18555669.
- Luis Fernando Camarillo-Guerrero, Alexandre Almeida, Guillermo Rangel-Pineros, Robert D. Finn, Trevor D. Lawley: Massive expansion of human gut bacteriophage diversity, in: Cell Resource Band 184, Nr. 4, S. 1098-1109.e9, 18. Februar 2021, doi:10.1016/j.cell.2021.01.029. PrePrint vom 3. September 2020: bioRxiv, Europe PMC, doi:10.1101/2020.09.03.280214. Dazu:
- Martin Vieweg: Tausende Virenarten der Darmflora entdeckt, auf: wissenschaft.de vom 18. Februar 2021 (deutsch)
- Daniel Lingenhöhl: Virobiom: Darm beherbergt zehntausende unbekannte Virenarten, auf: spektrum.de vom 19. Februar 2021 (deutsch)
- Biologists Find Almost 143,000 Bacteriophage Species in Human Gut, auf: sci-news vom 19. Februar 2021 (englisch)
- Peter Dockrill: Scientists Find 140,000 Virus Species in The Human Gut, And Most Are Unknown, auf: sciencealert vom 28. Februar 2021 (englisch)
- Scientists identify more than 140,000 virus species in the human gut, auf: ScienceDaily vom 18. Februar 2021 (englisch)
- Luis Fernando Camarillo Guerrero: Integrative Analysis of the Human Gut Phageome Using a Metagenomics Approach, Doktorarbeit, Gonville & Caius College, University of Cambridge, August 2020, doi:10.17863/CAM.63973