Molekülmarkierung
Die Molekülmarkierung (englisch labeling, labelling, tagging) ist eine Methode der Chemie und Biochemie zur selektiven Bindung eines Atoms oder Moleküls an ein Molekül. Die Markierung wird zur weiteren Verfolgung des markierten Moleküls oder zur Aufklärung eines Reaktionsmechanismus (englisch crossover experiment) verwendet.
Eigenschaften
Moleküle können je nach ihren charakteristischen funktionellen Gruppen mit einer selektiv bindenden Markierung versehen werden, die ein nachverfolgbares Signalmolekül (Reportermolekül) trägt. Die Markierung besteht aus einer bindenden Gruppe (Kopplungsgruppe), gelegentlich einem Verbinder (Linker) und einem nachweisbaren Molekül (Reporter). Die Markierung ist dabei über die Kopplungsgruppe meistens kovalent verbunden. Bei Nukliden kann sie aber auch ionisch und bei indirekten Nachweisen über eine Mischung aus ionischen Bindungen, Van-der-Waals-Bindungen, hydrophoben Effekten, Wasserstoffbrücken und teilweise auch Disulfidbrücken gebunden sein. Im Gegensatz zu einer Färbung mit den meisten selektiv bindenden Proteinfarbstoffen oder Nukleinsäurefarbstoffen erfolgt die kovalente Markierung von Biomolekülen entweder an einzelnen Atomen oder sequenzspezifisch.
Reportertypen
Die Reportermoleküle werden meist über selektiv reaktive Kopplungsgruppen kovalent mit einem flexiblen Abstandshalter (englisch linker ‚Verbinder‘) an bestimmte funktionelle Gruppen des zu markierenden Moleküls gekoppelt. Einen Sonderfall bilden die Nuklide, die aufgrund ihrer vergleichsweise geringen Molekülgröße auch direkt an ein anderes Molekül gekoppelt werden können (ohne reaktive Kopplungsgruppe oder linker). Während zur Markierung von kleinen Molekülen meistens eine Isotopenmarkierung verwendet wird, wurden für die Markierung der verschiedenen Biopolymere wie Proteine und DNA viele unterschiedliche Reportertypen entwickelt.
Als Reportermoleküle bei Markierungen werden Nuklide (radioaktive und nicht radioaktive), Biotin, Reporterenzyme, Oligonukleotide, Fluorophore (im Zuge einer Fluoreszenzmarkierung) oder bei Proteinen auch Protein-Tags eingesetzt.[1][2][3][4]
Kopplungsarten
Die Markierungen mit Nukliden besitzen unter den Markierungen die kleinsten Änderungen der Molmasse und führen daher zu einer vergleichsweise geringeren Änderung der Proteinstruktur und der biologischen Aktivität, die radioaktiven Nuklide sind jedoch von erhöhten Sicherheitsmaßnahmen und Anforderungen an das Sicherheitslabor begleitet. Nuklide (Isotope und Isobare) können direkt kovalent an das zu markierende Molekül gekoppelt werden (chemische Kopplung), daneben auch kovalent über eine Kopplungsgruppe angefügt werden, ionisch gekoppelt werden (z. B. über Chelatoren wie DTPA, TTHA oder chelierende Peptide wie das Protein-Tag His-Tag)[5][6][7] durch eine chemische Totalsynthese (z. B. per Peptidsynthese, per Phosphoramidit-Synthese) oder in einer Biosynthese durch den Umbau Nuklid-markierter Vorläufersubstanzen erzeugt werden (metabolische Markierung). Im Zuge einer chemischen Isotopenmarkierung oder einer metabolischen Isotopenmarkierung werden Moleküle mit radioaktiven Isotopen (z. B. 3H,[8] 11C,[9] 13C,[10] 14C,[11] 13N,[12] 15O,[12] 18F,[12] 26Al,[11] 32P,[13] 33P,[13] 35S,[14] 36Cl,[11] 41Ca,[11] 125I,[15] 131I[16]) markiert. Die Isotope von nicht natürlicherweise in Biomolekülen vorkommenden Elementen wie Iod, 99Technetium oder 113Indium müssen bei einer metabolischen Markierung zuvor chemisch an die Vorläufermoleküle gekoppelt werden.[17][18]
Die anderen Arten der Reportermoleküle können kovalent über eine Kopplungsgruppe angefügt werden, durch eine chemische Totalsynthese oder in einer Biosynthese erzeugt werden. Die Biosynthese erfolgt im Stoffwechsel durch den Umbau Reporter-markierter Vorläufersubstanzen (metabolische Markierung). Eine Markierung kann bei hoher Selektivität und geringer Toxizität auch teilweise metabolisch und teilweise synthetisch erfolgen, wie bei der bioorthogonalen Markierung.[19][20][21] Dabei werden metabolisch eingebaute Vorläufersubstanzen nach einer Proteinreinigung bzw. DNA-Reinigung anschließend zur nachträglichen chemischen Kupplung eines Reportermoleküls anhand der selektiv reaktiven Gruppe verwendet, z. B. per Click-Chemie[22][23] (1,3-dipolare Cycloaddition mit Aziden und Cyclooctynen, eine Kupfer-freie Click-Chemie),[24] per Cycloaddition zwischen Nitronen und Cyclooctynen,[25] per Oxim/Hydrazon-Bildung aus Aldehyden oder Ketonen,[26] per Tetrazin-Ligation,[27] per Isonitril-basierter Click-Reaktion,[28] per Quadricyclan-Ligation[29] und per Staudinger-Reaktion zwischen Aziden und Triarylphosphinen.[30][31][32]
Zur Verfolgung des zeitlichen Verlaufs eines markierten Moleküls im Kontext des Stoffwechsels wird als Variante der metabolischen Markierung die Puls-Markierung (englisch pulse labeling) verwendet. Bei der Puls-Markierung wird der Markierungszeitraum zeitlich begrenzt, wodurch die Markierung des Moleküls und seiner Metaboliten genauer eingrenzbar ist und der Wechsel der Markierung auf einzelne nachfolgende Stoffe in einem Stoffwechselweg beobachtet werden kann. Die Verfolgung mehrerer Metabolite gleichzeitig wird auch als metabolische Fluxanalyse (englisch metabolic flux analysis) bezeichnet.[33][34][35][36] Durch die Molekülmarkierung können durch eine Markierung von Oberflächenproteinen und/oder der Glykokalyx auch die Zellmembranen von ganzen Zellen markiert werden, z. B. für die Durchflusszytometrie, die Fluoreszenzmikroskopie oder die Fluoreszenztomographie.
Bei einem Protection Assay werden unter anderem Markierungen verwendet, um unbedeckte Bereiche auf der Oberfläche eines Moleküls zu markieren, wodurch die Oberfläche oder die Bindungsstelle eines bindenden Moleküls bestimmt werden kann. Die für die Markierung zugänglichen Bereiche eines Proteins sind ein Hinweis, dass die markierten Aminosäuren sich frei zugänglich auf der Proteinoberfläche befinden. Gefaltete Bereiche von Proteinen und Bereiche, die ein anderes Molekül gebunden haben, sind für eine Markierung weniger zugänglich.
Direkte und indirekte Nachweise
Bei einer Molekülmarkierung kann das nachzuweisende Molekül entweder direkt mit einem Reportermolekül versehen werden oder indirekt durch selektiv bindende Reporter-tragende Moleküle nachgewiesen werden. Methoden zur Molekülmarkierung sind:
- Direkte Molekülmarkierungen
- chemische Totalsynthese (nur in vitro)
- chemische Kopplung (in vitro Kupplung des Reportermoleküls)
- bioorthogonale Markierung (in vitro Kupplung des Reportermoleküls)
- metabolische Markierung in vitro (z. B. PCR, In-vitro-Translation)
- metabolische Markierung in vivo (Fütterung markierter Moleküle)
- Reporterproteine
- Protein-Tags bzw. RNA-Tags, die ein Reportermolekül binden.
- Indirekte Molekülmarkierungen
- Immunmarkierung (bei Proteinen, Protein-Tags und Kohlenhydraten)
- Lektinmarkierung (bei Kohlenhydraten und Glykoproteinen)
- Hybridisierungssonden (bei Nukleinsäuren)
- Biotinylierung
- Reportergene
Proteinmarkierung
Reaktive Gruppen in Proteinen
Im Gegensatz zu Kohlenhydraten und Nukleinsäuren kommen unter den Biomolekülen manche Strukturmotive aufgrund der verschiedenen enthaltenen Aminosäuren nur bei Proteinen vor, z. B. Sulfhydryl-enthaltende Cysteine oder Phenolreste in Tyrosinen. Diese Strukturmotive können mit entsprechenden Kopplungsreagenzien selektiv markiert werden. Dabei werden Strategien untersucht, nur eine bestimmte von mehreren Aminosäuren gleicher Sorte zu markieren.[37]
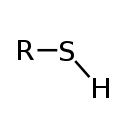 Thiol- oder Sulfhydrylgruppe an Cystein
Thiol- oder Sulfhydrylgruppe an Cystein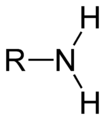 Aminogruppe an Lysin und am N-Terminus
Aminogruppe an Lysin und am N-Terminus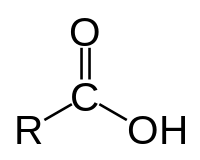 Carboxygruppe an Asparaginsäure, Glutaminsäure und am C-Terminus
Carboxygruppe an Asparaginsäure, Glutaminsäure und am C-Terminus
Kupplung
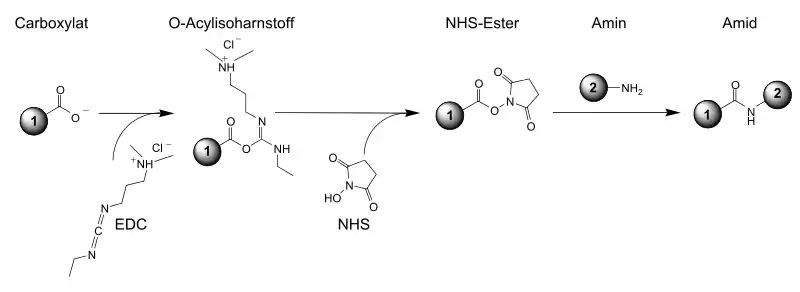
Cysteine können mit Maleimiden,[38][39] Disulfiden, Iodacetamid (z. B. IAEDANS), Haloacetylen, Aziridinen, Acryloylen, Arylierungsmitteln, Vinylsulfonen, Pyridyl- und anderen Disulfiden selektiv reagieren.[40][41] Aminosäuren mit primären Aminen an der Seitenkette wie Lysin können durch Succinimidester (N-Hydroxysuccinimid, Sulfosuccinimid- oder andere Succinimidylester) oder bestimmte Isothiocyanate wie PITC oder FITC markiert werden.[8] Carboxygruppen können durch eine Aktivierung mit Carbodiimiden an Amingruppen gekoppelt werden.[42] Nach einer Oxidation von Proteinen oder durch eine reduktive Alkylierung können verschiedene Reportermoleküle gekoppelt werden.[43][44] Über eine Tosylierung können nukleophile Gruppen in Proteinen gekoppelt werden.[45] Mit Enzymen können Proteine oftmals mit einer erhöhten Selektivität an Protein-Tags markiert werden, z. B. per Sortase,[46] per Transglutaminase,[47] per Haloalkandehalogenase oder per Phosphopantetheinyltransferase.[48][49] Auf dem gleichen Prinzip wie die Markierung durch Kupplung basiert auch die Vernetzung und die Fixierung verschiedener Aminosäureseitenketten in Proteinen.
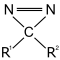
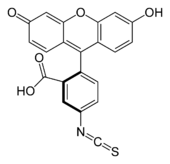
Auch photoreaktive Moleküle (z. B. Arylazide, Diazirine) können als reaktive Gruppe einer Markierung bei Proteinen verwendet werden, um den Zeitpunkt der Kupplung besser steuern zu können, da die Reaktion erst mit UV-Bestrahlung ausgelöst wird, z. B. im Zuge einer Photoaffinitätsmarkierung. Aufgrund der geringeren Selektivität dieser radikalisch reagierenden Kopplungsgruppen wird oftmals die Funktionsfähigkeit des Proteins durch Reaktion der radikalischen Kopplungsgruppe mit wichtigen Funktionen (z. B. ein aktives Zentrum eines Enzyms oder eine Bindungsstelle) gemindert.[50] Ein Vorteil radikalischer Kopplungen ist dagegen die Unabhängigkeit vom Vorkommen bestimmter Aminosäuren im zu koppelnden Protein. Daher werden photoreaktive Markierungen meistens eingesetzt, wenn keine oder nur eine Amin- oder Sulfhydrylgruppe zur selektiven Kupplung zur Verfügung steht oder eine anschließende Funktionsfähigkeit unerheblich ist. Es existieren auch photoreaktive Diazirin- oder Azid-enthaltende Analoga der Aminosäuren Leucin (Photo-Leucin), Methionin und p-Benzoyl-Phenylalanin, die während der Translation in das Protein eingebaut werden können.[51][52] Durch eine Peptidsynthese oder eine In-vitro-Translation können Peptide mit markierten Aminosäurederivaten hergestellt werden.[53][54]
Einige Proteaseinhibitoren führen zu einer selektiven Markierung von Proteinen. Bei einem Label-Transfer-Experiment wird eine spaltbare Quervernetzung mit einem Reporter zwischen zwei benachbarten Molekülen verwendet, um durch eine Spaltung der Quervernetzung das Reportermolekül zwischen diesen Molekülen zu übertragen und dadurch deren Nachbarschaft nachzuweisen.[55][56][57] Der dabei verwendete spaltbare Vernetzer enthält die Reportergruppe zwischen der Spaltstelle des Vernetzers und der Kopplungsgruppe für das meist unbekannte bindende Zielmolekül. In der DIGE werden unterschiedlich markierte Proben gemeinsam per SDS-PAGE oder 2D-Gelelektrophorese aufgetrennt.[58][59][60] In einem Proximity Ligation Assay wird die Nachbarschaft Oligonukleotid-markierter Proteine per PCR nachgewiesen.[61] Proteine können mit Oligonukleotiden für einen Nachweis per Hybridisierung markiert werden.[62] Auch können Proteine mit Biotin-Succinimidylestern in vitro biotinyliert oder in vivo mit einem Protein-Tag mit Biotinylierung (z. B. Avi-Tag, BCCP-Tag, Strep-Tag) versehen werden und anschließend indirekt mit Avidin- oder Streptavidin-Konjugaten markiert werden.[63]
Nuklidmarkierungen
Die Nuklidmarkierung verwendet meistens radioaktive Nuklide aufgrund der vergleichsweise hohen Sensitivität (geringe Nachweisgrenze) und der Einfachheit des Nachweises per Autoradiographie, per Szintillationszähler oder per Positronen-Emissions-Tomographie.[64][65] In der Kernspinresonanzspektroskopie und der Massenspektrometrie können auch nicht-radioaktive Nuklide verwendet werden.
Die Nuklidmarkierung erfolgt entweder chemisch oder biosynthetisch. Die biosynthetische Markierung erfolgt z. B. als metabolische Markierung durch Fütterung von Zellkulturen oder Versuchstieren mit markierten Vorläufermolekülen.[66] Durch eine Radioiodierung können Tyrosine in vitro mit radioaktivem Iod markiert werden.[67] Phosphorylierungen von Serin, Threonin und Tyrosin werden meist enzymatisch mit geeigneten Proteinkinasen und radioaktivem Phosphor-haltigem Adenosintriphosphat durchgeführt.[68] Eine radioaktiv markierte Prenylierung kann mit 3H-Mevalonsäure durchgeführt werden.[69] Daneben wird im Zuge einer Prenylierung der C-Terminus methyliert, der durch 3H-markiertes S-Adenosyl-Methionin reversibel radioaktiv markiert werden kann.[69] Durch eine Myristylierung kann N-terminal markiert werden.[70] Eine Geranylgeranylierung kann mit Azidogeranylen durchgeführt werden.[71] Sulfatierungen können mit 35S-markiertem Sulfat nachgewiesen werden.[69] Wasserstoffatome können durch eine Deuterierung mit Deuterium ausgetauscht werden. Die Markierung mit radioaktiven Isotopen erlaubt eine Verfolgung per Autoradiographie. In der Massenspektrometrie wird die Isobarenmarkierung zur Unterscheidung verschiedener Proben verwendet.[72][73] Neben den üblicherweise verwendeten Nukliden 1Wasserstoff oder 15Stickstoff werden in der NMR-Spektroskopie Markierungen mit 13Kohlenstoff oder 19Fluor verwendet.[74][75][76]
Bioorthogonale Markierungen
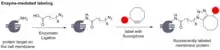
Proteine können bioorthogonal markiert werden. Hierbei werden verschiedene selektive Kopplungsreaktionen verwendet, z. B. per Sonogashira-Kupplung,[77] per kupferkatalysierter Alkin-Azid-Cycloaddition,[78][79][80] per Heck-Reaktion,[81] per Oxim-Ligation,[82] per Cyclopropen-Azid-Kopplung,[83] per Myristylierung,[70] per Cyanobenzothiazol-Kondensation oder per Tetrazol-alken Cycloaddition.[84][85][86][87] Membranproteine können nach der Biosynthese enzymatisch mit Aziden gekoppelt werden, die wiederum per Staudinger-Reaktion mit Reportermolekülen gekoppelt werden.[88]
Rekombinante Markierungen
Proteine können als rekombinante Proteine mit einem Protein-Tag oder mit einem Reporterprotein als Fusionsprotein hergestellt werden, die während der Translation N-terminal oder C-terminal an das Protein angehängt werden. Über das in das rekombinante Protein eingefügte Protein-Tag kann ein Protein aufgereinigt oder nachgewiesen werden, z. B. mit GFP oder mit Reporterenzymen. Manche Protein-Tags werden nach der Biosynthese nachträglich bioorthogonal mit einem Reportermolekül gekoppelt,[89] z. B. das Snap-Tag,[90][91][92] das Polyhistidin-Tag,[93] das Flash-Tag,[94] das ReAsH-Tag,[95] das Flag-Tag,[96] das Clip-Tag, das HyRe-Tag,[97] das beta-Lactamase-Tag,[98] das LAP-Tag und das Sortase-Tag.[99][46] Durch Inteine können Proteine posttranslational markiert werden.[100][101][102]
Indirekte Markierungen
Bei einer Immunmarkierung mit Immunkonjugaten basiert die Selektivität der Markierung auf der Bindung von Antikörpern, daneben ist der Antikörper meist selbst oder über einen Sekundärantikörper mit einem Reportermolekül markiert, der indirekt zum Nachweis dient, z. B. bei der Fluoreszenzmikroskopie, beim Western Blot und beim ELISA. Das vom Antikörper gebundene Epitop ist dabei entweder im Protein oder an einem Protein-Tag. Proteine können auch durch Reportergene indirekt nachgewiesen werden.
Nukleinsäuremarkierung
Reaktive Gruppen in Nukleinsäuren
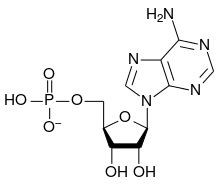
DNA besitzt im Vergleich zu Proteinen eine geringere Anzahl verfügbarer funktioneller Gruppen aufgrund der verwendeten Nukleotide, darunter eine zur Kupplung verwendbare Aminogruppe in der Nukleinbase Adenin.
Kopplungsarten

Die Phosphatgruppe kann durch radioaktives Phosphat mit einem 32Phosphoratom in vitro im Zuge eines Random Priming, einer Nick translation, einer Erzeugung per Phosphoramidit-Synthese oder in vivo durch Biosynthese mit radioaktivem Phosphat markiert werden. Auch DNA kann bioorthogonal markiert werden, z. B. mit 5-Ethynyl-2'dUTP.[23] Die Aminogruppe des Adenins kann in vitro mit Succinimidylestern oder Isothiocyanaten reagieren.
Durch eine Polymerasekettenreaktion kann DNA in vitro mit unnatürlichen Nukleotidanaloga markiert werden, z. B. BrdU, Digoxigenin-dUTP, Hydroxymethyl-dCTP sowie fluoreszente Nukleotide in der DNA-Sequenzierung nach Sanger, der QPCR oder der In-situ-Hybridisierung mit Hybridisierungssonden.[103][104][105][106]
RNA
RNA kann unter anderem biosynthetisch mit RNA-Tags, chemisch oder mit Hybridisierungssonden markiert werden.[107][108][109][110][111] Bei RNA-Tags werden meist DNA-Sequenzen von Aptameren in das offene Leseraster eines Gens kloniert. Nach einer Erzeugung der RNA mit dem RNA-Tag durch Transkription bilden sich Sekundärstrukturen, die z. B. selektiv an Dextran, Streptavidin oder Farbstoffe binden.[112][113][114]
Kohlenhydratmarkierung
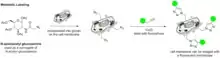
Markierte Kohlenhydrate werden durch metabolische oder bioorthogonale Markierung erzeugt, chemisch markiert oder die Kohlenhydrate werden indirekt über Lektine nachgewiesen.[115][116]
Literatur
- Hubert Rehm: Proteinbiochemie / Proteomics, Der Experimentator. 5. Auflage. Spektrum Akademischer Verlag, Heidelberg 2006, ISBN 3-8274-1726-0.
- Friedrich Lottspeich, Haralabos Zorbas: Bioanalytik. Spektrum Akademischer Verlag, Heidelberg 1998, ISBN 3-8274-0041-4.
- Juan S. Bonifacino: Protein Labeling and Immunoprecipitation. In: Current Protocols in Cell Biology. Wiley-VCH 1998. doi:10.1002/0471143030.cb0700s15. PDF.
Einzelnachweise
- M. J. Hinner, K. Johnsson: How to obtain labeled proteins and what to do with them. In: Current Opinion in Biotechnology. Band 21, Nummer 6, Dezember 2010, S. 766–776, doi:10.1016/j.copbio.2010.09.011. PMID 21030243.
- R. Wombacher, V. W. Cornish: Chemical tags: applications in live cell fluorescence imaging. In: Journal of biophotonics. Band 4, Nummer 6, Juni 2011, S. 391–402, doi:10.1002/jbio.201100018. PMID 21567974.
- H. M. O’Hare, K. Johnsson, A. Gautier: Chemical probes shed light on protein function. In: Current opinion in structural biology. Band 17, Nummer 4, August 2007, S. 488–494, doi:10.1016/j.sbi.2007.07.005. PMID 17851069.
- L. W. Miller, V. W. Cornish: Selective chemical labeling of proteins in living cells. In: Current opinion in chemical biology. Band 9, Nummer 1, Februar 2005, S. 56–61, doi:10.1016/j.cbpa.2004.12.007. PMID 15701454.
- D. R. Reddy, L. E. Pedró Rosa, L. W. Miller: Luminescent trimethoprim-polyaminocarboxylate lanthanide complex conjugates for selective protein labeling and time-resolved bioassays. In: Bioconjugate Chemistry. Band 22, Nummer 7, Juli 2011, S. 1402–1409, doi:10.1021/bc200131k. PMID 21619068. PMC 3140616 (freier Volltext).
- N. Maindron, S. Poupart, M. Hamon, J. B. Langlois, N. Plé, L. Jean, A. Romieu, P. Y. Renard: Synthesis and luminescence properties of new red-shifted absorption lanthanide(III) chelates suitable for peptide and protein labelling. In: Organic & biomolecular chemistry. Band 9, Nummer 7, April 2011, S. 2357–2370, doi:10.1039/c0ob00832j. PMID 21321764.
- G. Licini, P. Scrimin: Metal-ion-binding peptides: from catalysis to protein tagging. In: Angewandte Chemie. Band 42, Nummer 38, Oktober 2003, S. 4572–4575, doi:10.1002/anie.200301668. PMID 14533147.
- G. H. Müller: Protein labelling with 3H-NSP (N-succinimidyl-[2,3-3H]propionate). In: Journal of cell science. Band 43, Juni 1980, S. 319–328, PMID 7419623.
- B. Långström, F. Karimi, Y. Watanabe: Endogenous compounds labeled with radionuclides of short half-life-some perspectives. In: Journal of Labelled Compounds and Radiopharmaceuticals. Band 56, Nummer 3–4, 2013 Mar-Apr, S. 251–262, doi:10.1002/jlcr.3033. PMID 24285332.
- G. van Hall: Correction factors for 13C-labelled substrate oxidation at whole-body and muscle level. In: The Proceedings of the Nutrition Society. Band 58, Nummer 4, November 1999, S. 979–986, PMID 10817166.
- M. Palmblad, B. A. Buchholz, D. J. Hillegonds, J. S. Vogel: Neuroscience and accelerator mass spectrometry. In: Journal of Mass Spectrometry. Band 40, Nummer 2, Februar 2005, S. 154–159, doi:10.1002/jms.734. PMID 15706618.
- P. W. Miller, N. J. Long, R. Vilar, A. D. Gee: Synthesis of 11C, 18F, 15O, and 13N radiolabels for positron emission tomography. In: Angewandte Chemie. Band 47, Nummer 47, 2008, S. 8998–9033, doi:10.1002/anie.200800222. PMID 18988199.
- A. M. Tolkovsky, A. Wyttenbach: Differential phosphoprotein labelling (DIPPL) using 32P and 33P. In: Methods in molecular biology. Band 527, 2009, S. 21–9, xi, doi:10.1007/978-1-60327-834-8_2. PMID 19241002.
- M. A. Lydan, D. H. O’Day: Production of 35S-labeled proteins in E. coli and their use as molecular probes. In: Methods in molecular biology. Band 31, 1994, S. 389–396, doi:10.1385/0-89603-258-2:389. PMID 7921035.
- F. W. FITCH, J. WINEBRIGHT, P. V. HARPER: Iodine-125 as a protein label in immunology. In: Science. Band 135, Nummer 3508, März 1962, S. 1068–1069, PMID 13893326.
- L. R. MELCHER, S. P. MASOUREDIS: The in vivo stability of the I131 protein label of rabbit antibody in guinea pigs as determined by the quantitative precipitin reaction. In: Journal of Immunology (Baltimore, Md. : 1950). Band 67, Nummer 5, November 1951, S. 393–402, PMID 14898075.
- C. Xavier, N. Devoogdt, S. Hernot, I. Vaneycken, M. D’Huyvetter, J. De Vos, S. Massa, T. Lahoutte, V. Caveliers: Site-specific labeling of his-tagged Nanobodies with 99mTc: a practical guide. In: Methods in molecular biology. Band 911, 2012, S. 485–490, doi:10.1007/978-1-61779-968-6_30. PMID 22886271.
- M. H. Adatepe, P. Penkoske, A. Van Amberg, T. Wharton, R. G. Evens, E. J. Potchen: Red cell and plasma protein labeling with 113 m In. In: The International journal of applied radiation and isotopes. Band 22, Nummer 8, August 1971, S. 498–501, PMID 5097086.
- J. A. Prescher, D. H. Dube, Carolyn Bertozzi: Chemical remodelling of cell surfaces in living animals. In: Nature. Band 430, Nummer 7002, August 2004, S. 873–877, doi:10.1038/nature02791. PMID 15318217.
- J. A. Prescher, C. R. Bertozzi: Chemistry in living systems. In: Nature Chemical Biology. Band 1, Nummer 1, Juni 2005, S. 13–21, doi:10.1038/nchembio0605-13. PMID 16407987.
- A. B. Neef, N. W. Luedtke: Dynamic metabolic labeling of DNA in vivo with arabinosyl nucleosides. In: Proceedings of the National Academy of Sciences. Band 108, Nummer 51, Dezember 2011, S. 20404–20409, doi:10.1073/pnas.1101126108. PMID 22143759. PMC 3251089 (freier Volltext).
- H. C. Kolb, M. G. Finn, K. B. Sharpless: Click Chemistry: Diverse Chemical Function from a Few Good Reactions. In: Angewandte Chemie. Band 40, Nummer 11, Juni 2001, S. 2004–2021, PMID 11433435.
- A. Cieslar-Pobuda, M. J. Los: Prospects and limitations of „Click-Chemistry“-based DNA labeling technique employing 5-ethynyl-2'deoxyuridine (EdU). In: Cytometry. Part A : the journal of the International Society for Analytical Cytology. Band 83, Nummer 11, November 2013, S. 977–978, doi:10.1002/cyto.a.22394. PMID 24115755.
- J. M. Baskin, J. A. Prescher, S. T. Laughlin, N. J. Agard, P. V. Chang, I. A. Miller, A. Lo, J. A. Codelli, C. R. Bertozzi: Copper-free click chemistry for dynamic in vivo imaging. In: Proceedings of the National Academy of Sciences. Band 104, Nummer 43, Oktober 2007, S. 16793–16797, doi:10.1073/pnas.0707090104. PMID 17942682, PMC 2040404 (freier Volltext).
- Xinghai Ning, Rinske P. Temming u. a.: Protein Modification by Strain-Promoted Alkyne-Nitrone Cycloaddition. In: Angewandte Chemie International Edition. 49, 2010, S. 3065–3068, doi:10.1002/anie.201000408.
- K. J. Yarema, LK Mahal, RE Bruehl, EC Rodriguez, CR Bertozzi: Metabolic Delivery of Ketone Groups to Sialic Acid Residues. APPLICATION TO CELL SURFACE GLYCOFORM ENGINEERING. In: Journal of Biological Chemistry. 273, Nr. 47, 1998, S. 31168–79. doi:10.1074/jbc.273.47.31168. PMID 9813021.
- Melissa L. Blackman, Maksim Royzen, Joseph M. Fox: The Tetrazine Ligation: Fast Bioconjugation based on Inverse-electron-demand Diels-Alder Reactivity. In: Journal of the American Chemical Society. 130, Nr. 41, 2008, S. 13518–9. doi:10.1021/ja8053805. PMID 18798613. PMC 2653060 (freier Volltext).
- Henning Stöckmann, André A. Neves, Shaun Stairs, Kevin M. Brindle, Finian J. Leeper: Exploring isonitrile-based click chemistry for ligation with biomolecules. In: Organic & Biomolecular Chemistry. 9, Nr. 21, 2011, S. 7303. doi:10.1039/C1OB06424J.
- Ellen M. Sletten, Carolyn R. Bertozzi: A Bioorthogonal Quadricyclane Ligation. In: Journal of the American Chemical Society. 133, Nr. 44, 2011, S. 17570–3. doi:10.1021/ja2072934. PMID 21962173. PMC 3206493 (freier Volltext).
- E. Saxon, C. R. Bertozzi: Cell surface engineering by a modified Staudinger reaction. In: Science. Band 287, Nummer 5460, März 2000, S. 2007–2010, PMID 10720325.
- N. J. Agard, J. M. Baskin, J. A. Prescher, A. Lo, C. R. Bertozzi: A comparative study of bioorthogonal reactions with azides. In: ACS chemical biology. Band 1, Nummer 10, November 2006, S. 644–648, PMID 17175580.
- S. H. Weisbrod, A. Baccaro, A. Marx: Site-specific DNA labeling by Staudinger ligation. In: Methods in molecular biology. Band 751, 2011, S. 195–207, doi:10.1007/978-1-61779-151-2_12. PMID 21674332.
- J. O’Grady, J. Schwender, Y. Shachar-Hill, J. A. Morgan: Metabolic cartography: experimental quantification of metabolic fluxes from isotopic labelling studies. In: Journal of experimental botany. Band 63, Nummer 6, März 2012, S. 2293–2308, doi:10.1093/jxb/ers032. PMID 22371075. PDF.
- M. A. Orman, F. Berthiaume, I. P. Androulakis, M. G. Ierapetritou: Advanced stoichiometric analysis of metabolic networks of mammalian systems. In: Critical reviews in biomedical engineering. Band 39, Nummer 6, 2011, S. 511–534, PMID 22196224. PMC 3634616 (freier Volltext).
- S. B. Crown, M. R. Antoniewicz: Parallel labeling experiments and metabolic flux analysis: Past, present and future methodologies. In: Metabolic engineering. Band 16, März 2013, S. 21–32, doi:10.1016/j.ymben.2012.11.010. PMID 23246523.
- X. Chen, Y. Shachar-Hill: Insights into metabolic efficiency from flux analysis. In: Journal of experimental botany. Band 63, Nummer 6, März 2012, S. 2343–2351, doi:10.1093/jxb/ers057. PMID 22378949. PDF.
- W. H. So, Y. Zhang, W. Kang, C. T. Wong, H. Sun, J. Xia: Site-selective covalent reactions on proteinogenic amino acids. In: Current Opinion in Biotechnology. Band 48, Dezember 2017, S. 220–227, doi:10.1016/j.copbio.2017.06.003, PMID 28688251.
- I. M. Riederer, R. M. Herrero, G. Leuba, B. M. Riederer: Serial protein labeling with infrared maleimide dyes to identify cysteine modifications. In: Journal of proteomics. Band 71, Nummer 2, Juli 2008, S. 222–230, doi:10.1016/j.jprot.2008.04.006. PMID 18556256.
- J. Guy, R. Castonguay, N. B. Campos-Reales Pineda, V. Jacquier, K. Caron, S. W. Michnick, J. W. Keillor: De novo helical peptides as target sequences for a specific, fluorogenic protein labelling strategy. In: Molecular bioSystems. Band 6, Nummer 6, Juni 2010, S. 976–987, doi:10.1039/b918205e. PMID 20485742.
- H. Kratz, A. Haeckel, R. Michel, L. Schönzart, U. Hanisch, B. Hamm, E. Schellenberger: Straightforward thiol-mediated protein labelling with DTPA: Synthesis of a highly active 111In-annexin A5-DTPA tracer. In: EJNMMI research. Band 2, Nummer 1, 2012, S. 17, doi:10.1186/2191-219X-2-17. PMID 22541756. PMC 3444359 (freier Volltext).
- K. K. Han, A. Delacourte, B. Hemon: Chemical modification of thiol group(s) in protein: application to the study of anti-microtubular drugs binding. In: Comparative biochemistry and physiology. B, Comparative biochemistry. Band 88, Nummer 4, 1987, S. 1057–1065, PMID 3322663.
- R. A. Evangelista, A. Pollak, B. Allore, E. F. Templeton, R. C. Morton, E. P. Diamandis: A new europium chelate for protein labelling and time-resolved fluorometric applications. In: Clinical biochemistry. Band 21, Nummer 3, Juni 1988, S. 173–178, PMID 3390907.
- J. Roeser, R. Bischoff, A. P. Bruins, H. P. Permentier: Oxidative protein labeling in mass-spectrometry-based proteomics. In: Analytical and bioanalytical chemistry. Band 397, Nummer 8, August 2010, S. 3441–3455, doi:10.1007/s00216-010-3471-8. PMID 20155254. PMC 2911539 (freier Volltext).
- N. Jentoft, D. G. Dearborn: Protein labeling by reductive alkylation. In: Methods in enzymology. Band 91, 1983, S. 570–579, PMID 6855602.
- S. Tsukiji, M. Miyagawa, Y. Takaoka, T. Tamura, I. Hamachi: Ligand-directed tosyl chemistry for protein labeling in vivo. In: Nature chemical biology. Band 5, Nummer 5, Mai 2009, S. 341–343, doi:10.1038/nchembio.157. PMID 19330012.
- T. Matsumoto, R. Takase, T. Tanaka, H. Fukuda, A. Kondo: Site-specific protein labeling with amine-containing molecules using Lactobacillus plantarum sortase. In: Biotechnology journal. Band 7, Nummer 5, Mai 2012, S. 642–648, doi:10.1002/biot.201100213. PMID 21922670.
- N. Kamiya, H. Abe, M. Goto, Y. Tsuji, H. Jikuya: Fluorescent substrates for covalent protein labeling catalyzed by microbial transglutaminase. In: Organic & biomolecular chemistry. Band 7, Nummer 17, September 2009, S. 3407–3412, doi:10.1039/b904046c. PMID 19675894.
- G. V. Los, L. P. Encell, M. G. McDougall, D. D. Hartzell, N. Karassina, C. Zimprich, M. G. Wood, R. Learish, R. F. Ohana, M. Urh, D. Simpson, J. Mendez, K. Zimmerman, P. Otto, G. Vidugiris, J. Zhu, A. Darzins, D. H. Klaubert, R. F. Bulleit, K. V. Wood: HaloTag: a novel protein labeling technology for cell imaging and protein analysis. In: ACS chemical biology. Band 3, Nummer 6, Juni 2008, S. 373–382, doi:10.1021/cb800025k. PMID 18533659.
- Y. Zou, J. Yin: Phosphopantetheinyl transferase catalyzed site-specific protein labeling with ADP conjugated chemical probes. In: Journal of the American Chemical Society. Band 131, Nummer 22, Juni 2009, S. 7548–7549, doi:10.1021/ja902464v. PMID 19441828.
- J. Roeser, R. Bischoff, A. P. Bruins, H. P. Permentier: Oxidative protein labeling in mass-spectrometry-based proteomics. In: Analytical and bioanalytical chemistry. Band 397, Nummer 8, August 2010, S. 3441–3455, doi:10.1007/s00216-010-3471-8. PMID 20155254. PMC 2911539 (freier Volltext).
- M. Suchanek, A. Radzikowska, C. Thiele: Photo-leucine and photo-methionine allow identification of protein–protein interactions in living cells. In: Nature Methods. 2, Nr. 4, 2005, S. 261–268. doi:10.1038/nmeth752. PMID 15782218.
- J. W. Chin, S. W. Santoro, A. B. Martin, D. S. King, L. Wang, P. G. Schultz: Addition of p-azido-L-phenylalanine to the genetic code of Escherichia coli. In: Journal of the American Chemical Society. Band 124, Nummer 31, August 2002, S. 9026–9027, PMID 12148987.
- D. F. Winkler, P. L. McGeer: Protein labeling and biotinylation of peptides during spot synthesis using biotin p-nitrophenyl ester (biotin-ONp). In: Proteomics. Band 8, Nummer 5, März 2008, S. 961–967, doi:10.1002/pmic.200700909. PMID 18324722.
- S. Mamaev, J. Olejnik, E. K. Olejnik, K. J. Rothschild: Cell-free N-terminal protein labeling using initiator suppressor tRNA. In: Analytical biochemistry. Band 326, Nummer 1, März 2004, S. 25–32, doi:10.1016/j.ab.2003.11.002. PMID 14769332.
- D. A. Fancy: Elucidation of protein-protein interactions using chemical cross-linking or label transfer techniques. In: Current opinion in chemical biology. Band 4, Nummer 1, Februar 2000, S. 28–33, PMID 10679368.
- S. S. Andrews, Z. B. Hill, B. G. Perera, D. J. Maly: Label transfer reagents to probe p38 MAPK binding partners. In: ChemBioChem. Band 14, Nummer 2, Januar 2013, S. 209–216, doi:10.1002/cbic.201200673. PMID 23319368. PMC 3762675 (freier Volltext).
- J. B. Denny, Günter Blobel: 125I-labeled crosslinking reagent that is hydrophilic, photoactivatable, and cleavable through an azo linkage. In: Proceedings of the National Academy of Sciences. Band 81, Nummer 17, September 1984, S. 5286–5290, PMID 6433347. PMC 391688 (freier Volltext).
- M. M. Shaw, B. M. Riederer: Sample preparation for two-dimensional gel electrophoresis. In: Proteomics. Band 3, Nummer 8, August 2003, S. 1408–1417, doi:10.1002/pmic.200300471. PMID 12923765.
- J. F. Timms, R. Cramer: Difference gel electrophoresis. In: Proteomics. Band 8, Nummer 23–24, Dezember 2008, S. 4886–4897, doi:10.1002/pmic.200800298. PMID 19003860.
- B. M. Riederer: Non-covalent and covalent protein labeling in two-dimensional gel electrophoresis. In: Journal of proteomics. Band 71, Nummer 2, Juli 2008, S. 231–244, doi:10.1016/j.jprot.2008.05.001. PMID 18556257.
- O. Söderberg, M. Gullberg, M. Jarvius, K. Ridderstråle, K. J. Leuchowius, J. Jarvius, K. Wester, P. Hydbring, F. Bahram, L. G. Larsson, U. Landegren: Direct observation of individual endogenous protein complexes in situ by proximity ligation. In: Nature methods. Band 3, Nummer 12, Dezember 2006, S. 995–1000, doi:10.1038/nmeth947. PMID 17072308.
- D. J. Hnatowich, G. Mardirossian, M. Rusckowski, P. Winnard: Protein labelling via deoxyribonucleic acid hybridization. In: Nuclear medicine communications. Band 17, Nummer 1, Januar 1996, S. 66–75, PMID 8692476.
- K. H. Lim, H. Huang, A. Pralle, S. Park: Stable, high-affinity streptavidin monomer for protein labeling and monovalent biotin detection. In: Biotechnology and bioengineering. Band 110, Nummer 1, Januar 2013, S. 57–67, doi:10.1002/bit.24605. PMID 22806584.
- V. Tolmachev, A. Orlova: Influence of labelling methods on biodistribution and imaging properties of radiolabelled peptides for visualisation of molecular therapeutic targets. In: Current medicinal chemistry. Band 17, Nummer 24, 2010, S. 2636–2655, PMID 20491631.
- V. Tolmachev, S. Stone-Elander: Radiolabelled proteins for positron emission tomography: Pros and cons of labelling methods. In: Biochimica et Biophysica Acta. Band 1800, Nummer 5, Mai 2010, S. 487–510, doi:10.1016/j.bbagen.2010.02.002. PMID 20153401.
- J. Meisenhelder, T. Hunter: Radioactive protein-labelling techniques. In: Nature. Band 335, Nummer 6186, September 1988, S. 120, doi:10.1038/335120a0. PMID 3412469.
- S. Caplan, M. Baniyash: Radioiodination of cellular proteins. In: Current protocols in cell biology / editorial board, Juan S. Bonifacino … [et al.]. Chapter 7, August 2002, S. Unit 7.10, doi:10.1002/0471143030.cb0710s15. PMID 18228409.
- M. Sunbul, J. Yin: Site specific protein labeling by enzymatic posttranslational modification. In: Organic & biomolecular chemistry. Band 7, Nummer 17, September 2009, S. 3361–3371, doi:10.1039/b908687k. PMID 19675886.
- s. Bonifacino
- W. P. Heal, M. H. Wright, E. Thinon, E. W. Tate: Multifunctional protein labeling via enzymatic N-terminal tagging and elaboration by click chemistry. In: Nature protocols. Band 7, Nummer 1, Januar 2012, S. 105–117, doi:10.1038/nprot.2011.425. PMID 22193303.
- M. W. Rose, J. Xu, T. A. Kale, G. O’Doherty, G. Barany, M. D. Distefano: Enzymatic incorporation of orthogonally reactive prenylazide groups into peptides using geranylazide diphosphate via protein farnesyltransferase: implications for selective protein labeling. In: Biopolymers. Band 80, Nummer 2–3, 2005, S. 164–171, doi:10.1002/bip.20239. PMID 15810014.
- K. M. Coombs: Quantitative proteomics of complex mixtures. In: Expert review of proteomics. Band 8, Nummer 5, Oktober 2011, S. 659–677, doi:10.1586/epr.11.55. PMID 21999835.
- Y. Xu, S. Matthews: TROSY NMR spectroscopy of large soluble proteins. In: Topics in current chemistry. Band 335, 2013, S. 97–119, doi:10.1007/128_2011_228. PMID 21928013.
- A. Audhya, A. Desai: Proteomics in Caenorhabditis elegans. In: Briefings in functional genomics & proteomics. Band 7, Nummer 3, Mai 2008, S. 205–210, doi:10.1093/bfgp/eln014. PMID 18372286.
- S. Mizukami: Development of molecular imaging tools to investigate protein functions by chemical probe design. In: Chemical & pharmaceutical bulletin. Band 59, Nummer 12, 2011, S. 1435–1446, PMID 22130363.
- A. Ross, W. Kessler, D. Krumme, U. Menge, J. Wissing, J. van den Heuvel, L. Flohé: Optimised fermentation strategy for 13C/15N recombinant protein labelling in Escherichia coli for NMR-structure analysis. In: Journal of biotechnology. Band 108, Nummer 1, Februar 2004, S. 31–39, PMID 14741767.
- J. Li, S. Lin, J. Wang, S. Jia, M. Yang, Z. Hao, X. Zhang, P. R. Chen: Ligand-free palladium-mediated site-specific protein labeling inside gram-negative bacterial pathogens. In: Journal of the American Chemical Society. Band 135, Nummer 19, Mai 2013, S. 7330–7338, doi:10.1021/ja402424j. PMID 23641876.
- Ilya A. Osterman, Alexey V. Ustinov, Denis V. Evdokimov, Vladimir A. Korshun, Petr V. Sergiev, Marina V. Serebryakova, Irina A. Demina, Maria A. Galyamina, Vadim M. Govorun, Olga A. Dontsova: A nascent proteome study combining click chemistry with 2DE Archiviert vom Original am 30. Juni 2015. Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis. In: PROTEOMICS. 13, Nr. 1, Januar 2013, S. 17–21. doi:10.1002/pmic.201200393. PMID 23161590. Abgerufen am 27. Juni 2015.
- C. Uttamapinant, M. I. Sanchez, D. S. Liu, J. Z. Yao, A. Y. Ting: Site-specific protein labeling using PRIME and chelation-assisted click chemistry. In: Nature protocols. Band 8, Nummer 8, August 2013, S. 1620–1634, doi:10.1038/nprot.2013.096. PMID 23887180.
- F. Truong, T. H. Yoo, T. J. Lampo, D. A. Tirrell: Two-strain, cell-selective protein labeling in mixed bacterial cultures. In: Journal of the American Chemical Society. Band 134, Nummer 20, Mai 2012, S. 8551–8556, doi:10.1021/ja3004667. PMID 22575034. PMC 3443257 (freier Volltext).
- M. E. Ourailidou, J. Y. van der Meer, B. J. Baas, M. Jeronimus-Stratingh, A. L. Gottumukkala, G. J. Poelarends, A. J. Minnaard, F. J. Dekker: Aqueous oxidative heck reaction as a protein-labeling strategy. In: Chembiochem : a European journal of chemical biology. Band 15, Nummer 2, Januar 2014, S. 209–212, doi:10.1002/cbic.201300714. PMID 24376051.
- S. Voss, L. Zhao, X. Chen, F. Gerhard, Y. W. Wu: Generation of an intramolecular three-color fluorescence resonance energy transfer probe by site-specific protein labeling. In: Journal of peptide science : an official publication of the European Peptide Society. [elektronische Veröffentlichung vor dem Druck] Januar 2014, doi:10.1002/psc.2590. PMID 24395760.
- Z. Yu, Y. Pan, Z. Wang, J. Wang, Q. Lin: Genetically encoded cyclopropene directs rapid, photoclick-chemistry-mediated protein labeling in mammalian cells. In: Angewandte Chemie. Band 51, Nummer 42, Oktober 2012, S. 10600–10604, doi:10.1002/anie.201205352. PMID 22997015. PMC 3517012 (freier Volltext).
- D. P. Nguyen, T. Elliott, M. Holt, T. W. Muir, J. W. Chin: Genetically encoded 1,2-aminothiols facilitate rapid and site-specific protein labeling via a bio-orthogonal cyanobenzothiazole condensation. In: Journal of the American Chemical Society. Band 133, Nummer 30, August 2011, S. 11418–11421, doi:10.1021/ja203111c. PMID 21736333.
- R. K. Lim, Q. Lin: Photoinducible bioorthogonal chemistry: a spatiotemporally controllable tool to visualize and perturb proteins in live cells. In: Accounts of chemical research. Band 44, Nummer 9, September 2011, S. 828–839, doi:10.1021/ar200021p. PMID 21609129. PMC 3175026 (freier Volltext).
- G. C. Rudolf, W. Heydenreuter, S. A. Sieber: Chemical proteomics: ligation and cleavage of protein modifications. In: Current opinion in chemical biology. Band 17, Nummer 1, Februar 2013, S. 110–117, doi:10.1016/j.cbpa.2012.11.007. PMID 23273612.
- Y. Takaoka, A. Ojida, I. Hamachi: Protein organic chemistry and applications for labeling and engineering in live-cell systems. In: Angewandte Chemie. Band 52, Nummer 15, April 2013, S. 4088–4106, doi:10.1002/anie.201207089. PMID 23426903.
- M. Fernández-Suárez, H. Baruah, L. Martínez-Hernández, K. T. Xie, J. M. Baskin, C. R. Bertozzi, A. Y. Ting: Redirecting lipoic acid ligase for cell surface protein labeling with small-molecule probes. In: Nature Biotechnology. Band 25, Nummer 12, Dezember 2007, S. 1483–1487, doi:10.1038/nbt1355. PMID 18059260. PMC 2654346 (freier Volltext).
- D. W. Romanini, V. W. Cornish: Protein labelling: Playing tag with proteins. In: Nature chemistry. Band 4, Nummer 4, April 2012, S. 248–250, doi:10.1038/nchem.1325. PMID 22437705.
- D. Maurel, S. Banala, T. Laroche, K. Johnsson: Photoactivatable and photoconvertible fluorescent probes for protein labeling. In: ACS chemical biology. Band 5, Nummer 5, Mai 2010, S. 507–516, doi:10.1021/cb1000229. PMID 20218675.
- D. Srikun, A. E. Albers, C. I. Nam, A. T. Iavarone, C. J. Chang: Organelle-targetable fluorescent probes for imaging hydrogen peroxide in living cells via SNAP-Tag protein labeling. In: Journal of the American Chemical Society. Band 132, Nummer 12, März 2010, S. 4455–4465, doi:10.1021/ja100117u. PMID 20201528. PMC 2850560 (freier Volltext).
- A. A. Ruggiu, M. Bannwarth, K. Johnsson: Fura-2FF-based calcium indicator for protein labeling. In: Organic & biomolecular chemistry. Band 8, Nummer 15, August 2010, S. 3398–3401, doi:10.1039/c000158a. PMID 20556282.
- C Zhao, LM Hellman, X Zhan, WS Bowman, SW Whiteheart, MG Fried: Hexahistidine-tag-specific optical probes for analyses of proteins and their interactions. In: Analytical Biochemistry. 399, Nr. 2, 2010, S. 237–45. doi:10.1016/j.ab.2009.12.028. PMID 20036207. PMC 2832190 (freier Volltext).
- S. R. Adams, R. E. Campbell, L. A. Gross, B. R. Martin, G. K. Walkup, Y. Yao, J. Llopis, Roger Yonchien Tsien: New biarsenical ligands and tetracysteine motifs for protein labeling in vitro and in vivo: synthesis and biological applications. In: Journal of the American Chemical Society. Band 124, Nummer 21, Mai 2002, S. 6063–6076, PMID 12022841.
- T. Machleidt, M. Robers, G. T. Hanson: Protein labeling with FlAsH and ReAsH. In: Methods in molecular biology. Band 356, 2007, S. 209–220, PMID 16988405.
- H. Nonaka, S. H. Fujishima, S. H. Uchinomiya, A. Ojida, I. Hamachi: FLAG-tag selective covalent protein labeling via a binding-induced acyl-transfer reaction. In: Bioorganic & medicinal chemistry letters. Band 19, Nummer 23, Dezember 2009, S. 6696–6699, doi:10.1016/j.bmcl.2009.09.122. PMID 19837586.
- G. M. Eldridge, G. A. Weiss: Hydrazide reactive peptide tags for site-specific protein labeling. In: Bioconjugate Chemistry. Band 22, Nummer 10, Oktober 2011, S. 2143–2153, doi:10.1021/bc200415v. PMID 21905743. PMC 3199291 (freier Volltext).
- A. Yoshimura, S. Mizukami, Y. Hori, S. Watanabe, K. Kikuchi: Cell-surface protein labeling with luminescent nanoparticles through biotinylation by using mutant beta-lactamase-tag technology. In: Chembiochem : a European journal of chemical biology. Band 12, Nummer 7, Mai 2011, S. 1031–1034, doi:10.1002/cbic.201100021. PMID 21425232.
- C. Uttamapinant, K. A. White, H. Baruah, S. Thompson, M. Fernández-Suárez, S. Puthenveetil, A. Y. Ting: A fluorophore ligase for site-specific protein labeling inside living cells. In: Proceedings of the National Academy of Sciences. Band 107, Nummer 24, Juni 2010, S. 10914–10919, doi:10.1073/pnas.0914067107. PMID 20534555. PMC 2890758 (freier Volltext).
- I. V. Thiel, G. Volkmann, S. Pietrokovski, H. D. Mootz: An atypical naturally split intein engineered for highly efficient protein labeling. In: Angewandte Chemie. Band 53, Nummer 5, Januar 2014, S. 1306–1310, doi:10.1002/anie.201307969. PMID 24382817.
- I. Ghosh, N. Considine, E. Maunus, L. Sun, A. Zhang, J. Buswell, T. C. Evans, M. Q. Xu: Site-specific protein labeling by intein-mediated protein ligation. In: Methods in molecular biology. Band 705, 2011, S. 87–107, doi:10.1007/978-1-61737-967-3_6. PMID 21125382.
- J. Y. Yang, W. Y. Yang: Site-specific two-color protein labeling for FRET studies using split inteins. In: Journal of the American Chemical Society. Band 131, Nummer 33, August 2009, S. 11644–11645, doi:10.1021/ja9030215. PMID 19645470.
- Y. Du, C. E. Hendrick, K. S. Frye, L. R. Comstock: Fluorescent DNA labeling by N-mustard analogues of S-adenosyl-L-methionine. In: Chembiochem : a European journal of chemical biology. Band 13, Nummer 15, Oktober 2012, S. 2225–2233, doi:10.1002/cbic.201200438. PMID 22961989.
- S. E. Walker, J. Lorsch: Sanger dideoxy sequencing of DNA. In: Methods in enzymology. Band 529, 2013, S. 171–184, doi:10.1016/B978-0-12-418687-3.00014-8. PMID 24011045.
- R. Redon, T. Fitzgerald, N. P. Carter: Comparative genomic hybridization: DNA labeling, hybridization and detection. In: Methods in molecular biology. Band 529, 2009, S. 267–278, doi:10.1007/978-1-59745-538-1_17. PMID 19381974. PMC 2867219 (freier Volltext).
- H. Zohar, S. J. Muller: Labeling DNA for single-molecule experiments: methods of labeling internal specific sequences on double-stranded DNA. In: Nanoscale. Band 3, Nummer 8, August 2011, S. 3027–3039, doi:10.1039/c1nr10280j. PMID 21734993. PMC 3322637 (freier Volltext).
- E. Paredes, M. Evans, S. R. Das: RNA labeling, conjugation and ligation. In: Methods. Band 54, Nummer 2, Juni 2011, S. 251–259, doi:10.1016/j.ymeth.2011.02.008. PMID 21354310.
- D. V. Bann, L. J. Parent: Application of live-cell RNA imaging techniques to the study of retroviral RNA trafficking. In: Viruses. Band 4, Nummer 6, Juni 2012, S. 963–979, doi:10.3390/v4060963. PMID 22816035. PMC 3397357 (freier Volltext).
- J. S. Kaddis, D. H. Wai, J. Bowers, N. Hartmann, L. Baeriswyl, S. Bajaj, M. J. Anderson, R. C. Getts, T. J. Triche: Influence of RNA labeling on expression profiling of microRNAs. In: The Journal of Molecular Diagnostics, Band 14, Nummer 1, Januar 2012, S. 12–21, doi:10.1016/j.jmoldx.2011.08.005. PMID 22074760. PMC 3338349 (freier Volltext).
- K. Cole, V. Truong, D. Barone, G. McGall: Direct labeling of RNA with multiple biotins allows sensitive expression profiling of acute leukemia class predictor genes. In: Nucleic Acids Research, Band 32, Nummer 11, 2004, S. e86, doi:10.1093/nar/gnh085. PMID 15205470. PMC 443553 (freier Volltext).
- V. Olieric, U. Rieder, K. Lang, A. Serganov, C. Schulze-Briese, R. Micura, P. Dumas, E. Ennifar: A fast selenium derivatization strategy for crystallization and phasing of RNA structures. In: RNA. Band 15, Nummer 4, April 2009, S. 707–715, doi:10.1261/rna.1499309. PMID 19228585. PMC 2661828 (freier Volltext).
- S. C. Walker, F. H. Scott, C. Srisawat, D. R. Engelke: RNA affinity tags for the rapid purification and investigation of RNAs and RNA-protein complexes. In: Methods in molecular biology. Band 488, 2008, S. 23–40, doi:10.1007/978-1-60327-475-3_3. PMID 18982282. PMC 2807123 (freier Volltext).
- J. S. Paige, K. Y. Wu, S. R. Jaffrey: RNA mimics of green fluorescent protein. In: Science. Band 333, Nummer 6042, Juli 2011, S. 642–646, doi:10.1126/science.1207339. PMID 21798953. PMC 3314379 (freier Volltext).
- R. M. Martin, J. Rino, C. Carvalho, T. Kirchhausen, M. Carmo-Fonseca: Live-cell visualization of pre-mRNA splicing with single-molecule sensitivity. In: Cell reports. Band 4, Nummer 6, September 2013, S. 1144–1155, doi:10.1016/j.celrep.2013.08.013. PMID 24035393. PMC 3805459 (freier Volltext).
- J. A. Prescher, C. R. Bertozzi: Chemical technologies for probing glycans. In: Cell. Band 126, Nummer 5, September 2006, S. 851–854, doi:10.1016/j.cell.2006.08.017. PMID 16959565.
- X. L. Sun, W. Cui, C. Haller, E. L. Chaikof: Site-specific multivalent carbohydrate labeling of quantum dots and magnetic beads. In: Chembiochem : a European journal of chemical biology. Band 5, Nummer 11, November 2004, S. 1593–1596, doi:10.1002/cbic.200400137. PMID 15515080.