Transkription (Biologie)
Als Transkription (von spätlateinisch transcriptio „Übertragung“ zu lateinisch transcribere „um-/ überschreiben“) wird in der Genetik die Synthese von RNA anhand einer DNA als Vorlage bezeichnet. Die dabei entstehende RNA lässt sich in verschiedene Hauptgruppen einteilen, von denen insbesondere drei bei der Proteinbiosynthese in der Zelle eine bedeutende Rolle spielen: mRNA (messenger RNA) sowie tRNA (transfer RNA) und rRNA (ribosomale RNA). Die Transkription ist, wie auch die Translation, ein wesentlicher Teilprozess der Genexpression.
| Übergeordnet |
| Genexpression RNA-Biosynthese |
| Untergeordnet |
| Initiation Elongation Termination |
| Gene Ontology |
|---|
| QuickGO |
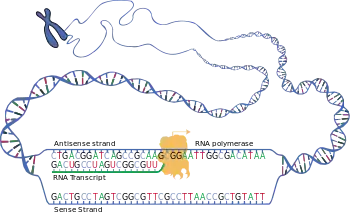
Bei der Transkription wird ein Gen abgelesen und seine Information kopiert, indem dessen Basensequenz in die Basensequenz eines neuaufgebauten RNA-Moleküls umgeschrieben wird. Der jeweils spezifische DNA-Abschnitt dient hierbei als Vorlage (Matrize, englisch template) für die Synthese eines neuen RNA-Strangs. Bei diesem Vorgang werden die Nukleinbasen der DNA (A – T – G – C) in die Nukleinbasen der RNA (U – A – C – G) umgeschrieben. Anstelle des Thymins kommt Uracil und anstelle der Desoxyribose kommt Ribose in der RNA vor.
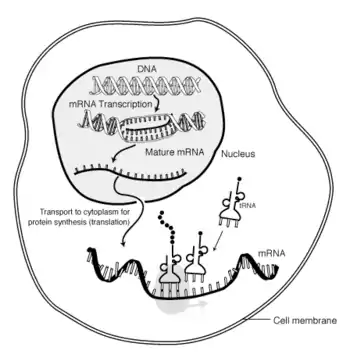
Der Vorgang der Transkription verläuft bei Eukaryoten und Prokaryoten grundsätzlich gleich. Unterschiede gibt es bei der Steuerung und bei der anschließenden Modifikation. Bei Prokaryoten erfolgt die Steuerung über einen Operator, während bei den Eukaryoten die Regulation über einen Enhancer oder Silencer geregelt werden kann, der jeweils dem Promotor vor- oder nachgeschaltet ist. Weiterhin erfolgt bei Prokaryoten die Transkription im Cytoplasma der Zelle, bei Eukaryoten im Zellkern (Karyoplasma). Bei Eukaryoten wird außerdem die prä-mRNA während beziehungsweise nach ihrer Synthese noch prozessiert, bevor sie aus dem Zellkern in das Cytoplasma transportiert wird. Nach der Transkription erfolgt im Cytoplasma am Ribosom die Translation der mRNA in ein Protein.
Synthese einer mRNA
Beginn der Transkription
Die Transkription beginnt, nachdem eine RNA-Polymerase an die Promotor genannte Region des DNA-Abschnitts eines Gens gebunden hat. Hierfür sind zusätzliche Proteinkomplexe erforderlich, die als generelle Transkriptionsfaktoren (TF) die Bindung vermitteln können. Sie erhöhen die Bindungswahrscheinlichkeit und führen die DNA-Vorlage näher an das katalytische Zentrum der Polymerase. Zusammen mit einer RNA-Polymerase bilden sie einen großen Proteinkomplex, den sogenannten RNA-Polymerase-Präinitiationskomplex.
Mit einer Helikase-Aktivität eines dieser Transkriptionsfaktoren (TFIIH) wird nun an einer bestimmten Stelle des DNA-Moleküls die Doppelhelix entspiralisiert, der Doppelstrang aufgetrennt und so auf einem DNA-Strang ein Abschnitt von jeweils ca. 10–20 Basen zur Paarung freigelegt. Nach der Entwindung der Doppelhelix phosphoryliert TFIIH die RNA-Polymerase II an ihrer carboxyterminalen Domäne (CTD), sodass diese mit der Elongation beginnen kann.[1] Infolge Basenpaarung lagern sich am codogenen Strang (Matrizenstrang, auch Antisense-Strang) der DNA dann Ribonukleotide mit komplementären Basen an. Sie werden unter Eliminierung von Pyrophosphat aus den Nukleosidtriphosphaten durch eine Esterbindung zwischen Phosphat und Ribose miteinander verknüpft. Mit der Bindung der ersten beiden Ribonukleotide beginnt die RNA-Strangbildung und damit die eigentliche Transkription. Die RNA-Polymerase benötigt keinen Primer.
Bei der eukaryotischen mRNA-Synthese kommt zum gerade beschriebenen Ablauf noch die Synthese einer Cap-Struktur am 5’-Ende der mRNA hinzu, die deren Schutz dient sowie später als Signal für den Export aus dem Zellkern fungiert. Dieses sogenannte Capping passiert bereits, wenn das Transkript nur wenige Nukleotide lang ist, also noch vor dem üblichen Verlängerungsprozess, Elongation genannt.
Verlängerungsphase
Die Ableserichtung längs des DNA-Matrizenstrangs verläuft vom 3’-Ende zum 5′-Ende, die Syntheserichtung des komplementär dazu aufgebauten RNA-Strangs läuft dementsprechend von 5’ nach 3’.
Ende der Transkription
Beendet wird die Transkription am Terminator. Danach wird das mRNA-Transkript entlassen und die Polymerase löst sich von der DNA.
In eukaryotischen Zellen kann die RNA-Polymerase das Ende eines Gens nicht von alleine erkennen, sie braucht dazu Hilfsfaktoren, die mit der Polymerase in Wechselwirkung treten. Diese Proteinkomplexe erkennen die Polyadenylierungsstelle (5’-AAUAAA-3’), schneiden die RNA und leiten die Polyadenylierung ein, während die RNA-Polymerase gleichzeitig weiterarbeitet. Ein Modell für die Termination der Transkription ist, dass das noch immer weiter wachsende, nutzlose RNA-Ende von einer Exonuklease (Rat1) abgebaut wird, und zwar schneller, als es von der Polymerase verlängert wird. Erreicht die Exonuklease die Transkriptionsstelle, löst sich die Polymerase von der DNA, die Transkription ist endgültig beendet (Torpedo model of transcriptional termination). Darüber hinaus scheinen weitere Proteinkomplexe für eine Termination wichtig zu sein.
Zusätzliche Prozessionsschritte der mRNA
- Bei prokaryoten Zellen gelangt die mRNA nach dem Kopiervorgang direkt zu den Ribosomen, denn einen Zellkern als abgeteiltes Kompartiment haben diese Zelle nicht. Häufig lagern sich auch bereits Ribosomen an die eben entstehende mRNA an und beginnen schon mit der Translation – noch bevor die Transkription endgültig abgeschlossen ist (Poly-Ribosom-Complex).
- Bei eukaryoten Zellen verlässt das primäre RNA-Transkript noch nicht den Zellkern. Die im ersten Teil der Transkription entstandene RNA wird als unreife RNA, prä-mRNA oder hnRNA (heterogene nucleäre RNA) bezeichnet. Sie wird noch weiter prozessiert durch Spleißen (Splicing) sowie am 3′-Ende durch Polyadenylierung (Tailing). Über alternatives Splicing können aus demselben vorliegenden DNA-Abschnitt unterschiedliche mRNA-Moleküle entstehen. Anschließend verlässt die gereifte mRNA durch eine Kernpore den Zellkern und gelangt exportiert ins Zytoplasma, wo sie dann mit den Ribosomen in Wechselwirkung treten kann.
Synthese von tRNA und von rRNA
Die Transfer-RNA (tRNA) und die ribosomale RNA (rRNA) werden durch zwei andere RNA-Polymerasen an der DNA synthetisiert. Diese arbeiten beide nach einem etwas anderen Prinzip als die für die mRNA-Synthese zuständige RNA-Polymerase II und gleichen mehr den RNA-Polymerasen der Prokaryoten. Bei Eukaryoten erfolgt die Synthese der tRNA, der 5S rRNA und der 7SL-RNA durch die RNA-Polymerase III, die Synthese der rRNA und teilweise auch der sn-RNA (small-nuclear RNA) durch die RNA-Polymerase I.
Archaeelle Transkription
Die Gene der Archaea besitzen im Promotor eine TATA-Box genannte Konsensussequenz. Am Promotor binden die zwei Initiationsfaktoren der Archaea, TBP und TFB. An diese bindet wiederum eine RNA-Polymerase, die ortholog zur eukaryotischen RNA-Polymerase II ist und aus zwölf Untereinheiten besteht.
Obgleich bei Genen für transfer-RNA (tRNA) und ribosomale RNA (rRNA) von Archaeen Introns – und deren Spleißen durch eine Endoribonuklease mit Ähnlichkeiten zu Untereinheiten entsprechender eukaryoter Enzymkomplexe – nicht selten sind,[2] kommen sie in proteincodierender pre-mRNA nur ausnahmsweise vor.[3]
Bakterielle Transkription
Im Gegensatz zu den Eukaryoten besitzen Bakterien nur eine RNA-Polymerase. Das Core- beziehungsweise Minimal-Enzym besteht aus vier Untereinheiten (2× α, β, β’), das die Transkription katalysiert, sie aber nicht initiiert. Der Core des Enzyms wirkt wechselseitig mit der losen Sigma-Untereinheit und es bildet sich das Holo-Enzym (2× α, β, β’, σ (Sigma)), welches die Initiation durchführen kann (Sigma ermöglicht das Entlanggleiten an der DNA und Auffinden der Pribnow-Box des Promotors). Die Sigma-Untereinheit bindet am Promotor des Nicht-Matrizenstranges und löst dort die Wasserstoffbrücken zwischen den Basenpaaren; sie besitzt eine Helicasefunktion, was die wichtigste Funktion dieser Polymerase ist.
Funktion der α-Untereinheit ist zum einen durch die aminoterminale Domäne bedingt der Erhalt und die Stabilität der Struktur, zum anderen durch die carboxyterminale Domäne eine Bindung an den Promotor und die Wechselwirkung mit Transkriptions-regulatorischen Elementen.
Die β- und β’-Untereinheiten wirken zusammen und sorgen für die Bindung an die DNA-Matrize und für eine wachsende RNA-Kette.
Die σ-(Sigma)-Untereinheit erkennt Transkriptionsstartpunkte. Es gibt mehrere Sigma-Untereinheiten, die verschiedene Gengruppen erkennen. Am weitesten verbreitet ist die σ70 Untereinheit.
Die ω-(Omega)-Untereinheit dient der Stabilisierung und der Strukturaufrechterhaltung und ist nicht zwingend notwendig.
Reverse Transkription
In einigen Viren gibt es den Sonderfall der reversen Transkription, bei der nach einer RNA-Vorlage DNA hergestellt wird.
Einzelnachweise
- Emmanuel Compe, Jean-Marc Egly: TFIIH: when transcription met DNA repair. In: Nature Reviews Molecular Cell Biology. Band 13, Nr. 6, Juni 2012, ISSN 1471-0080, S. 343–354, doi:10.1038/nrm3350 (nature.com [abgerufen am 27. Juli 2021]).
- J. Lykke-Andersen, C. Aagaard, M. Semionenkov, R. Garrett: Archaeal introns: splicing, intercellular mobility and evolution.` In: Trends in Biochemical Sciences. Band 22, Nr. 9, September 1997, S. 326–331; DOI:10.1016/S0968-0004(97)01113-4, PMID 9301331.
- Yoh-Ichi Watanabe et al.: Introns in protein-coding genes in Archaea. In: FEBS Letters. Band 510, Nr. 1–2, Januar 2002, S. 27–30; DOI:10.1016/S0014-5793(01)03219-7, PMID 11755525.
Literatur
- Rolf Knippers: Molekulare Genetik. 9. komplett überarbeitete Auflage. Thieme, Stuttgart u. a. 2006, ISBN 3-13-477009-1, S. 49–58.
Weblinks
- W. H. Freeman: Animation der Transkription, www.sumanasinc.com
- Zentrale für Unterrichtsmedien im Internet e. V. (ZUM): Transkription/Genetischer Code, www.zum.de