2D-Gelelektrophorese
Die zweidimensionale Gelelektrophorese oder 2D-Gelelektrophorese ist eine analytische Methode in Biochemie, Molekularbiologie und Proteomik. Sie wurde 1975 durch O'Farrell[1] und Klose[2] unabhängig voneinander entwickelt. Sie kombiniert die isoelektrische Fokussierung (IEF) mit der SDS-Polyacrylamidgelelektrophorese (SDS-PAGE) zur Trennung komplexer Proteingemische (Bakterienlysate, Lysate von höheren Zellen oder Geweben, Körperflüssigkeiten) in Einzelproteine. Durch die Kombination der beiden orthogonal zueinander ausgeführten Trenntechniken wird eine besonders hochauflösende Trennung erreicht.


Jeder Fleck (Spot) im Proteinmuster entspricht einer Sorte (Spezies) von Proteinmolekülen. Da sich Proteinmuster in biologischen Systemen umwelt- und zustandsabhängig verändern, können sie zur Unterscheidung geschädigter und gesunder oder auch optimal und suboptimal gewachsener Zellen herangezogen werden. Sie geben beispielsweise Aufschluss über Krankheitsursachen oder den Wirkungsmechanismus von Medikamenten auf molekularer Ebene. Aufgrund der Komplexität von zweidimensionalen Proteinmustern wird für deren Auswertung auf speziell entwickelte Computerprogramme zurückgegriffen.
Probenvorbereitung
Die Gewinnung und Verarbeitung der Probe erfolgt unter möglichst identischen Bedingungen. Damit wird ausgeschlossen, dass neben den zu untersuchenden experimentellen Variablen verfälschende Einflüsse auf die Probe einwirken. Aus extrazellulären Kompartimenten (sezernierte Proteine) werden die Proteine meist gefällt. Intrazelluläre Proteine extrahiert man durch schonende Zerstörung der Zellstrukturen und eine teilweise Proteinreinigung. Außerhalb ihrer natürlichen Umgebung sind die Proteine besonders anfällig für die Bildung von Proteinaggregaten und den Abbau durch Proteasen. Ferner wird bei der Probenvorbereitung bei 4 °C gearbeitet. In der Regel werden Harnstoff als Chaotrop und nichtionische Detergentien sowie proteasehemmende Stoffe zugesetzt,[3] um Wechselwirkungen und Veränderungen der Proteine zu vermeiden.
Erste Dimension (IEF)
Während der IEF (erste Dimension) wird der Proteinextrakt in einem pH-Gradienten-Gel im elektrischen Feld eindimensional aufgetrennt. Die sauren und basischen Aminosäurereste der Proteine durchlaufen in Abhängigkeit vom Umgebungs-pH-Wert verschiedene (De)protonierungszustände und bestimmen so die Ladung des Proteins. Damit sind sie für die Wirkung des elektrischen Feldes auf die Proteine verantwortlich. Am isoelektrischen Punkt (pI) heben sich positive und negative Ladungen des Proteins auf. Der pI entspricht dem pH-Wert an dem das Protein eine Nettoladung von 0 besitzt. Es findet keine Kraftwirkung des elektrischen Feldes auf das nun ladungsneutrale Protein mehr statt und das Protein lagert sich ab. Durch Diffusion verursachte Ortsveränderungen in benachbarte pH-Bereiche führen zur erneuten elektrischen Ladung des Proteins. Das wieder wirksame elektrische Feld jedoch fördert das Protein sofort zurück an seinem isoelektrischen Punkt. Zwei verschiedene IEF-technologien stehen zur Verfügung.
- Immobilisierte pH-Gradienten (IPG): Der Gelstreifen besteht aus einer Polyacrylamidmatrix. In diese Matrix werden sogenannte Immobiline einpolymerisiert. Immobiline sind Acrylamidderivate mit funktionellen Gruppen für schwache Säuren oder Base, mit definiertem pK-Wert. Durch die Kopolymerisation des neutralen Acrylamids mit den im Gradienten zugesetzten sauren und basischen Immobilinen entsteht ein unveränderlicher imobiler pH-Gradient.[4][5]
- pH-Gradienten auf Basis von Trägerampholyten: Trägerampholyte sind synthetische Aminosäuren und liegen in einem Gelstreifen oder einem langen zylindrischen Gel (meist in einem Röhrchen) frei beweglich vor. Beim Anlegen eines elektrischen Feldes formen sie einen pH-Gradienten, der allerdings zeitinstabil ist. Mit zunehmender Zeit wandern die den Gradienten definierenden Ladungsträger zu den Gelenden und verformen über kurz oder lang dessen Verlauf.
Weiterhin kann als erste Dimension alternativ eine Polyacrylamid-Gelelektrophorese mit einem kationischen Detergens wie 16-BAC durchgeführt werden.[6][7]
Äquilibrierung
Bei der sogenannten Äquilibrierung, die sich der Trennung nach dem pI anschließt, wird das Gel mit den Proteinen vorerst reduziert (beispielsweise mit Mercaptoethanol oder Dithiothreitol). Dies dient der Beseitigung von Disulfidbrücken. Um eine Reoxidation der dabei entstandenen -SH HS-Gruppen zu Disulfid(-S-S-)Gruppen zu verhindern, werden im nächsten Schritt die HS-Gruppen z. B. mit Iodacetamid alkyliert. Schlussendlich werden die Proteine mit Natriumdodecylsulfat (sodium dodecyl sulphate – SDS) beladen. SDS ist ein negativ geladenes Detergenz. Pro 3 Aminosäuren bindet sich ca. ein SDS-Molekül über sein aliphatisches Ende durch hydrophobe Wechselwirkung an das Proteinmolekül. Mit der negativ geladenen Seite stößt es sich von den geladenen Enden in der Nachbarschaft gebundener SDS-Moleküle ab. Dies führt zur völligen Entfaltung (Linearisierung) der Proteinmoleküle. Je größer ein Proteinmolekül ist, desto länger sind die entstehenden mit SDS beladenen Ketten. Da abhängig von der Länge des Proteins sehr viele negativ geladene SDS-Moleküle binden, kann die Eigenladung für die meisten Proteine im Weiteren vernachlässigt werden.
Zweite Dimension (SDS-PAGE)
Der Gelstreifen mit den nach dem pH-Wert aufgetrennten und äquilibrierten Proteinen wird bei vertikalen Systemen auf die Kante eines quadratischen bzw. rechteckigen ebenfalls SDS-haltigen Polyacrylamidgels gelegt. Bei horizontalen Systemen wird der Gelstreifen dagegen einige Millimeter vom Gelrand entfernt auf das flache SDS-Gel gelegt. Die Proteine werden nun senkrecht zur ersten Dimension in einer zweiten Elektrophorese nach ihrer Größe getrennt. Beim Anlegen des elektrischen Feldes (Anode gegenüber vom IEF-Gelstreifen) wandern die entfalteten und von SDS umgebenen Proteine mit ihrem Überschuss negativer Ladungen durch das Gel, welches den Proteinen entsprechend ihrer Molekülgröße einen mehr oder minder großen Widerstand entgegensetzt. Kleine Moleküle wandern relativ ungestört und erreichen schnell die dem IEF-Gel abgewandte Gelkante, große Moleküle werden bei der Wanderung ständig vom Gel gebremst und kommen kaum voran. Die Trennung in der zweiten Dimension wird mit Ankunft der kleinen Proteine am, dem IEF-Gel abgewandten, Gelrand gestoppt. Sichtbar wird dies durch einen mitlaufenden Farbstoff, wie zum Beispiel Bromphenolblau. Dieser kann bereits bei der Äquilibrierung zugegeben werden. Damit das Proteinmuster nach erfolgter Trennung vorhanden bleibt, muss es in einem anschließenden Schritt fixiert werden. Dazu werden verschiedene Alkohole (Methanol oder Ethanol) und Essigsäure genutzt. Diese denaturieren die getrennten Proteine und entfernt das Tensid, wodurch die Proteine unlöslich werden. Dadurch wird Diffusion verhindert und das 2D Muster wird somit zeitstabil.
 Leeres SDS-Gel
Leeres SDS-Gel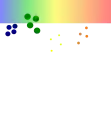 SDS-Gel während der Elektrophorese
SDS-Gel während der Elektrophorese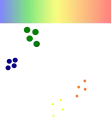 fertiges SDS-Gel
fertiges SDS-Gel
Die Proteine markieren und detektieren
Markierung in der lebenden Zelle (in vivo)
Parameter wie die Produktion von Proteinen in einem bestimmten Zeitraum (Proteinsyntheserate) oder die Phosphorylierung von Proteinen pro Zeiteinheit werden bevorzugt durch den Einbau von (radioaktiven) Isotopen bestimmt. Dazu wird der Bakterien/Zellkultur ein Nährstoffsubstrat mit einem außergewöhnlichen Isotop verabreicht, welches dann in die Proteine (35S) oder in die Phosphatgruppen phosphorylierter (32/33P) Proteine eingebaut wird. Wird die Zeitspanne des Einbaus relativ kurz gewählt, ist im Ergebnis eher eine Momentaufnahme des zellulären Geschehens erfassbar, bei langer Zeitspanne eher das kumulierte Bild vieler Einzelereignisse. Der Proteinextrakt der markierten Zellen wird separiert und der Anteil der markierten Proteine über Autoradiographie oder massenspektrometrische Verfahren im 2D Muster bestimmt. Eine metabolische Markierung kann auch mit chemisch modifizierten Aminosäuren erfolgen, welche für die spätere Markierung mit Fluoreszenzfarbstoffen mit einem biologisch inerten Linker ausgerüstet wurden (Bioorthogonale Markierung). Zur Bestimmung der akkumulierten Proteinmenge kann neben einer dauernden metabolischen oder Isotopenmarkierung auch auf eines der folgend erklärten Verfahren zurückgegriffen werden.
Markierung vor der gelelektrophoretischen Trennung
Besonders um mehrere Proben auf einem 2D-Gel zu trennen, werden kovalent bindende Fluoreszenzfarbstoffe eingesetzt. Dazu werden maximal drei verschiedene Proteinextrakte mit je einem Farbstoff markiert, gemischt und gemeinsam auf demselben Gel getrennt. Da die Farbstoffe separat voneinander detektiert werden können, ist die Generierung und der differentielle Vergleich dreier probenspezifischer Proteinmuster möglich (Difference Gel Electrophoresis, DIGE). Problematisch ist die Massenbeeinflussung der Proteine durch den gebundenen Farbstoff. Zudem sind die Farbstoffe relativ instabil und teuer.
Verwendet werden:
- Cy2; Cy3; Cy5
- Flashpro Farbstoffe
- G-Dyes
Markierung bzw. Färbung nach der Trennung
Die klassischen Proteinfärbungen erfolgen nach der elektrophoretischen Trennung. Eine Mengenbestimmung kann bei den nicht-linearen Färbeverfahren nur näherungsweise durch Versetzen der Probe mit Proteinen bekannter Konzentrationen erfolgen (z. B. ein Komigrationsstandard). Je nach gewünschter Spezifität und Sensitivität werden eingesetzt:
Adsorptionsfarbstoffe
- Coomassie-Brillant-Blau – Proteinmenge / nicht linear
- colloidal Coomassie-Brillant-Blau – Proteinmenge / nicht linear
- Silberfärbung – Proteinmenge / nicht linear / hoch sensitiv
- negative Zink Färbung – Proteinmenge / nicht linear / wenig sensitiv
Fluoreszenzfarbstoffe
- SYPRO Ruby – Proteinmenge / linear / sensitiv
- Krypton-Färbung – Proteinmenge
- Flamingo-Färbung – Proteinmenge / linear / hoch sensitiv
- DeepPurple-Färbung – Proteinmenge
- Diamond ProQ – phosphorylierte Proteine (s. auch Posttranslationale Modifikation)
- Emerald ProQ – glykosylierte Proteine (s. auch Posttranslationale Modifikation)
Durch die zweidimensionale Gelelektrophorese lassen sich in bakteriellen Extrakten nach Färbung der Proteine oft weit über tausend verschiedene Proteinspezies nachweisen. In Mausembryonen ließen sich circa 10.000 Spots darstellen.
2D Gele analysieren und interpretieren
2D Gele digitalisieren
Beim Digitalisieren von 2D-Gelen werden die 2D-Muster in Pixel mit verschiedenen Grauwerten zerlegt. Die Auflösung bestimmt die Genauigkeit in x- und y-Richtung, die Farbtiefe die Menge der Graustufen, welche für die Abbildung der Proteinmenge pro Pixel zur Verfügung steht.
Imaging-Geräte
- Für alle sichtbaren Absorptionsfarbstoffe werden höherwertige Weißlichtscanner mit hohem Probendurchdringungsvermögen (Durchlichtscans mit einer OD bis 4.0) eingesetzt. Ohne eine entsprechende Kalibrierung der Proteinkonzentrationen, Farbstoffsignale und Grauwerte sind quantitative Aussagen jedoch später nur bedingt möglich. Ebenfalls verfügbar für das Digitalisieren der Gele mit sichtbaren Farbstoffen sind inzwischen kamerabasierte Systeme. Farbfilter können u. U. die Ausnutzung des dynamischen Bereiches verbessern helfen.
- Zur Detektion von Fluoreszenzfarbstoffen werden hauptsächlich lichtstarke Laserscanner genutzt, bei denen die Wellenlänge des Anregungslichtes und die Filteroptik für das Abstrahlungslicht der Fluoreszenzfarbstoffe flexibel angepasst werden können. Auch hier werden die etablierten Systeme inzwischen durch kamerabasierte Geräte komplementiert.
- Für den Nachweis von radioaktiven Isotopen, welche in die Proteine eingebaut worden sind, werden sogenannte Phosphorimager eingesetzt. Hier wird eine sogenannte Imaging-Platte dem getrockneten 2D-Gel für mehrere Stunden ausgesetzt. Die freiwerdende radioaktive Strahlung wird in der Imaging-Platte gespeichert und vom Phosphorimager ausgelesen. Die dabei aus der Imaging-Platte freiwerdende gespeicherte Energie wird linear in Graustufen des digitalisierten Bildes übersetzt (s. Autoradiogramm oben).
- Systeme zur Detektion der Eigenfluoreszenz von Proteinen unter UV-Licht durch Anregung aromatischer Aminosäuren wie Tyrosin, Phenylalanin und Tryptophan konnten sich bisher nicht durchsetzen, versprechen aber eine interessante Alternative, da keine teuren Fluoreszenzfarbstoffe eingesetzt werden müssen. Dies würde zu massiver Zeitersparnis führen und darüber hinaus zur Minderung von Diffusions- und anderen Effekten, die durch Färbe- und Waschschritte bedingt werden.
Für weitere Informationen zum Scannen von Gelen kann man eine entsprechende Anleitung (in Englisch) herunterladen.[8]
Single Channel-Techniken
Bei den Single Channel Techniken wird in der Regel eine Serie von Gelen aufgenommen, die vollständig auf die gleiche Art und Weise eingefärbt wurde.
Multiplexing
Beim Gel-Multiplexing werden von ein und demselben Gel mehrere Bilder unabhängig voneinander generiert. Das kann unter verschiedenen Umständen möglich sein:
- Verschiedene Farbstoffe oder Labels werden eingesetzt, die verschiedene Eigenschaften der getrennten Proteine darstellen.
- Beispielsweise können die Proteinmenge durch den Flamingofarbstoff und die Proteinphosphorylierung durch den ProQ-Diamond Farbstoff im selben Gel detektiert werden. Beide Fluoreszenzfarbstoffe werden separat vom Scanner detektiert und in zwei Graustufenbildern gespeichert. Diese können mit einer entsprechenden Imagingsoftware zu einem Falschfarbenbild zusammengesetzt werden.
- Eine weitere Möglichkeit ist die Kombination eines Autoradiogramms (dieses stellt die Proteinsynthese ermittelt durch eine radioaktive Pulsmarkierung dar) und eines Proteinmengenbildes (Detektion der Proteine mit der Silberfärbung). Im dargestellten Falschfarbenbild sind in rot die neu synthetisierten und in grün die akkumulierten Proteine sichtbar.
 Visualisierung von Proteinmenge (grün) und Proteinsynthese (rot) (Software:Delta2D)
Visualisierung von Proteinmenge (grün) und Proteinsynthese (rot) (Software:Delta2D) - Kombinationen von Emerald ProQ Bildern (Färbung von Proteinen mit Zuckermolekülen als Seitenketten), Diamond ProQ Bildern (Färbung der Phosphatreste am Protein) und SyproRuby (Färbung der Proteinmenge) in gleichzeitig drei Farbkanälen kann gleichzeitig Phosphorylierung und Glykosylierung von Proteinen detektieren.
- Alternativ zur unabhängigen Detektion von verschiedenen Proteineigenschaften über verschieden spezifische Farbstoffe bzw. Labels können bei der DIGE bis zu drei Proben gleichzeitig auf einem Gel detektiert werden. Dazu werden die Proteine der drei Proben separat vor dem elektrophoretischen Lauf kovalent mit den Cyanin-Farbstoffen Cy2, Cy3 und Cy5 verknüpft. Danach werden die Proben zusammengefasst und simultan auf dem gleichen Gel getrennt. Die Detektion der Gele erfolgt mit drei Lasern verschiedener Wellenlänge und mit entsprechender Detektionsoptik.
2D Proteinmuster auswerten
Klassischerweise werden 2D-Gele visuell am Licht- oder bei Fluoreszenzfarbstoffen am UV-Tisch ausgewertet. Aufgrund der Komplexität von Proteinmustern führt eine softwaregestützte Analyse zu verlässlicheren Ergebnissen. Für einen Überblick über den aktuellen Stand der Vorgehensweise siehe[9][10]
Gelbilder aufbereiten
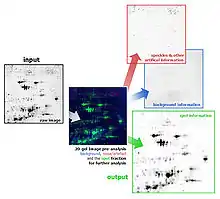
Um Gelbilder quantitativ zu analysieren, sollten sie vom 2D-Gel-typischen inhomogenen Hintergrund befreit und von artifiziellen Signalen bereinigt werden. Das nebenstehende Bild zeigt beispielsweise die Zerlegung eines Gelbildes in eine Hintergrundkomponente, eine Komponente mit artifiziellen Signalen und die für die weiterführende quantitative Analyse genutzte Spot-Komponente.
Gelbilder positionell korrigieren
Ein lange Zeit ungelöstes Problem stellte die schwierige positionelle Reproduzierbarkeit der Proteinmuster im 2D-Gel dar. Eine mögliche Lösung bestand in der Vermeidung unabhängig hergestellter Gele.
- Die DIGE mit der simultanen Trennung von bis zu drei Proben pro Gel ist ein gangbarer Weg, wird allerdings bei mehr als drei Proben wieder mit dem Reproduzierbarkeitsproblem konfrontiert, da dann wiederum mehrere Gele miteinander verglichen werden müssen. Da es bis heute keine befriedigende experimentelle Lösung zur Vermeidung von Laufunterschieden gibt, musste auf Ebene der Softwareanalytik eine Lösung gefunden werden.
- Beim Spot Pattern Matching wird mit Hilfe einer Software im rohen 2D-Gelbild im ersten Schritt nach Proteinspots gesucht. Mit Hilfe der computergenerierten Spot-Information wird versucht, zu einem Expressionsprofil gehörende Partner zu finden (Spot Matching). Schwächen bei der Spotdetektion führen jedoch zu Spotzuordnungsfehlern bei diesen Verfahren.
- Das sogenannte Image Warping (Bildver- bzw. -entzerrung) wurde im Jahr 2000 in die 2D-Gel-Analyse eingeführt. Automatische Verfahren des Image Warpings nutzen die gesamte in den 2D-Gel-Bildern vorhandene Pixel-Information für die Berechnung einer Bildtransformation, die zur bestmöglichen positionellen Übereinstimmung der zu vergleichenden Gelbilder führt. Durch die positionelle Korrektur ist ein aufwendiges Spotmatching nicht mehr nötig, da zueinander gehörige Spots bereits an den gleichen Positionen der zu vergleichenden Gele liegen. Positionell transformierte Gele entsprechen sozusagen idealen Gelen, die keine systembedingten Verzerrungen mehr aufweisen.
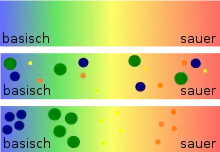
 Image Warping: Zwei Gelbilder wurden in einem Falschfarbenbild kombiniert, das erste Bild orange, das zweite blau. Wegen der Laufunterschiede befinden sich einander entsprechende Spots nicht an der gleichen Position |
 Image Warping: Die gleichen Bilder im Falschfarbenbild und transformiert. Einander entsprechende Spots befinden sich nun an der gleichen Position. Gemeinsame Spots in beiden Mustern sind schwarz, orange Spots sind nur im ersten Bild (verstärkt) vorhanden, blaue Spots nur im zweiten Bild. |
Referenzgele und Proteomkarten
Für eine wissenschaftlich fundierte Interpretation der 2D-Gele wird die Identität der hinter den Proteinspots stehenden Proteine bestimmt. Dies kann über verschiedene Technologien wie beispielsweise den Edman-Abbau oder massenspektrometrische Verfahren wie MALDI-TOF Massenspektrometrie erfolgen. Während in den 1990er Jahren die Proteinspots noch manuell aus den Gelen ausgeschnitten wurden, haben heute Roboter zum Ausstechen der Spots und zum Pipettieren in die Laboratorien Einzug gehalten. Mit Hilfe der Robotertechnik können mehrere hundert Proteine quasi über Nacht identifiziert werden.

Aufgrund der Einführung der computergestützten positionellen Korrektur der Proteinmuster wurde es möglich, Proteinspotidentifikationen problemlos von einem Gel auf ein anderes zu übertragen, ohne nochmals die Spots identifizieren zu müssen. Ein 2D-Gel, welches den Proteinextrakt aus einer Zellkultur zeigt, bildet nur ein Subset aller möglichen Zellproteine ab. Erst Gelserien aus Zellkulturen, die unter verschiedenen Wachstumsbedingungen gezogen wurden, können die Gesamtzahl aller möglichen Proteine zeigen. Grund ist die differentielle Genexpression, die nur die Produktion momentan wichtiger Proteine erlaubt und die Synthese gerade nicht benötigter Proteine verhindert. Zur Konstruktion umfassender Proteomekarten, die einen Großteil aller möglichen Proteine enthalten, werden Gel-Einzelbilder positionell angeglichen und über Bildfusionsalgorithmen zu einem Kompositbild zusammengefasst. Das Kompositbild wird mit den Daten aus den Proteinidentifikationen kombiniert und kann dann als Referenz für die Interpretation weiterer Experimente genutzt werden.
Proteinspots detektieren und quantifizieren
Für eine (semi)quantitative Auswertung der 2D-Gele wird die Gesamt-Absorption (Absorptionsfarbstoffe), das Gesamt-Radiosignal (radioaktiv markierte Proteine, Autoradiogramm) bzw. das Gesamt-Fluoreszenzsignal (Fluoreszenzfarbstoffe) eines Proteinspots über alle Bildpunkte ermittelt. Im ersten Spotdetektionsschritt werden die Koordinaten der Proteinspots und im zweiten die entsprechenden Spotformen bestimmt. Die Bestimmung der Spotumrisse kann nah an der Pixelinformation oder aber über mathematische Modelle erfolgen. Störinformationen, wie z. B. Hintergrund, Artefakte und Bildrauschen (s. Bildvorbereitung) werden vor der Quantifizierung ausgeschlossen. Die Grauwerte der Bildpunkte werden wenn nötig mit geräte- und farbstoffabhängigen Kalibrierungskurven korrigiert und dann innerhalb der gefundenen Spotumrisse zu einer Rohquantität aufsummiert. Die Rohquantitäten werden normiert und den entsprechenden Proteinspots zugeordnet. Da wie weiter oben schon erwähnt die Spotdetektion von Gel zu Gel nicht absolut reproduzierbare Ergebnisse liefert, kann es beim Zuordnen von Proteinen zu ihren Expressionsprofilen zu Irrtümern kommen, die mit den etablierten Methoden manchmal nicht aufgelöst werden können. Darum wurde 2003 eine neue Methode zum Spotmatching eingeführt. Diese beruht auf der Definition eines Spotconsensus aus allen zu analysierenden Gelen eines Experiments auf der Basis eines Kompositbildes. Weil das Kompositbild sämtliche Spotinformationen aus dem Gesamtexperiment enthält, kann der Spotconsensus mindestens auf all jenen Gelen zur Spotquantifizierung angewendet werden, aus denen das Kompositbild erstellt wurde. Spotzuordnungsfehler können bei Anwendung dieser Methode ausgeschlossen werden.
Daten visualisieren

Durch die Anwendung von Proteomekarten und kompositbildbasierter Spotdetektion ergeben sich völlig neue Möglichkeiten der Visualisierung von Proteinspots, die im analysierten Experiment auffällig wurden. Das nebenstehende Bild beispielsweise zeigt eine Zusammenfassung aus vier verschiedenen Proteinsynthesemustern. Die Farben zeigen an, unter welchen Umweltbedingungen welches Protein in seiner Synthese mindestens zweifach erhöht wird. Mit Hilfe derartiger Farbcodierungen wird es erstmals möglich, neben den positionellen Daten der Proteinspots nun auch Regulationsdaten zu visualisieren. Biomarker sind somit schnell und zuverlässig identifizierbar.
Abwandlungen der klassischen 2D-Gele
Chemie des Trennsystems
Während die klassischen 2D-Gele Proteine in der 1. Dimension nach ihrem isoelektrischen Punkt und in der 2. Dimension nach ihrer Größe trennen, werden in der Praxis für Proteine durchaus auch andere Trennmethoden in zwei Dimensionen kombiniert. Beispiele sind:
- die Kombination von zwei verschiedene Detergenzien in den beiden Dimensionen.
- "Blue native" Electrophorese in der ersten und SDS Gel Electrophorese in der zweiten Dimension
- Trennung unter oxischen (erste Dimension) und reduzierenden (zweite Dimension) Bedingungen
Geometrie des Trennsystems
- Die Trennung der Proteine nach dem Isoelektrischen Punkt erfolgt meist horizontal in Gelstreifen oder auch vertikal in langen zylindrischen Gelen, welche in Röhren hergestellt wurden.
- Die zweite Trennung wird durchgeführt in Sandwiches aus 2 Glasplatten mit dazwischenliegendem Gel. Diese Glasplatten-Gel-Sandwiches werden vertikal in Stapeln von 1, 2, 5, 6, 10 oder 12 Gelen in Tanks eingehängt, die den Laufpuffer enthalten und während der Trennung durch einen Kryostaten gekühlt werden.
- Kommerziell verfügbare Fertiggele werden oft in Horizontalsystemen auf Kühlplatten zur Proteintrennung eingesetzt. Je nach Dimensionierung können auch hier 1 bis 4 oder mehr Gele parallel verarbeitet werden.
- Neu ist beispielsweise der Einsatz von radialsymmetrischen Apparaturen, welche die IEF-Streifen in der 2. Dimension von innen nach außen in kreisförmigen Gelscheiben auftrennen. Je nach Länge der IEF-Gele können bis zu sechs Ansätze auf einer Gelscheibe aufgetrennt werden. Es ist vorgesehen, mehrere solcher Disks in einer Anlage zu stapeln. Da das elektrische Feld hier von innen nach außen verläuft, entfernen sich die Feldlinien voneinander, je weiter die Proteine nach außen wandern. Damit wird verhindert, dass sich insbesondere kleine Proteine zu schnell bewegen und das Gel wieder verlassen, noch bevor die großen langsamen Proteine ausreichend aufgetrennt wurden.
- Um die parallele Auftrennung von Proben auf die Spitze zu treiben, wurden geometrisch dreidimensionale Gele entwickelt (www.3d-gel.com), welche simultan 36 IEF-Gele auf einem Gelblock trennen können. Die Detektion der Proteine erfolgt hier wie bei traditionellen DNA-Sequenzierern in einer durch einen Laser illuminierten Gelebene. Bei der Passage der vor der Trennung mit Fluoreszenzfarbstoffen markierten Proteine wird Emissionslicht detektiert. Die gemessenen Signale werden gespeichert. Die Gelbilder werden aus dem gespeicherten Bilderstapel durch eine Software rekonstruiert und konventionell ausgewertet.
Vorteile von 2D-Gelen
- Die Visualisierung des Proteinmusters erlaubt bereits ohne weitergehende Analyse eine qualitative Einschätzung der Ergebnisse
- Verschiedene Proteinspezies ein und desselben Primärproteins sind ohne Vorbedingung einer Analyse sofort zugänglich, soweit sie hinreichend unterschiedliche Massen und/oder isoelektrische Punkte haben.
- Die Methode ist hoch parallelisierbar. Je nach 2D-Gelelektrophoresegerät können bis zu 12 Gele gleichzeitig produziert werden.
- Die nötigen Investitionen sind gegenüber gelfreien Methoden relativ überschaubar.
- Das separierte Gel kann längere Zeit gelagert und später weiterverarbeitet werden.
- Inzwischen stehen Analysesysteme zur Verfügung, die in kurzer Zeit und ohne erweiterte Computerkenntnisse eine Vielzahl von Proben vergleichen können.
Probleme der 2D-Gel-Elektrophorese
Wie jede andere Technik in der Proteinbiochemie birgt auch die 2D-Gel-Elektrophorese einige Probleme:
- Aufgrund des Trennsystems im wässrigen Milieu werden hauptsächlich hydrophile Proteine mit einem GRAVY Index kleiner und nahe 1 aufgetrennt.
- Trotz des großen Separationspotenzials können starke Proteinspots durchaus schwache Spots überlappen bzw. völlig maskieren. Diese maskierten Spots sind einer quantitativen Analyse nicht mehr zugänglich.
- Durch die begrenzte Sensitivität der Coomassie- bzw. Silberfärbung einerseits und die begrenzte Kapazität eines 2D-Geles andererseits ist die Sensitivität der Methodik eingeschränkt. 2D-Gele können daher nur die abundantesten Proteine einer Zelle darstellen (z. B. Zytoskelettproteine, metabolische Enzyme), während gering exprimierte Proteine wie (z. B. Transkriptionsfaktoren) meist unerkannt bleiben.
- Zu große Gesamtproteinmengen führen zu Diskriminierungsphänomenen beim Eintritt verschiedener Proteinspezies in das IEF-Gel und somit zu einer Verzerrung der Proteinspezies-Verhältnisse.
- Je basischer ein Protein ist, desto schwerer wird dessen Separation. Ferner ist das 2D-Gelsystem hauptsächlich in einem pH-Bereich von 3 bis 10 effizient.
- Die 2D-Gele haben eine begrenzte Reproduzierbarkeit, die vom Hersteller und Anwender sowie von den verwendeten Gelbestandteilen und Elektrophoresepufferzusammensetzungen abhängt.
Siehe auch
Einzelnachweise
- P. H. O'Farrell: High resolution two-dimensional electrophoresis of proteins. In: J. Biol. Chem. 250, 1975, S. 4007–4021. PMID 236308; PDF (freier Volltextzugriff)
- J. Klose: Protein mapping by combined isoelectric focusing and electrophoresis in mouse tissues. A novel approach to testing for induced point mutations in mammals. In: Humangenetik. 26, 1975, S. 231–243. PMID 1093965
- Thierry Rabilloud: Two-Dimensional Electrophoresis Protocols. Humana Springer, 2009, ISBN 978-1-58829-937-6. Kapitel 2: Solubilization of proteins in 2DE: An outline (PDF; 174 kB)
- A. Görg, C. Obermaier, G. Boguth, A. Harder, B. Scheibe, R. Wildgruber, W. Weiss: The current state of two-dimensional electrophoresis with immobilized pH gradients. In: Electrophoresis. 21(6), Apr 2000, S. 1037–1053. PMID 10786879
- R. Westermeier, W. Postel, J. Weser, A. Görg: High-resolution two-dimensional electrophoresis with isoelectric focusing in immobilized pH gradients. In: J Biochem Biophys Methods. 8(4), Dez 1983, S. 321–330. PMID 6663005
- Joachim Hartinger, Katinka Stenius, Dagmar Högemann, Reinhard Jahn: 16-BAC/SDS-PAGE: a two-dimensional gel electrophoresis system suitable for the separation of integral membrane Proteins. In: Analytical Biochemistry. Band 240, Nr. 1, 1996, S. 126–133. PMID 8811889, doi:10.1006/abio.1996.0339.
- R. P. Zahedi, J. Moebius, A. Sickmann: Two-dimensional BAC/SDS-PAGE for membrane proteomics. In: Sub-cellular biochemistry. Band 43, 2007, S. 13–20. PMID 17953388.
- decodon.com: Imaging Guide (englisch)
- M. Berth, F. M. Moser, M. Kolbe u. a.: The state of the art in the analysis of two-dimensional gel electrophoresis images. In: Appl Microbiol Biotechnol. 76(6), 2007, S. 1223–1243, PMC 2279157 (freier Volltext)
- J. E. Bandow, J. D. Baker, M. Berth, C. Painter, O. J. Sepulveda, K. A. Clark, I. Kilty, R. A. VanBogelen: Improved image analysis workflow for 2-D gels enables large-scale 2-D gel-based proteomics studies–COPD biomarker discovery study. In: Proteomics. Band 8, Nummer 15, August 2008, S. 3030–3041, ISSN 1615-9861. doi:10.1002/pmic.200701184. PMID 18618493.
Literatur
- R. C. Allen, B. Budowle: Gel Electrophoresis of Proteins and Nucleic Acids: Selected Techniques. Walter de Gruyter, 1994, ISBN 3-11-013896-4.
- J. E. Bandow, J. D. Baker, M. Berth, C. Painter, O. J. Sepulveda, K. A. Clark, I. Kilty, R. A. VanBogelen: Improved image analysis workflow for 2-D gels enables large-scale 2-D gel-based proteomics studies–COPD biomarker discovery study. In: Proteomics. Band 8, Nummer 15, August 2008, S. 3030–3041, ISSN 1615-9861. doi:10.1002/pmic.200701184. PMID 18618493.
- M. Berth, F. M. Moser, M. Kolbe u. a.: The state of the art in the analysis of two-dimensional gel electrophoresis images. In: Appl Microbiol Biotechnol. 76(6), 2007, S. 1223–1243, PMC 2279157 (freier Volltext)
- M. H. Hamdan, P. G. Righetti: Proteomics Today: Protein Assessment and Biomarkers Using Mass Spectrometry, 2D Electrophoresis,and Microarray Technology. Wiley-Interscience, 2005, ISBN 0-471-64817-5.
- T. Rabilloud (Hrsg.): Proteome Research: Two-Dimensional Gel Electrophoresis and Identification Methods (Principles and Practice). Springer, 1999, ISBN 3-540-65792-4.
- Sabine Schmitz: Der Experimentator: Zellkultur. 1. Auflage. Spektrum Akademischer Verlag, 2007, ISBN 978-3-8274-1564-6.