Sortase
Sortasen bilden eine Familie Membran-assoziierter bakterieller Enzyme und werden der Klasse der Transpeptidasen zugeordnet.[1]
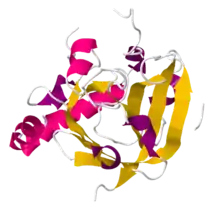
Eigenschaften
Die biologische Funktion der Sortasen besteht darin, sezernierte Proteine in der Peptidoglycanschicht grampositiver Bakterien kovalent zu verankern.[2] Zusätzlich gibt es Sortasen, die für den Aufbau von Pilusstrukturen aus einzeln exprimierten Proteinuntereinheiten verantwortlich sind.[3] Daher sind Sortasen in hohem Maße verantwortlich für den strukturellen Aufbau der Zelloberfläche grampositiver Bakterien.[4]
Da sich auf der bakteriellen Oberfläche viele Virulenzfaktoren befinden, werden Sortasen als mögliches therapeutisches Ziel bei der Behandlung von Infektionskrankheiten diskutiert.[2][5]
Der Sortase-Reaktionsmechanismus kann in vitro genutzt werden, um Proteine (auch andere Moleküle) kovalent und ortsspezifisch miteinander zu verknüpfen, weshalb die Enzyme auch in der Biotechnologie Anwendung finden. Dabei ist es von Vorteil, dass funktionsfähige Sortasen heterolog (z. B. in E. coli) exprimiert werden können, was eine Herstellung in größerem Maßstab mit relativ geringem Aufwand ermöglicht.[6]
Sortasen benötigen zweiwertige Kationen (z. B. Ca2+) als Cofaktoren.[7]
Vorkommen und Einteilung
Sortasen werden in nahezu allen grampositiven Bakterien gefunden, sind aber auch vereinzelt in gramnegativen und Archaeen nachweisbar.[8] Bis jetzt wurden über 800 Gene in mehr als 260 verschiedenen Spezies identifiziert, die für sortaseverwandte Proteine codieren. Dabei können circa 60 % dieser Proteine in insgesamt sechs verschiedene Gruppen (A–F) eingeteilt werden.
Die einzelnen Gruppen werden dabei nach ihrer Funktion und ihrer Substratspezifität unterschieden. Während die Gruppen A und B hauptsächlich die Funktion haben Proteine in der Peptidoglycanschicht der Bakterien zu verankern, sind Sortasen der Gruppen C und D am Aufbau von Pilusstrukturen beteiligt.[9][3] Des Weiteren kommen die Sortasen der Gruppe D häufig in Bakterien der Gattung Bacillus vor, in welchen sie mit der Bildung von Endosporen in Verbindung stehen.[10] Die Funktionen der Sortasen E und F sind noch nicht vollständig geklärt.[9]
In der Regel besitzen grampositive Bakterien mehr als einen Typ von Sortasen.[8]
Reaktionsmechanismus
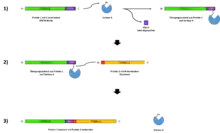
Grundsätzlich sind Sortasen dazu in der Lage, zwei Peptidreste kovalent miteinander zu verknüpfen. Dafür benötigen sie ein spezifisches Erkennungsmotiv, welches häufig aus fünf Aminosäuren besteht. Sortasen können diese Erkennungsmotive innerhalb eines Proteins spalten und anschließend mit einem anderen Liganden wieder zusammensetzen. Die Erkennungsmotive können sich dabei, je nach Sortasetyp und Organismus, unterscheiden.
| Organismus | Sortasetyp | Erkennungsmotiv |
|---|---|---|
| Staphylococcus aureus | Sortase A | LPXTG |
| Staphylococcus aureus | Sortase B | NPQTN |
| Streptococcus pneumoniae | Sortase B | YPRTG |
In-vitro-Reaktion
Im folgenden Abschnitt wird der Sortasereaktionsmechanismus beispielhaft anhand der gut charakterisierten Sortase A aus Staphylococcus aureus dargestellt. Das Erkennungsmotiv dieser Sortase lautet LPXTG (Leucin-Prolin-Xaa-Threonin-Glycin). Dabei steht X für eine beliebige Aminosäure.
Die Reaktion erfolgt in zwei Schritten. Zuerst bindet die Sortase A an das Protein/Peptid. Die kovalente Bindung erfolgt zwischen dem Thiolrest eines Cysteins im aktiven Zentrum der Sortase A und dem Threonin innerhalb der LPXTG-Erkennungssequenz. Dadurch wird der C-terminale Rest des Proteins, inklusive des Glycins, abgespalten. Es entsteht ein Übergangszustand, in welchem die Sortase A kovalent mit dem Proteinrest verbunden ist. Im zweiten Schritt trifft dieser Übergangszustand auf ein Protein/Peptid mit freiem, N-terminalem, Glycinrest. Dabei werden die beiden Proteine durch die Sortase A kovalent miteinander verbunden, wodurch die ursprüngliche Peptidbindung (das LPXTG-Motiv) wieder hergestellt wird.[11]
In-vivo-Funktion der Sortase A von Staphylococcus aureus
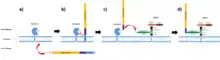
Proteine, die für den Einbau in die Zellwand von S. aureus vorgesehen sind, besitzen mehrere funktionelle Untereinheiten. Dazu zählt ein N-terminales Signalpeptid (SP), das dafür verantwortlich ist, dass das Protein aus dem Cytoplasma – durch die Membran hindurch – in den Extrazellulärraum sezerniert wird. Dabei wird das Signalpeptid entfernt.
Durch eine C-terminale hydrophobe Region (HR) wird das sezernierte Protein anschließend in der Zellmembran befestigt. Das nun membran-assoziierte Protein trägt das LPXTG-Erkennungsmotiv, an welches die ebenfalls membran-assoziierte Sortase bindet. Durch die Bindung der Sortase an das LPXTG-Motiv wird die hydrophobe Region abgespalten und ein Übergangszustand entsteht, in welchem die Sortase mit dem Protein verbunden ist.
Durch die Reaktion des Übergangszustandes mit dem freien Pentaglycinrest von Lipid II (einer Vorstufe der Peptidoglycanbausteine), wird das sezernierte Protein kovalent mit dem Zellwandbaustein (Lipid II) verankert. Durch den weiteren Verlauf der Zellwandsynthese wird das Protein somit Bestandteil der Peptidoglycanschicht.[4]
Anwendungen

Sortasen werden in der Biotechnologie verwendet, um Moleküle miteinander zu verbinden und damit ihre Funktion räumlich zu koppeln. Dabei können nicht nur Proteine verknüpft werden, sondern auch Lipide und andere Moleküle. Voraussetzung ist, dass die Erkennungssequenzen für die Sortase an den Molekülen angebracht werden können. Die Motive müssen außerdem korrekt orientiert und für die Sortase räumlich zugänglich sein.[12]
Proteine oder auch andere Moleküle können mit Hilfe von Sortasen für analytische Zwecke markiert werden. Beispielsweise können Antikörper oder Rezeptorliganden mit einem Fluoreszenzfarbstoff markiert werden um deren Bindungseigenschaften zu untersuchen.[13]
Mit Hilfe des Sortasereaktionsmechanismus können therapeutische Moleküle PEGyliert werden. Durch die Verbindung eines therapeutischen Proteins mit Polyethylenglycol (PEG) kann der Abbau des Proteins im Körper verlangsamt werden. Mit der längeren biologischen Halbwertszeit des therapeutischen Proteines werden die pharmakodynamischen Eigenschaften verbessert.[14]
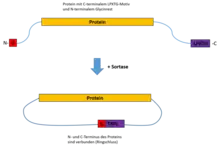
Durch das Verbinden des C- und N-Terminus eines Proteins wird ein Ringschluss erreicht. Damit wird der Abbau therapeutischer Proteine durch Proteasen gehemmt. Dies führt, wie die PEGylierung, zu einer längeren biologischen Halbwertszeit des Proteins.[14]
Die Verwendung von mehreren Sortasen mit unterschiedlichen Erkennungsmotiven erlaubt die Synthese eines Komplexes aus mehreren Liganden. Dabei lässt sich die Orientierung und Reihenfolge der einzelnen Liganden steuern.[15]
Vorteile der Nutzung von Sortasen
Sortasen sind Enzyme und funktionieren unter physiologischen Bedingungen. Daher können Sortasen, im Gegensatz zu manchen chemischen Verfahren, eingesetzt werden, ohne dass eine Schädigung der Substrate (z. B. durch Temperatur, pH-Wert oder Salzkonzentration) zu erwarten ist.
Die Reaktion der Sortasen ist immer spezifisch, was bedeutet, dass die Ligation der Substrate immer an derselben Stelle erfolgt. Bei der chemischen Synthese hingegen kann die Verknüpfung der Substrate unter Umständen an verschiedenen Stellen erfolgen.
Nachteile der Nutzung von Sortasen
Sortasen katalysieren die Hin- und Rückreaktion. Das bedeutet, sie katalysieren sowohl die Verknüpfung der Substrate, sowie auch das Lösen der Bindung. Der Grund dafür ist, dass durch die Verknüpfung das Erkennungsmotiv der Sortase wiederhergestellt wird. Dadurch bildet sich nach einer gewissen Zeit ein Reaktionsgleichgewicht zwischen den Substraten und dem Produkt. Die Produktausbeute der Sortasereaktion ist von den Eigenschaften der Substrate abhängig und in der Regel erfolgt der Umsatz nicht vollständig.
Auch die Aufreinigung des Produkts aus dem Reaktionsansatz kann problematisch sein. Dies hängt jedoch auch stark von den eingesetzten Substraten ab und ist deshalb nicht pauschal zu beurteilen.
Sortasen als Ziel therapeutischer Anwendungen
Sortasen sind in hohem Maße für die Architektur der Zellwand grampositiver Bakterien verantwortlich. Zu den Oberflächenstrukturen die von Sortasen aufgebaut werden zählen auch viele Virulenzfaktoren, welche z. B. für den Stoffwechsel der Bakterien[16] oder für die Adhäsion an menschliches Gewebe nötig sind.[3] Untersuchungen haben gezeigt, dass Krankheitserreger deren Sortasen genetisch inaktiviert wurden, nicht mehr fähig sind Infektionen auszulösen. Daraus schließt man, dass eine medikamentöse Inhibition der Sortasen eine antibiotische Therapie darstellen könnte.[17]
Einzelnachweise
- S. K. Mazmanian, G. Liu, E. R. Jensen, E. Lenoy, O. Schneewind: Staphylococcus aureus sortase mutants defective in the display of surface proteins and in the pathogenesis of animal infections. In: Proceedings of the National Academy of Sciences. Band 97, Nummer 10, Mai 2000, ISSN 0027-8424, S. 5510–5515, doi:10.1073/pnas.080520697, PMID 10805806, PMC 25859 (freier Volltext).
- Ki-Bong Oh et al.: Inhibition of sortase-mediated Staphylococcus aureus adhesion to fibronectin via fibronectin-binding protein by sortase inhibitors. In: Applied Microbiology and Biotechnology. 70, Nr. 1 2006, S. 102–106.
- Markus Hilleringmann et al.: Molecular architecture of Streptococcus pneumoniae TIGR4 pili. In: The EMBO Journal. 28, Nr. 24 2009, S. 3921–3930.
- Luciano A. Marraffini, Andrea C. Dedent und Olaf Schneewind: Sortases and the art of anchoring proteins to the envelopes of gram-positive bacteria. In: Microbiology and molecular biology reviews : MMBR. 70, Nr. 1 2006, S. 192–221.
- Stella Cascioferro, Makrina Totsika und Domenico Schillaci: Sortase A: an ideal target for anti-virulence drug development. In: Microbial pathogenesis.77 2014, S. 105–112.
- C. S. Theile, M. D. Witte, A. E. Blom, L. Kundrat, H. L. Ploegh, C. P. Guimaraes: Site-specific N-terminal labeling of proteins using sortase-mediated reactions. In: Nature protocols. Band 8, Nummer 9, September 2013, ISSN 1750-2799, S. 1800–1807, doi:10.1038/nprot.2013.102, PMID 23989674, PMC 3941705 (freier Volltext).
- U. Ilangovan et al.: Structure of sortase, the transpeptidase that anchors proteins to the cell wall of Staphylococcus aureus. In: Proceedings of the National Academy of Sciences.98, Nr. 11 2001, S. 6056–6061.
- M. J. Pallen et al.: An embarrassment of sortases – a richness of substrates? In: Trends in microbiology. 9, Nr. 3 2001, S. 97–102.
- Thomas Spirig, Ethan M. Weiner und Robert T. Clubb: Sortase enzymes in Gram-positive bacteria. In: Molecular microbiology. 82, Nr. 5 2011, S. 1044–1059.
- Jonathan M. Budzik, Luciano A. Marraffini und Olaf Schneewind: Assembly of pili on the surface of Bacillus cereus vegetative cells. In: Molecular microbiology. 66, Nr. 2 2007, S. 495–510.
- Gavin K. Paterson und Timothy J. Mitchell: The biology of Gram-positive sortase enzymes. In: Trends in microbiology. 12, Nr. 2 2004, S. 89–95.
- John M. Antos et al.: Lipid modification of proteins through sortase-catalyzed transpeptidation. In: Journal of the American Chemical Society. 130, Nr. 48 2008, S. 16338–16343.
- Maximilian W. Popp et al.: Sortagging: a versatile method for protein labeling. In: Nature Chemical Biology.3, Nr. 11 2007, S. 707–708
- Maximilian W. Popp et al.: Sortase-catalyzed transformations that improve the properties of cytokines. In: Proceedings of the National Academy of Sciences. 108, Nr. 8 2011, S. 3169–3174.
- John M. Antos et al.: Site-specific N- and C-terminal labeling of a single polypeptide using sortases of different specificity. In: Journal of the American Chemical Society. 131, Nr. 31 2009, S. 10800–10801.
- Sarkis K. Mazmanian et al.: An iron-regulated sortase anchors a class of surface protein during Staphylococcus aureus pathogenesis. In: Proceedings of the National Academy of Sciences. 99, Nr. 4 2002, S. 2293–2298.
- Anthony W. Maresso und Olaf Schneewind: Sortase as a target of anti-infective therapy. In: Pharmacological Reviews. 60, Nr. 1 2008, S. 128–141.