In-situ-Hybridisierung
In-situ-Hybridisierung (ISH, auch Hybridisierung in situ) ist eine molekularbiologische Methode zum Nachweis von Nukleinsäuren (RNA oder DNA) in Geweben, einzelnen Zellen oder auf Metaphase-Chromosomen. Dabei wird eine künstlich hergestellte Sonde aus einer Nukleinsäure eingesetzt, die über Basenpaarungen an die nachzuweisende Nukleinsäure bindet (Hybridisierung).
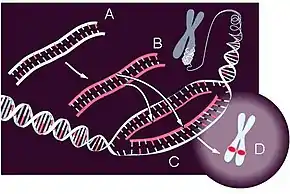
Weite Verbreitung haben die Fluoreszenz-in-situ-Hybridisierung (FISH) für den Nachweis von DNA oder RNA (RNA-FISH) in Zellkernen einzelner Zellen oder auf Metaphase-Chromosomen sowie die Untersuchung der Verteilung von mRNA in ganzen Embryonen, Schnitten oder Geweben mit farbgebenden (chromogenen) Molekülen (FISH-Test).
Weitere verwendete Bezeichnungen sind genomische In-situ-Hybridisierung (GISH), bei der als Sonde gesamtgenomische DNA eingesetzt wird, sowie chromosomale In-situ-Suppressionshybridisierung (CISS-Hybridisierung), mit der die Markierung von ganzen Chromosomen möglich ist.
Geschichte
In-situ-Hybridisierung wurde Ende der 1960er Jahre von den US-amerikanischen Biologen Mary Lou Pardue und Joe Gall entwickelt.[1][2] Sie verwendeten radioaktiv markierte Sonden (Tracer), die Präparate mussten anschließend auf einen Röntgenfilm gelegt werden. Neben den allgemeinen mit Radioaktivität verbundenen Problemen war die räumliche Auflösung dabei verglichen mit heutigen Techniken schlecht. Daher setzten sich später Sonden durch, die kovalent an die Markierungsmoleküle gebunden waren (siehe unten).
Funktionsweise
Die In-situ-Hybridisierung beruht auf der Paarung von komplementären Basen auf zwei Nukleinsäure-Einzelsträngen. Einer der beiden Stränge kommt dabei von einer zuvor hergestellten und markierten Sonde, der andere liegt im Präparat vor und soll nachgewiesen werden. Die ersten Arbeitsschritte sind demnach Vorbereitung des Präparats sowie Vorbereitung der Sonde. Die Sonde besteht meist aus DNA, da diese stabiler ist als RNA und somit im Labor einfacher zu handhaben ist. Auch lässt sich DNA mit heutigen Verfahren einfacher vermehren. Die Markierung der Sonde kann indirekt mit Haptenen (z. B. Digoxigenin, Biotin oder 2,4-Dinitrophenol) oder bei der FISH auch direkt mit fluoreszierenden Molekülen wie FITC oder Cy3 erfolgen. Häufig eingesetzte Verfahren zur Markierung der Sonden sind Nick-Translation, PCR und In-vitro-Transkription.
Da DNA normalerweise als Doppelstrang vorliegt, muss dieser zuvor getrennt (auch: aufgeschmolzen oder denaturiert) werden. Eine Denaturierung kann entweder durch Verschiebung des pH-Werts (sowohl sauer als auch alkalisch) oder durch Hitze erfolgen. Bei einer Hitzedenaturierung wird der Schmelzpunkt meist durch Zugabe von Formamid abgesenkt, welches durch seine starke Polarität die Wasserstoffbrückenbindungen zwischen den DNA-Einzelsträngen abschwächt. Dadurch kann ein Aufschmelzen bereits bei Temperaturen um 70–75 °C erreicht werden, wodurch die Struktur der Chromosomen weniger stark zerstört wird. Weiterhin kann die DNA auch durch ein Molecular Combing gestreckt und ausgerichtet werden.
Die eigentliche Hybridisierung dauert, je nach Sondenmaterial und Zielsequenz, zwischen einer Stunde und mehreren Tagen. Anschließend liegen im Präparat Hybridmoleküle aus den Nukleinsäuren des Präparats und der Sonde vor. Nicht oder unspezifisch gebundene Sondenmoleküle werden herausgewaschen und gebundene Sondenmoleküle können nachgewiesen werden. Bei der indirekten Markierung geschieht dies über eine Antikörperfärbung oder im Fall von Biotin mit Avidin. Die Antikörper (oder das Avidin) sind wiederum an Fluorophore (bei der FISH) oder an Enzyme gebunden, welche aus zuzugebenden chromogenen Substraten Farbstoffe bilden. Beides wird anschließend mikroskopisch ausgewertet.
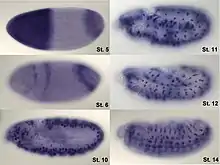 Beispiel für eine RNA-in-situ-Hybridisierung an verschiedenen Stadien von Drosophila melanogaster-Embryonen. Die Digoxigenin-markierte antisense-RNA-Sonde ist gegen die mRNA des Transkriptionsfaktors hunchback gerichtet. Sie wurde mit einer Färbereaktion mit Alkalischer Phosphatase sichtbar gemacht. Es ist deutlich erkennbar, dass sich das Expressionsmuster des Gens in den verschiedenen Entwicklungsstadien (St. steht abgekürzt für Stadium) verändert. |
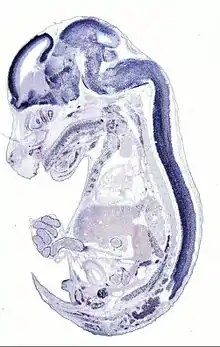 Die Aktivität eines Gens in einem Mausembryo des Embryonalstadiums E14,5 (nach 14,5 von insgesamt 19,5 Tagen der Schwangerschaft). Durch In-situ-Hybridisierung wurde in einem 25 Mikrometer dünnen Gefrierschnitt das Vorhandensein der mRNA und somit die Aktivität des Gens Cannabinoid-Rezeptor Typ 1 (Cnr1) sichtbar gemacht, die als dunkelblaue Färbung u. a. in verschiedenen Gehirnbereichen und im Rückenmark zu erkennen ist. |
Nachweis von mRNA mit farbgebenden Enzymen
Bei dieser Anwendung findet vor allem Digoxigenin-markierte RNA Verwendung. Das Digoxigenin kann mit Hilfe eines Antikörpers, der beispielsweise mit einem Enzym gekoppelt ist, erkannt werden. Das Enzym, meistens Alkalische Phosphatase oder Peroxidase, kann dann durch Zusatz von Reagenzien einen Farbstoff umsetzen, der kovalent im Gewebe gebunden bleibt und sich daher nicht durch Diffusion verteilt. Beim Nachweis von mRNA in Geweben werden nur jene Zellen angefärbt (hybridisiert), in denen ein zu untersuchendes Gen aktiv ist: Nur hier liegt die entsprechende mRNA vor. Dieses Verfahren findet besonders in der Entwicklungsbiologie Anwendung. Hier ist es von besonderem Interesse, die Aktivität eines Gens beispielsweise während der Embryogenese in situ zu verfolgen. Das embryonale oder auch adulte Gewebe muss für die Färbung zunächst fixiert werden; die Aktivität kann daher nicht in Echtzeit verfolgt werden, sondern ist nur eine Momentaufnahme des Zustands, in dem sich das Gewebe befand, als es fixiert wurde.
Ablauf
Das zu färbende Gewebe – beispielsweise Embryonen von verschiedenen Modellorganismen wie Arabidopsis thaliana, Drosophila melanogaster, Xenopus laevis, Mus musculus, Danio rerio – wird mit Hilfe von formaldehydhaltigen Lösungen fixiert. Anschließend wird es in einen formamidhaltigen Puffer überführt und die markierte Sonde dazugegeben. Die Hybridisierungszeit hängt von der Größe des Embryos ab und dauert mindestens einige Stunden. In dieser Zeit kann die Sonde durch das Gewebe diffundieren und bindet überall dort, wo sich komplementäre Sequenzen in der mRNA finden. Es folgen einige Waschschritte, in denen überschüssige, nicht gebundene Sonden ausgewaschen werden, und am Schluss die Färbung, die dann mit einem Mikroskop genau analysiert und dokumentiert werden kann.
Neben Totalpräparaten, beispielsweise von Fliegenembryonen (linke Abbildung), kann die In-situ-Hybridisierung an Gewebe-Dünnschnitten durchgeführt werden. Auf diese Weise können auch größere Präparate wie menschliches Gewebe oder komplette Mausembryonen untersucht werden (rechte Abbildung).
Fluoreszenz-in-situ-Hybridisierung
Bei der Fluoreszenz-in-situ-Hybridisierung (FISH) wird die Sonde mit Hilfe eines fluoreszierenden Farbstoffes nachgewiesen. Dies ermöglicht den gleichzeitigen Nachweis mehrerer Strukturen durch den Einsatz verschiedener Fluoreszenzfarbstoffe, zum Beispiel den Nachweis eines Chromosoms mit zwei darauf liegenden Genen. Auf Chromosomenpräparaten und auf Zellkernen konnten bereits bis zu sieben verschiedene Farbstoffe erfolgreich eingesetzt werden.[3]
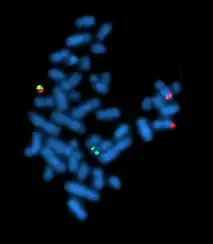 Metaphasechromosomen (blau) mit Nachweis einer Translokation durch den Einsatz zweier genspezifischer Sonden (grün und rot), siehe Philadelphia-Chromosom
Metaphasechromosomen (blau) mit Nachweis einer Translokation durch den Einsatz zweier genspezifischer Sonden (grün und rot), siehe Philadelphia-Chromosom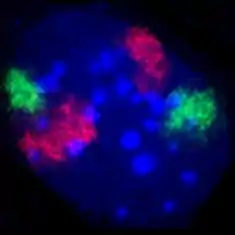 Zellkern eines Fibroblasten der Maus. Durch Fluoreszenz-in-situ-Hybridisierung wurden die Territorien der Chromosomen 2 (rot) und 9 (grün) angefärbt. DNA-Gegenfärbung in Blau. (Hier ist eine Tafel mit weiteren Beispielen auch aus anderen Zelltypen der Maus zu finden.)
Zellkern eines Fibroblasten der Maus. Durch Fluoreszenz-in-situ-Hybridisierung wurden die Territorien der Chromosomen 2 (rot) und 9 (grün) angefärbt. DNA-Gegenfärbung in Blau. (Hier ist eine Tafel mit weiteren Beispielen auch aus anderen Zelltypen der Maus zu finden.) Genreiche und genarme Regionen bei menschlichen Chromosomen. Auf Metaphasechromosomen eines menschlichen weiblichen Lymphozyten wurden per Fluoreszenz-in-situ-Hybridisierung die Alu-Sequenzen markiert (grün), die besonders in genreichen Chromosomenabschnitten häufig vorkommen. Zur Sichtbarkeit genarmer Regionen ist die DNA rot eingefärbt.
Genreiche und genarme Regionen bei menschlichen Chromosomen. Auf Metaphasechromosomen eines menschlichen weiblichen Lymphozyten wurden per Fluoreszenz-in-situ-Hybridisierung die Alu-Sequenzen markiert (grün), die besonders in genreichen Chromosomenabschnitten häufig vorkommen. Zur Sichtbarkeit genarmer Regionen ist die DNA rot eingefärbt.
Anwendungsgebiete
Die Möglichkeit, das Vorhandensein bzw. die Anzahl von Teilen des Genoms vergleichsweise einfach im Fluoreszenzmikroskop zu bestimmen, wird für die klinische Diagnostik in verschiedenen Disziplinen benutzt.
Für den Nachweis von Erbkrankheiten werden in der Regel Präparate von Chromosomen in der Metaphase eingesetzt, die aus im Labor vermehrten Zellen von Patienten oder, bei der Pränataldiagnostik, aus Zellen des ungeborenen Kindes hergestellt wurden (siehe auch FISH-Test). Auf diese werden dann beispielsweise Sonden für das Chromosom 21 hybridisiert, um zu testen, ob es drei- statt zweimal vorhanden ist und somit die Ursache für das Down-Syndrom vorliegt. Ob bestimmte chromosomale Regionen fehlen, wie etwa beim Katzenschrei-Syndrom, lässt sich mit Sonden für solche Regionen ebenfalls feststellen. Prinzipiell lassen sich solche Untersuchungen auch an Zellkernen durchführen, also ohne dass Metaphasepräparate hergestellt werden müssen. Dies hat jedoch den Nachteil, dass nicht jeder Kern ein richtiges Ergebnis liefert: Wenn zwei nachzuweisende Chromosomen im Kern zufällig nebeneinander liegen, dann lassen sich beide nicht als zwei Signale erkennen. Es müssen daher viele Kerne ausgezählt werden und ein Vergleich mit bekanntermaßen normalen Kernen muss durchgeführt werden, um eine sichere Aussage treffen zu können. Der Vorteil dieses Verfahrens liegt in der höheren Geschwindigkeit, da eine Vermehrung der Zellen im Labor nicht oder nur in geringerem Umfang nötig ist. Auch bei der Charakterisierung von Markerchromosomen wird FISH eingesetzt.[4]
Bei der Untersuchung des Genoms von Tumor-Zellen wird FISH eingesetzt, um Chromosomenaberrationen festzustellen. Beispielsweise können Metaphasepräparate mit FISH untersucht werden, um chromosomale Umbauten zu erkennen, die sich durch G-Bänderung nicht diagnostizieren lassen. Hierfür können Sonden für verschiedene menschliche Chromosomen der Reihe nach ausprobiert werden. Vielfarben-FISH erlaubt es auch, sämtliche Chromosomen in unterschiedlichen Farbkombinationen nachzuweisen, so dass sämtliche Umbauten zwischen verschiedenen Chromosomen in einem Experiment erkannt werden können.[5][6] Die hierfür erforderlichen Methoden sind jedoch anspruchsvoll und erfordern eine spezielle mikroskopische Ausstattung. Manche chromosomale Umbauten treten bei bestimmten Tumorarten gehäuft auf, beispielsweise das Philadelphia-Chromosom bei manchen Leukämien. Bei Non-Hodgkin-Lymphomen haben verschiedene Untergruppen unterschiedliche typische genetische Veränderungen. Das Vorhandensein solcher Veränderungen kann mit FISH gezielt getestet werden.
Literatur
- A. R. Leitch, T. Schwarzacher, D. Jackson, I. J. Leitch: In situ-Hybridisierung. Spektrum Akademischer Verlag, Heidelberg/ Berlin 1994.
- J. M. Levsky, R. Singer: Fluorescence in situ hybridization: past, present and future. In: Journal of Cell Science. 116, Nr. 14, S. 2833–2838, PMID 12808017, doi:10.1242/jcs.00633.
Weblinks
- GenePaint.org (Datenbank für Genexpressionsmuster im Mausembryo, die mit In-situ-Hybridisierung untersucht wurden)
Einzelnachweise
- Joseph G. Gall, Mary Lou Pardue: Formation and detection of RNA-DNA hybrid molecules in cytological preparations. In: Proc. Natl. Acad. Sci. USA 63, Nr. 2, 1969, S. 378–383, PMID 4895535, PMC 223575 (freier Volltext).
- Mary Lou Pardue, Joseph G. Gall: Molecular hybridization of radioactive DNA to the DNA of cytological preparations. In: Proc. Natl. Acad. Sci. U.S.A. 64, Nr. 2, 1969, S. 600–604, PMID 5261036, PMC 223386 (freier Volltext).
- Andreas Bolzer, Gregor Kreth, Irina Solovei, Daniela Koehler, Kaan Saracoglu, Christine Fauth, Stefan Müller, Roland Eils, Christoph Cremer, Michael R. Speicher, Thomas Cremer: Three-dimensional maps of all chromosome positions demonstrate a probabilistic order in human male fibroblast nuclei and prometaphase rosettes. In: PLoS Biology. 3, Nr. 5, 2005, S. e157, PMID 15839726, doi:10.1371/journal.pbio.0030157.
- L. Brecevic, S. Michel, H. Starke, K. Müller, N. Kosyakova, K. Mrasek, A. Weise, T. Liehr: Multicolor FISH used for the characterization of small supernumerary marker chromosomes (sSMC) in commercially available immortalized cell lines. In: Cytogenetic & Genome Research. 114, Nr. 3,4, 2006, S. 319–324, doi:10.1159/000094220.
- Michael R. Speicher, Stephen Gwyn Ballard, David C. Ward: Karyotyping human chromosomes by combinatorial multi-fluor FISH. In: Nature Genetics. 12, 1996, S. 368–375, doi:10.1038/ng0496-368.
- E. Schröck, S. du Manoir, T. Veldman, B. Schoell, J. Wienberg, M. A. Ferguson-Smith, Y. Ning, D. H. Ledbetter, I. Bar-Am, D. Soenksen, Y. Garini, T. Ried: Multicolor Spectral Karyotyping of Human Chromosomes. In: Science. 273, Nr. 5274, 1996, S. 494–497, doi:10.1126/science.273.5274.494.