Denaturierung (Biochemie)
Denaturierung bezeichnet eine strukturelle Veränderung von Biomolekülen wie Proteinen (Eiweiße) oder Desoxyribonukleinsäure (DNS), die in den meisten Fällen mit einem Verlust der biologischen Funktion dieser Moleküle verbunden ist, obgleich deren Primärstruktur unverändert bleibt. Eine Denaturierung kann auf physikalische oder auf chemische Einflüsse zurückzuführen sein.

Prinzip
Äußere Einflüsse können nach dem Prinzip des kleinsten Zwangs Strukturveränderungen in Biopolymeren wie Proteinen oder Nukleinsäuren hervorrufen. Bei der Denaturierung eines Proteins verändert sich die Sekundär- und die Tertiärstruktur (und damit eventuell auch die Quartärstruktur), ohne dass sich die Reihenfolge der Aminosäuren ändert, dessen Primärstruktur. Das Protein verliert jedoch dabei seine ursprüngliche Faltungsform, die auch als native Konfiguration oder Konformation der Polypeptidkette bezeichnet wird. Mit dem Verlust der Faltung geht auch die Funktion des Proteins verloren, mit Ausnahme bei den intrinsisch unstrukturierten Proteinen.[1] Der Vorgang der Denaturierung kann irreversibel (unumkehrbar) oder reversibel (umkehrbar) sein, abhängig davon, ob die native Form auch den energetisch günstigsten Zustand darstellt. Der Umkehrvorgang einer Denaturierung heißt auch Renaturierung.[2] Da viele Proteine renaturierbar sind und ihre Funktion wiederhergestellt werden kann,[3] wurde postuliert, dass die Proteinfaltung bei der Proteinbiosynthese deshalb erfolgt, weil die native Form meist den energetisch günstigsten Zustand darstellt (Anfinsen-Dogma).[4]
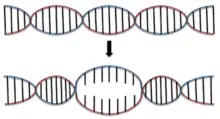
Solche reversiblen Veränderungen der Molekülstruktur liegen beispielsweise bei der Hitzedenaturierung von DNA vor, wenn diese durch Erhitzen in Einzelstränge aufgebrochen wird – etwa für eine Polymerase-Kettenreaktion (PCR) – und anschließend wieder abkühlt. Doppelsträngige DNA und RNA wird bei der Basenpaarung durch Wasserstoffbrückenbindungen zusammengehalten, die bei einer Denaturierung reversibel gelöst werden. Dagegen kann nach einer irreversiblen Veränderung der Molekülstruktur der ursprüngliche räumliche Aufbau des Moleküls nicht wiederhergestellt werden. Solche Veränderungen erfährt zum Beispiel ein Frühstücksei beim Kochen,[5] das weder durch Abkühlen in den Vorzustand überführbar ist, wenn es erst einmal ein „hartes Ei“ geworden ist, noch durch weiteres Kochen. Die Denaturierung von Proteinen führt in der Regel dazu, dass das Molekül inaktiviert wird, was heißt, dass es seine biologische Funktion kaum noch oder gar nicht mehr erfüllen kann. Daher wird die Denaturierung auch bei der Konservierung und der Desinfektion eingesetzt.
Allen Denaturierungsvorgängen ist gemeinsam, dass kovalente Bindungen nicht gespalten werden (außer den Disulfid-Brücken in Proteinen) und somit die Primärstruktur erhalten bleibt.[6] Die Kettenstruktur und damit die Abfolge der Bausteine als Primärstruktur bleibt also erhalten. Doch werden durch Energiezufuhr einzelne Bausteine, Nukleotide und Aminosäuren oder auch die ganze Molekülkette so sehr ins Schwingen gebracht, dass andere bindend wirkende Kräfte (ionische, polare und Van-der-Waals-Wechselwirkungen, Wasserstoffbrückenbindungen, hydrophobe Effekte) zwischen verschiedenen Bereichen der Molekülkette aufgehoben und solche Bindungen gelöst werden. Disulfidbrücken in Proteinen werden meistens durch Reduktion mit Sulfhydrylen gespalten.[7] Bei Proteinen wird bei einer Denaturierung oftmals eine Absenkung der Löslichkeit beobachtet,[3] teilweise begleitet von der Bildung von Proteinaggregaten. Ebenfalls steigt bei einer Denaturierung die Empfindlichkeit gegenüber einem Abbau durch Proteasen.[3]
Konformationsänderungen in Biopolymeren können mit FTIR, dualer Polarisationsinterferometrie, Circulardichroismus, QCM-D, NMR-Spektroskopie und multiparametrischer Oberflächenplasmonenresonanzspektroskopie gemessen werden.
Denaturierung durch physikalische Einflüsse
Die häufigsten Denaturierungen unter physikalischem Einfluss sind die Hitzedenaturierung und die Strahlungsdenaturierung. Physikalisch kann Denaturierung daneben auch durch hohen Druck, starkes Rühren, Schütteln, durch Ultraschalleinwirkung und durch Grenzflächenabsorption hervorgerufen werden.[8]
Hitzedenaturierung
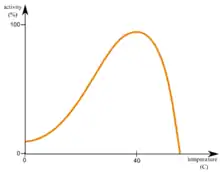

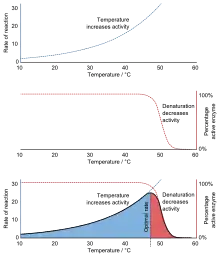
Die Wärme- oder Hitzedenaturierung ist eine Art der Denaturierung, bei der eine Veränderung der Molekülstruktur durch eine Erhöhung der Temperatur herbeigeführt wird. Dabei werden durch die Hitzeeinwirkung meist keine kovalenten chemischen Bindungen gebrochen oder gebildet, die Primärstruktur bleibt also unverändert. Stattdessen werden Wasserstoffbrücken gebrochen oder neu gebildet, das sind in der Regel Bindungen zwischen Kettenabschnitten, wodurch häufig eine Veränderung der Tertiärstruktur bei Enzymen und anderen Proteinen eintritt. Dies hat meist einen Verlust der biologischen Aktivität sowie eine Abnahme der Löslichkeit zur Folge. Letzteres macht sich dann als „Ausflocken“ oder „Gerinnung“ bemerkbar. Da bei der Proteinfaltung auch hydrophobe Effekte eine Rolle spielen, wird die Denaturierung auch von der Abnahme des hydrophoben Effektes mit zunehmender Temperatur erzeugt. Eine Hitzedenaturierung kann (wie andere Denaturierungen) reversibel sein, wenn die strukturellen Veränderungen noch nicht zu tiefgreifend sind, häufig ist sie aber irreversibel (unumkehrbar). Eine Umkehrung ist jedoch unter Laborbedingungen mit Hilfe von Zentrifugen und der Zugabe von Harnstoff möglich. Durch die auftretenden Scherkräfte können Hühnereier beispielsweise teilweise „entkocht“ werden.[9] Die Temperatur, bei der die Denaturierung der Proteine beginnt, ist je nach Aufbau und Organismus recht unterschiedlich. Die Enzyme hyper-thermophiler Archaeen müssen Temperaturen weit über 80 °C aushalten. Die Denaturierung durch Erhitzen wird in leicht saurer Lösung verstärkt.[10] Die thermische Denaturierung von Proteinen erfolgt meist in einem relativ engen Temperaturbereich, weshalb eine kooperative Entfaltung bei der Denaturierung vermutet wurde, d. h. eine Entfaltung begünstigt weitere.[7]
Durch Autoklavieren werden Krankheitserreger auf Gegenständen mittels Denaturieren lebenswichtiger Biopolymere inaktiviert. Beim Autoklavieren muss eine Temperatur deutlich über 100 °C bei erhöhtem Druck über eine vorgegebene Zeit eingehalten werden, um sicher zu sterilisieren.
Nukleinsäuren denaturieren innerhalb eines recht engen Temperaturintervalls, auch „Schmelzpunkt“ genannt, der meist oberhalb von 80 °C liegt. Die Denaturierung ist reversibel. Durch ein Abkühlen der Nukleinsäuren lagern sich die Einzelstränge wieder zusammen. Diesen Vorgang macht man sich in der Molekularbiologie bei der Durchführung von PCR zunutze, um bestimmte Gene aus einem Organismus in vitro zu vervielfältigen: extrahierte DNA wird bei hohen Temperaturen von etwa 95 °C in einem Reaktionsgefäß geschmolzen (Denaturierung). Anschließend wird die Temperatur wieder bis zu einer bestimmten Temperatur abgesenkt. Diese Annealing-Temperatur hängt von den Primern ab und liegt normalerweise 2–3 °C unter ihrem jeweiligen Schmelzpunkt (meistens 50 bis 65 °C). Die in der Lösung enthaltenen Primer lagern sich an die DNA-Einzelstränge an (Annealing oder auch Primerhybridisierung genannt). Nun werden mit Hilfe einer Taq-Polymerase die Stränge bei einer Temperatur von 68 bis 72 °C wieder vervollständigt (Elongation). Der Zyklus von Denaturierung, Annealing und Elongation beginnt von vorn. Es werden etwa 25 bis 50 Zyklen durchgeführt. Man macht sich also die reversible Denaturierung der DNA bis zu 50-mal zunutze, um ein gesuchtes Gen eines Organismus zu vervielfältigen. Bei einer Denaturierung nimmt die Extinktion von DNA bei einer Wellenlänge von 260 nm um etwa 40 % zu.[11]
Denaturierung durch Druck
Da die Reaktionsvolumina bei der Proteinfaltung sehr klein sind, muss man in der Regel Druck von mehreren 1000 bar aufwenden um Proteine zu entfalten. Trotzdem wird in der Praxis die Hochdruckbehandlung von Lebensmitteln[12] immer bedeutsamer. Dazu werden die Lebensmittel, zumeist in Folien verpackt, in ein Druckmedium, wie z. B. Wasser, gegeben und der Druck wird auf dieses Medium ausgeübt. In diesem „nichtthermischen Verfahren“, einer Hochdruckpasteurisierung, werden unerwünschte Mikroorganismen und Enzyme inaktiviert und die Lebensmittel haltbar gemacht. Qualitätsverluste, wie bei der Anwendung von Hitze, werden dabei vermieden.
Generell gilt, dass durch Druck die Tertiär- und Quartärstruktur der Proteine beeinflusst wird, während die Sekundärstruktur kaum verändert werden kann.
Denaturierung durch Strahlung
Infrarot, Mikrowellen oder andere langwellige Strahlung wirkt aufgrund der hervorgerufenen Temperaturerhöhung denaturierend. Bei ionisierender Strahlung (Ultraviolett, Gamma- und Röntgenstrahlung) können zusätzlich kovalente Bindungen z. B. von Nukleinsäuren aufgebrochen werden und so zu DNA-Schäden wie Kettenbrüchen (Depolymerisationen) führen. Daneben können in der Folge der Bindungsspaltung auch neue kovalente Bindungen (zum Beispiel Dimerisierung in Nukleinsäuren) entstehen.
Denaturierung durch chemische Einflüsse
Ursachen der Proteindenaturierung können zum Beispiel chemische Substanzen wie einige Säuren, Basen, Salze (z. B. Guanidiniumsalze), Detergentien oder Harnstoff sein.[13] Proteinstrukturen können auch durch Schwermetalle beeinflusst werden, da die Ionen Komplexstrukturen mit den Aminosäureresten bilden und so die biologisch aktive Struktur des Proteins verändern.
Säure- und Lauge-Denaturierung
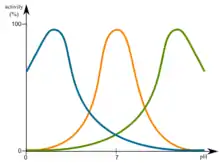
Je nachdem, wie der pH-Wert der natürlichen Umgebung des Proteins ist, besitzen Proteine ein pH-Optimum. Dieses Optimum kann im sauren pH-Bereich liegen, wie beispielsweise bei lysosomalen Proteinen. In anderen Fällen kann dieses jedoch auch im Basischen liegen. Im Bereich des pH-Optimums ist ein Protein am stabilsten und denaturiert daher nicht.
Die Säuredenaturierung, beispielsweise mit 40 % (V/V) Essigsäure,[14] führt zu Ladungsverschiebungen zwischen den Molekülen und letzten Endes zu einer Umfaltung des Proteins in den unter den jeweiligen Bedingungen energetisch günstigsten Zustand. Die Säure gibt Protonen (H+) ab und verursacht damit die Ladungsänderung in der Proteinstruktur, sodass die Wasserstoffbrückenbindungen teilweise zerstört werden und die gleichen positiven Ladungen sich gegenseitig abstoßen. Zusätzlich gibt die Säure Protonen (H+) an die Carboxylatgruppe (COO−) der Aminosäuren Aspartat und Glutamat ab, sodass Carboxygruppen –COOH entstehen und deren vorherigen negativen Ladungen verschwinden. Dies führt dazu, dass keine ionischen Wechselwirkungen zwischen der Carboxygruppe und den positiven Ladungen im Protein mehr möglich sind.
Entsprechendes können Laugen bewirken, auch sie ändern die Zusammensetzung der Ionen über den pH-Wert, jedoch werden Aminogruppen von Lysin oder Arginin deprotoniert, wodurch weniger positive Ladungen im Protein vorkommen, die mit negativ geladenen Gruppen wechselwirken könnten. Zusätzlich werden Carbonsäuregruppen zu Carboxylaten deprotoniert, wodurch Wasserstoffbrückenbindungen zerstört werden können und mehr negative Ladungen im Protein auftreten, die sich gegenseitig abstoßen.
Bei der Säure- oder Laugendenaturierung kann gleichzeitig eine Hydrolyse des Proteins auftreten.
Im Hinblick auf den pH-Wert ist auch der isoelektrische Punkt (pI) eines Proteins von Bedeutung. An diesem Punkt ist ein Protein in der Nettoladung ungeladen und fällt daher schnell aus der Lösung aus. Am pI ist das Protein somit sehr empfindlich.[15]
Denaturierung durch Chaotrope
Auch Salze und andere Chaotrope haben einen Einfluss auf hydrophobe Effekte und können daher eine Denaturierung hervorrufen, wobei je nach Stoff, der Einfluss auch in Richtung Renaturierung gehen kann. Man spricht dann, bezüglich der Ausfällung, auch von „Einsalzen“ und „Aussalzen“. Der relative Einfluss der Salze bildenden Anionen und Kationen wird durch die „Hofmeister-Reihe“ beschrieben.
DNA wird durch Formamid (70 % V/V),[16] Dimethylformamid,[17] Guanidiniumsalze (6 M),[18] Natriumsalicylat,[17] Sulfoxid,[17] Dimethylsulfoxid (DMSO, 60 % V/V),[19] Natriumhydroxid (1 M),[19] verschiedene Alkohole,[17] Propylenglykol und Harnstoff (6 M)[18] denaturiert, meist in Kombination mit Wärme. Bei doppelsträngiger DNA erfolgt eine Absenkung der Schmelztemperatur.
Denaturierung durch Detergentien
Manche Tenside führen zur Denaturierung. Dies liegt daran, dass sie sich überall an das Protein anlagern und es dadurch linearisieren. Ionische Detergentien denaturieren am stärksten, z. B. eine einprozentige Lösung von Natriumlaurylsulfat (SDS). Bei Raumtemperatur werden bereits die meisten Proteine denaturiert. Daneben werden auch Membranlipide aus den Zellmembranen gelöst, indem sich ab einer bestimmten Konzentration Mizellen aus dem Tensid und der Membran bilden. Die SDS-Denaturierung wird z. B. in der Probenvorbereitung zur SDS-PAGE verwendet.
Denaturierung durch Ethanol
Entsprechend der Säuredenaturierung können Ethanol oder andere wasserlösliche, organische Lösungsmittel die in Biopolymeren zur Aufrechterhaltung der Struktur erforderlichen Wasserstoffbrücken und hydrophoben Wechselwirkungen stören, indem sie als polare organische Lösungsmittel interferieren. 50- bis 70-prozentiges Ethanol denaturiert die meisten Proteine. Da durch das Herauslösen der Membranlipide sowie durch die Denaturierung der Raumstruktur auch die Membranproteine ihre Funktion verlieren und die betreffenden Zellen seifenblasenartig platzen, kann so mit höherprozentigen Alkoholen (z. B. Ethanol, Isopropanol) desinfiziert werden: Bakterien- und Pilzzellen werden über die Denaturierung ihrer Membranproteine und der Durchlöcherung ihrer Zellmembran irreversibel inaktiviert, entsprechend werden behüllte Viren gleichzeitig zur Denaturierung der Proteine auch ihrer Lipidhülle beraubt, in der die Andockproteine sitzen.
Denaturierung durch reines Wasser
Proteine liegen in ihrer natürlichen Umgebung in Gegenwart von anderen Proteinen, gelösten Salzen, Cofaktoren oder Metaboliten vor, die auf mehr oder weniger komplexe Weise die natürliche Proteinstruktur stabilisieren. Entfernt man Salze und andere kleinere Moleküle durch Dialyse einer Proteinlösung gegen bidestilliertes Wasser – vorzugsweise in der Kälte –, kann man oft selektive (und reversible) Denaturierung vor allem von großen Proteinen erreichen, die unter diesen Bedingungen ausgefällt werden (präzipitieren).
Denaturierung durch Modifikation und Vernetzung
Auch durch die Verwendung von Molekülmarkierungen, Fixierungslösungen und Gerbstoffen, kovalenten Vernetzern (z. B. Formaldehyd, Paraformaldehyd oder Glutaraldehyd) und Lösungen von Schwermetallionen, die stabile Komplexe ausbilden, wird gelegentlich das katalytische Zentrum oder eine Bindungsstelle eines Proteins so verändert, dass manche Funktionen nicht mehr erfüllt werden. Das Protein wird hierbei nicht (wie bei den thermischen, chaotropen oder pH-abhängigen Denaturierungen) entfaltet, es kann dabei jedoch verändert oder in einer nicht-nativen Konformation fixiert werden und Funktionen verlieren. Bleiben notwendige Funktionen des Proteins von der Fixierung unberührt, so können durch eine Vernetzung auch andere Eigenschaften wie die biologische Halbwertszeit verändert werden. Im Zuge einer Antigendemaskierung wird versucht, die Effekte der Fixierung rückgängig zu machen.
Denaturierung in Lebewesen
Proteine werden in Zellen teilweise vor einem Membrantransport durch Chaperone entfaltet und falten sich anschließend zurück. Die Basenpaarung von DNA wird durch verschiedene DNA-bindende Proteine abschnittsweise aufgehoben, zum Beispiel bei der Replikation oder der Transkription. Der Ort des Denaturierungsbeginns wird als Denaturierungsblase bezeichnet[20] und im Poland-Scheraga-Modell beschrieben.[21] Jedoch wird die DNA-Sequenz, die Steifigkeit und die Torsion nicht miteinbezogen.[22] Die Lebensdauer einer Denaturierungsblase beträgt zwischen einer Mikrosekunde und einer Millisekunde.[23]
Renaturierung
Nach der Denaturierung eines Proteins (z. B. eines Enzyms) während der Aufreinigung aus einem Proteingemisch ist zur Messung der biologischen Aktivität eine Rückkehr des Proteins in die native Form notwendig. Dies geht jedoch nur bei Proteinen, deren native Konformation zugleich den energetisch günstigsten Zustand unter isotonischen Bedingungen darstellt, nicht aber bei metastabilen Proteinen. Eine Renaturierung nach chemischer Denaturierung kann durch langsame Verdünnung des Denaturierungsmittels erreicht werden, begleitet von einer Wiederherstellung der Cofaktoren und der isotonischen Umgebung. Teilweise werden weitere Zusätze verwendet.[24] Eine Rekonstitution kann im Anschluss erfolgen. Bei einer thermischen Denaturierung von Nukleinsäuren hybridisieren diese wieder bei Absenken der Temperatur.[25] Die Geschwindigkeit der Renaturierung der Basenpaarung von Nukleinsäuren steigt mit dem Anteil an korrekter Basenpaarung.[10]
Abgrenzung zu anderen Veränderungen
Nicht als Denaturierung bezeichnet werden die durch Proteine vermittelten Strukturänderungen:
- durch Enzyme synthetisierte, umgewandelte oder abgebaute Moleküle, Aktivierungs- und Deaktivierungsreaktionen; beispielsweise wird Milchprotein durch Lab-Enzyme koaguliert
- Konformationsänderungen durch Chaperone oder Prionen.
Bei sehr hoher Temperatur kann es auch zur Spaltung kovalenter Bindungen und damit zu Kettenbrüchen (Depolymerisation) kommen. Solche Änderungen der Primärstruktur werden nicht zu den Denaturierungen gezählt. Ebenso können Säuren wie Laugen bei hohen Konzentrationen und Reaktionstemperaturen zur Spaltung kovalenter Bindungen führen. Durch Hydrolyse ändert sich dann die Primärstruktur. Solche Veränderungen der Primärstruktur sind gewöhnliche chemische Reaktionen und werden nicht zu den Denaturierungen gerechnet.
Ein Grenzfall ist die Spaltung von Disulfidbrücken zwischen zwei Proteinsträngen. Dabei wird zwar eine kovalente chemische Bindung gelöst, die Aminosäuresequenz in jedem einzelnen Strang bleibt jedoch erhalten, deshalb zählt eine solche reduktive Spaltung von Disulfidbrücken, welche prinzipiell reversibel ist, zu den Denaturierungen.[26]
Anwendungen
Bei Tieren findet eine Denaturierung von Proteinen im Magen statt.[27]
Die Denaturierung wird unter anderem bei der Herstellung von proteinhaltigen Lebensmitteln verwendet, z. B. beim Garen sowie bei der Herstellung von Panir oder Tofu. Durch die Denaturierung beim Garen wird die Verdauung erleichtert,[3] daneben werden im Nahrungsmittel befindliche Mikroorganismen abgetötet und Viren inaktiviert. Verschiedene Methoden der Desinfektion[3] und teilweise auch der Lebensmittelkonservierung (beim Erhitzen oder Trocknen) verwenden die Denaturierung der Proteine von unerwünschten Mikroorganismen, wodurch diese abgetötet werden. Bei der Erzeugung einer Dauerwelle werden Haare denaturiert, um ihre Form zu verändern.[28]
In der Biochemie wird die Denaturierung zur Entfaltung von Proteinen eingesetzt, z. B. chemisch bei der SDS-PAGE oder einer Proteinsequenzierung. Bei einer Bindung von Molekülen an ein Protein kann die Thermostabilität des Proteins erhöht werden. Die Veränderung der Denaturierungstemperatur eines Proteins bei einer Bindung eines anderen Moleküls kann mit einem Thermal Shift Assay gemessen werden und somit die Bindung nachgewiesen werden. Weiterhin wird die Denaturierung zur Inaktivierung von Enzymen bei einer DNA-Extraktion oder einer RNA-Extraktion verwendet. Bei Nukleinsäuren wird die PCR, die DNA-Sequenzierung, der Southern und Northern Blot, die in-situ-Hybridisierung, die TGGE mit jeweils thermischer Denaturierung verwendet, während die DGGE und die Harnstoff- oder Formamid-Polyacrylamid-Gelelektrophorese mit chemischer Denaturierung erfolgt.
Literatur
- Friedrich Lottspeich, Haralabos Zorbas: Bioanalytik. Spektrum Akademischer Verlag, Heidelberg 1998, ISBN 978-3827400413.
- Hubert Rehm, Thomas Letzel: Der Experimentator: Proteinbiochemie / Proteomics. 6. Auflage, Spektrum Akademischer Verlag, Heidelberg 2009, ISBN 978-3827423122.
Weblinks
Einzelnachweise
- Jane H. Dyson, Peter E. Wright: Intrinsically unstructured proteins and their functions In: Nature Reviews Molecular Cell Biology. (2005), Band 6, Ausgabe 3, S. 197–208. doi:10.1038/nrm1589.
- Hans-Dieter Jakubke, Hans Jeschkeit: Aminosäuren, Peptide, Proteine, Verlag Chemie, Weinheim, 402, 1982, ISBN 3-527-25892-2.
- Philipp Christen, Rolf Jaussi, Roger Benoit: Biochemie und Molekularbiologie. Springer-Verlag, 2015, ISBN 978-3-662-46430-4, S. 36, 37.
- C. B. Anfinsen: Principles that govern the folding of protein chains. In: Science. Band 181, Nummer 4096, Juli 1973, S. 223–230, PMID 4124164.
- Yoshinori Mine, Tatsushi Noutomi, Noriyuki Haga: Thermally induced changes in egg white proteins. In: Journal of Agricultural and Food Chemistry. 38, 1990, S. 2122, doi:10.1021/jf00102a004.
- Charles Tanford: Protein denaturation. In: Advances in protein chemistry. Band 23, 1968, S. 121–282, PMID 4882248.
- H. Robert Horton et al.: Biochemie. Pearson Deutschland GmbH, 2008, ISBN 978-3-827-37312-0, S. 144–146.
- Hans-Dieter Jakubke, Hans Jeschkeit: Aminosäuren, Peptide, Proteine, Verlag Chemie, Weinheim, 403, 1982, ISBN 3-527-25892-2.
- Yuan et al.: Shear-Stress-Mediated Refolding of Proteins from Aggregates and Inclusion Bodies. In: ChemBioChem. Nr. 16, 2015, S. 393–396.
- Peter Karlson: Karlsons Biochemie und Pathobiochemie. Georg Thieme Verlag, 2005, ISBN 978-3-133-57815-8, S. 35.
- Gerhard Richter: Praktische Biochemie. Georg Thieme Verlag, 2003, ISBN 978-3-131-32381-1, S. 144, 195.
- E. Palou, A. Lopet-Malo, G. V. Barbosa-Canovas und B. G. Swanson: High Pressure Treatment in Food Preservation, Marcel Dekker, New York, 1999.
- Hans-Dieter Jakubke, Hans Jeschkeit: Aminosäuren, Peptide, Proteine, Verlag Chemie, Weinheim, 403–404, 1982, ISBN 3-527-25892-2.
- J. P. López-Alonso, M. Bruix, J. Font, M. Ribó, M. Vilanova, M. A. Jiménez, J. Santoro, C. González, D. V. Laurents: NMR spectroscopy reveals that RNase A is chiefly denatured in 40% acetic acid: implications for oligomer formation by 3D domain swapping. In: Journal of the American Chemical Society. Band 132, Nummer 5, Februar 2010, S. 1621–1630, doi:10.1021/ja9081638, PMID 20085318.
- Popova, E.: Phasengleichgewichte beim Ausfällen von Proteinen aus wässrigen Lösungen. 2007, abgerufen am 20. November 2019.
- J. Marmur, P. O. Ts'O: Denaturation of deoxyribonucleic acid by formamide. In: Biochimica et Biophysica Acta. Band 51, Juli 1961, S. 32–36, PMID 13767022.
- Academic Press: PROG NUCLEIC ACID RES&MOLECULAR BIO. Academic Press, 1963, ISBN 978-0-080-86289-7, S. 267.
- Hyone-Myong Eun: Enzymology Primer for Recombinant DNA Technology. Elsevier, 1996, ISBN 978-0-080-53113-7, S. 67.
- X. Wang, H. J. Lim, A. Son: Characterization of denaturation and renaturation of DNA for DNA hybridization. In: Environmental health and toxicology. Band 29, 2014, S. e2014007, doi:10.5620/eht.2014.29.e2014007, PMID 25234413, PMC 4168728 (freier Volltext).
- François Sicard, Nicolas Destainville, Manoel Manghi: DNA denaturation bubbles: Free-energy landscape and nucleation/closure rates. In: The Journal of Chemical Physics. 142, Nr. 3, 21. Januar 2015, S. 034903. arxiv:1405.3867. doi:10.1063/1.4905668.
- Simon Lieu: The Poland-Scheraga Model. Massachusetts Institute of Technology (MIT), 14. Mai 2015, S. 1–5. Auf web.MIT.edu (PDF; 522,7 kB, englisch), abgerufen am 25. Februar 2022.
- C. Richard, A. J. Guttmann: Poland–Scheraga Models and the DNA Denaturation Transition. In: Journal of Statistical Physics. Band 115, 2004, S. 925, doi:10.1023/B:JOSS.0000022370.48118.8b.
- Grégoire Altan-Bonnet, Albert Libchaber, Oleg Krichevsky: Bubble Dynamics in Double-Stranded DNA. In: Physical Review Letters. 90, Nr. 13, 1. April 2003. doi:10.1103/physrevlett.90.138101.
- Winfried Storhas: Bioverfahrensentwicklung. John Wiley & Sons, 2013, ISBN 978-3-527-67385-8, S. 621.
- Rolf Knippers: Molekulare Genetik. Georg Thieme Verlag, 2015, ISBN 978-3-131-68330-4.
- Brockhaus ABC Chemie, VEB F. A. Brockhaus Verlag Leipzig 1965, S. 274.
- Robert Ebermann: Lehrbuch Lebensmittelchemie und Ernährung. Springer-Verlag, 2011, ISBN 978-3-709-10211-4, S. 57.
- Peter W. Atkins: Physikalische Chemie. John Wiley & Sons, 2006, ISBN 978-3-527-31546-8, S. 754.