Nukleinsäuren
Nukleinsäuren, auch Nucleinsäuren,[1] sind aus einzelnen Bausteinen, den Nukleotiden, aufgebaute Makromoleküle, die bei allen Organismen (Viren und zellulären Organismen) die genetische Information[2] enthalten. Abwechselnde Einfachzucker und Phosphorsäureester bilden eine Kette, wobei an jedem Zucker eine Nukleinbase hängt. Nukleinsäuren bilden neben Proteinen, Kohlenhydraten und Lipiden die vierte große Gruppe der Biomoleküle. Ihr bekanntester Vertreter als Grundtyp der Nukleinsäuren ist die Desoxyribonukleinsäure (DNS bzw. DNA); diese ist bei allen zellulären Organismen (Lebewesen) der Speicher der Erbinformation, lediglich bei manchen Viren kommt stattdessen Ribonukleinsäure (RNS bzw. RNA) in dieser Funktion vor. Neben ihrer Aufgabe als Informationsspeicher können die als „Schlüsselmoleküle des Lebens“[2] geltenden Nukleinsäuren auch als Signalüberträger dienen oder biochemische Reaktionen katalysieren (Ribozyme).
Geschichte

Erstmals beschrieben wurde die Nukleinsäure von dem Schweizer Mediziner Friedrich Miescher im Jahr 1869 nach seinen Untersuchungen im Labor der ehemaligen Küche des Tübinger Schlosses. Er war Mitarbeiter des Begründers der Biochemie, Felix Hoppe-Seyler. Nachdem Miescher seine Forschungen an Proteinen aufgab, weil diese zu komplex und zu vielfältig waren, wandte er sich der Untersuchung von Zellkernen zu. Deren Funktion war zu damaliger Zeit völlig unbekannt. Aus den Kernen von weißen Blutkörperchen isolierte er eine Substanz, die sich durch ihren hohen Phosphorgehalt deutlich von Proteinen unterschied. Er nannte sie Nuclein nach dem lateinischen Wort nucleus (Kern). Obwohl Miescher der Funktion von Nuclein schon sehr nahekam, glaubte er letztendlich nicht, dass ein einziger Stoff für die Vererbung verantwortlich sein könnte.
„Sofern wir (…) annehmen wollten, dass eine einzelne Substanz (…) auf irgendeine Art (…) die spezifische Ursache der Befruchtung sei, so müsste man ohne Zweifel vor allem an das Nuclein denken.“
1885 teilte Albrecht Kossel mit, dass aus einer größeren Menge Rinder-Bauchspeicheldrüse eine stickstoffreiche Base mit der Summenformel C5H5N5 isoliert wurde, für die er, abgeleitet von dem griechischen Wort „aden“ für Drüse, den Namen Adenin vorschlug.[3][4] 1889 isolierte Richard Altmann aus dem Nuklein neben einem eiweißartigen Bestandteil eine phosphorhaltige, organische Säure, die er Nucleinsäure nannte.[5] 1891 konnte Kossel (nach Altmanns Verfahren) Hefe-Nukleinsäure herstellen und Adenin und Guanin als Spaltprodukte nachweisen. Es stellte sich heraus, dass auch ein Kohlenhydrat Bestandteil der Nukleinsäure sein musste. Kossel wählte für die basischen Substanzen Guanin und Adenin sowie seine Derivate den Namen Nucleinbasen.[6] 1893 berichtete Kossel, dass er aus den Thymusdrüsen des Kalbes Nukleinsäure gewonnen und ein gut kristallisiertes Spaltprodukt erhalten hatte, für das er den Namen Thymin vorschlug. 1894 isolierte er aus den Thymusdrüsen eine weitere (basische) Substanz. Kossel gab dieser Nukleinbase den Namen Cytosin.[7]
Nachdem am Ende des 19. Jahrhunderts – im Wesentlichen durch die Synthesen Emil Fischers – die Strukturformeln des Guanins und Adenins als Purinkörper und des Thymins als Pyrimidinkörper endgültig aufgeklärt worden waren, konnte Kossel mit Hermann Steudel auch die Strukturformel der Nukleinbase Cytosin als Pyrimidinkörper zweifelsfrei ermitteln.[8] Es hatte sich inzwischen erwiesen, dass Guanin, Adenin sowie Thymin und Cytosin in allen entwicklungsfähigen Zellen zu finden sind.
Die Erkenntnisse über diese vier Nukleinbasen sollten für spätere die Strukturaufklärung der DNA von wesentlicher Bedeutung sein. Es war Albrecht Kossel, der sie – zusammen mit einem Kohlenhydrat und der Phosphorsäure – eindeutig als Bausteine der Nukleinsäure charakterisierte:
„Es gelang mir, eine Reihe von Bruchstücken zu erhalten […] welche durch eine ganz eigentümliche Ansammlung von Stickstoffatomen gekennzeichnet sind. Es sind hier nebeneinander […] das Cytosin, das Thymin, das Adenin und das Guanin.“
Phoebus Levene schlug eine kettenartige Struktur der Nukleinsäure vor. Er prägte den Begriff „Nukleotid“ für die Baueinheiten der Nukleinsäure. 1929 konnte er den Zuckeranteil der „tierischen“ Nukleinsäure als Desoxyribose identifizieren.[10] Nachfolgend wurde sie als Desoxyribonukleinsäure bezeichnet. Es wurde erkannt, dass die Desoxyribonukleinsäure auch in pflanzlichen Zellkernen vorkommt.
1944 konnten Oswald Avery, Colin McLeod und Maclyn McCarty nachweisen, dass Nukleinsäuren die Speicher der Erbinformation sind und nicht – wie bis dahin angenommen – Proteine.
Dem Amerikaner James Watson (* 1928) und den Engländern Francis Crick (1916–2004), Rosalind Franklin (1920–1958) und Maurice Wilkins (1916–2004) gelang es schließlich, den Aufbau der Desoxyribonukleinsäure aufzuklären. Watson, Crick und Wilkins erhielten 1962 den Nobelpreis.
Frederick Sanger, sowie Allan Maxam und Walter Gilbert entwickelten 1977 unabhängig voneinander Verfahren, mit denen die Reihenfolge der Nukleotid-Bausteine, die Sequenz, bestimmt werden konnte. Die Kettenabbruchmethode wird heute in automatisierten Verfahren verwendet, um DNA zu sequenzieren.
Aufbau
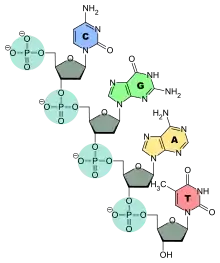
Chemische Struktur
Nukleinsäuren sind Ketten mit Nukleotiden als Gliedern. Der zentrale Teil eines Nukleotids ist das ringförmige Zuckermolekül (Im Bild grau: die Ribose). Nummeriert man die Kohlenstoffatome dieses Zuckers im Uhrzeigersinn von 1 bis 5, so ist am C1 eine Nukleinbase (Bild 1: rot, grün, gelb und blau) über eine glykosidische Bindung angeknüpft. Am C3 hat ein Phosphatrest des nachfolgenden Nukleotids (blau) mit der OH-Gruppe des Zuckers eine Esterbindung ausgebildet. Am C4 des Zuckers ist über die andere der beiden Phosphodiesterbindungen ebenfalls ein Phosphatrest gebunden.
Die Phosphorsäure besitzt in ungebundenem Zustand drei acide Wasserstoffatome (an den OH-Gruppen), die abgespaltet werden können. In einer Nukleinsäure sind zwei der drei OH-Gruppen verestert und können somit kein Proton mehr freisetzen. Für den sauren Charakter, der der Nukleinsäure ihren Namen gab, ist die dritte ungebundene Säurefunktion verantwortlich. Sie kann als Protonendonator agieren oder liegt in der Zelle deprotoniert vor (negative Ladung am Sauerstoff-Atom). Unter physiologischen Bedingungen (pH 7) ist die Nukleinsäure aufgrund dieses negativ geladenen Sauerstoffatoms insgesamt ein großes Anion. Bei der Auftrennung von Nukleinsäuren nach ihrer Größe kann man daher ein elektrisches Feld nutzen, in dem Nukleinsäuren grundsätzlich zur Anode wandern (siehe Agarose-Gelelektrophorese).
Die Ketten der Nukleinsäuren sind gewöhnlich unverzweigt (entweder linear oder ringförmig geschlossen, d. h. zirkulär). Zu Ausnahmen siehe beispielsweise Okazaki-Fragment, Holliday-Struktur und Kleeblattstruktur.
Orientierung
Ihr Aufbau verleiht der Nukleinsäure eine Polarität respektive Orientierung in der Kettenbausteinabfolge. Sie hat ein 5′-Ende (sprich: 5-Strich-Ende, englisch five prime end), benannt nach dem C5-Atom des Zuckers, an dem ein Phosphatrest gebunden ist, und ein 3′-Ende, an dem die freie OH-Gruppe am C3-Atom die Kette abschließt. Üblicherweise schreibt man Sequenzen, also Nukleotidfolgen, mit dem 5′-Ende beginnend zum 3′-Ende hin auf. In Organismen ist die Polarität sehr wichtig. So gibt es beispielsweise DNA-Polymerasen, die einen DNA-Strang nur in 5′→3′-Richtung aufbauen können, und wieder andere korrigieren falsch eingebaute Nukleotide nur in 3′→5′-Richtung.
Räumliche Struktur
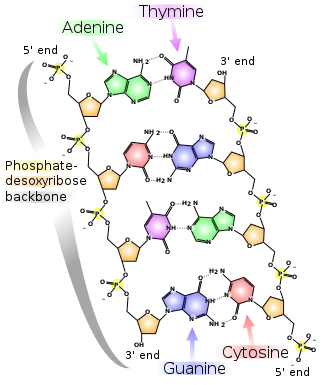
Als Sekundärstruktur bezeichnet man bei Nukleinsäuren die räumliche Ausrichtung. Während die Primärstruktur (die Sequenz) die Informationen speichert, bestimmt die Sekundärstruktur über Größe, Haltbarkeit und auch Zugriff auf die gespeicherten Informationen.
Die einfachste räumliche Struktur ist der Doppelstrang. Hier liegen sich zwei Nukleinsäureketten in entgegengesetzter Orientierung gegenüber. Sie sind über Wasserstoffbrückenbindungen zwischen den Nukleinbasen miteinander verbunden. Dabei paaren sich jeweils eine Pyrimidinbase mit einer Purinbase, wobei die Art des jeweiligen Paares die Stabilität des Doppelstranges bestimmt. Zwischen Guanin und Cytosin bilden sich drei Wasserstoffbrückenbindungen aus, während Adenin und Thymin nur durch zwei Wasserstoffbrücken verbunden sind (siehe Bild 2). Je höher der GC-Gehalt (Anteil an Guanin-Cytosin-Paaren) ist, desto stabiler ist der Doppelstrang und desto mehr Energie (Wärme) muss aufgewendet werden, um ihn in Einzelstränge zu spalten. Ein Doppelstrang kann aus zwei verschiedenen Nukleinsäuremolekülen bestehen oder nur aus einem einzigen Molekül. Am Ende des Doppelstranges bildet sich dann eine Schlaufe, in der die Kette „umkehrt“, so dass die entgegengesetzte Orientierung entsteht.
Bei der DNA windet sich der Doppelstrang als Ergebnis der vielen verschiedenen Bindungswinkel um seine eigene Achse und bildet eine Doppelhelix. Es gibt sowohl links- als auch rechtsgängige Helices. Dieser um sich selbst gewundene Doppelstrang kann dann noch weiter verdrillt werden und sich um andere Strukturen wie Histone (spezielle Proteine) wickeln. Sinn dieser weiteren Verknäulung ist das Sparen von Platz. Unverdrillt und ausgestreckt wäre die DNA eines einzigen menschlichen Chromosoms etwa 4 cm lang.
Natürliche Nukleinsäuren
Nukleinsäuren kommen in allen lebenden Organismen vor. Ihre Aufgabe ist es unter anderem die genetische Information, den Bauplan des jeweiligen Organismus, zu speichern, mit anderen ihrer Art auszutauschen und an nachfolgende Generationen zu vererben. In allen Organismen tut das die DNA. Nur einige Viren (Retroviren wie zum Beispiel HIV) nutzen die weniger stabile RNA als Speichermedium. Jedoch könnten hypothetische Ribozyten als Vorläufer der heutigen zellulären Organismen in den Urzeiten der Erde ebenfalls ein RNA-Genom besessen haben (RNA-Welt-Hypothese), einen Nachweis gibt es dafür bislang nicht. Es wird auch diskutiert, ob das Leben stattdessen mit einer chimären DNA-RNA-Nukleinsäure begann.[11][12] Darüber hinaus werden auch andere Nukleinsäuren als Vorgänger von RNA bzw. DNA diskutiert (XNA, s. u.).
Desoxyribonukleinsäure (DNS, DNA)
DNA hat als Zuckerbestandteil Desoxyribose (daher der Name Desoxyribonukleinsäure), die sich von der Ribose nur durch die fehlende OH-Gruppe am C2-Atom unterscheidet. Die Reduktion der OH-Gruppe zum einfachen H findet erst am Ende der Nukleotidsynthese statt. Desoxyribonukleotide entstehen also aus den Ribonukleotiden, den RNA-Bausteinen. Der Unterschied jedoch macht DNA chemisch sehr viel stabiler als RNA (Begründung siehe Abschnitt RNA) und zwar so stabil, dass sie gelöst in Meerwasser (1 ppb) und Flussmündungen (bis 44 ppb) nachzuweisen ist. In der DNA kommen die Nukleinbasen Adenin, Cytosin, Guanin und Thymin vor, wobei letztere spezifisch für DNA ist. Trotz der geringen Menge von vier verschiedenen Grundbausteinen kann viel Information gespeichert werden.
- Rechenbeispiel:
- Ein DNA-Stück aus 4 möglichen Grundbausteinen mit einer Gesamtlänge von 10 Basenpaaren ergibt 410 = 1.048.576 mögliche Kombinationen
- Das Genom des Bakteriums E. coli hat eine ungefähren Umfang von 4 × 106 Basenpaaren. Da es für ein Basenpaar 4 Möglichkeiten (A, C, G oder T) gibt, entspricht es 2 bit (22 = 4). Damit hat das gesamte Genom einen Informationsgehalt von 1 Megabyte.
Die DNA liegt als Doppelstrang vor, der um sich selbst gewunden eine Doppelhelix bildet. Von den durch Röntgenstrukturanalyse identifizierten drei Helixtypen, ist bisher nur die B-DNA in vivo nachgewiesen worden. Sie ist eine rechtsgängige Helix mit einer Ganghöhe (Länge der Helix für eine komplette Windung) von 3,54 nm und 10 Basenpaaren und einem Durchmesser von 2,37 nm. Weiterhin existieren die breitere A-Helix (Ganghöhe 2,53 nm; Durchmesser 2,55 nm) und die gestrecktere Z-Helix (Ganghöhe 4,56 nm; Durchmesser 1,84 nm). Soll ein in der DNA codiertes Gen abgelesen oder die DNA selbst im Zuge der Zellteilung verdoppelt werden, so wird die Helix auf einem Teilstück durch Enzyme entwunden (Topoisomerasen) und der Doppelstrang in Einzelstränge gespalten (Helikasen).
In Bakterien liegt die DNA als ringförmiges Molekül vor, während sie bei Eukaryoten freie Enden, die sogenannten Telomere, besitzt. Die Beschaffenheit des DNA-Replikationsmechanismus führt dazu, dass lineare DNA-Moleküle pro Verdopplung um ein paar Basenpaare verkürzt werden. Je häufiger sich eine Zelle teilt, desto kürzer wird die DNA. Das bleibt bei begrenzter Zellteilung ohne Folgen, da sich am Ende eines solchen Stranges kurze Sequenzen befinden, die sich mehrere tausend Male wiederholen. Es geht also keine Erbinformation verloren. Teilweise wird die Verkürzung auch durch das Enzym Telomerase ausgeglichen (nur in Stammzellen und Krebszellen). Unterschreitet die Länge der repetitiven Sequenzen am Strangende eine bestimmte Länge, so teilt sich die Zelle nicht mehr. Hier liegt einer der Gründe für eine begrenzte Lebensdauer. Da Bakterien ein ringförmiges DNA-Molekül haben, kommt es bei ihnen nicht zu einer Verkürzung des Stranges.
Ribonukleinsäure (RNS, RNA)
Die OH-Gruppe am C2-Atom der Ribose ist für die geringere Stabilität der RNA verantwortlich. Sie kann nämlich, ebenso wie die OH-Gruppe am C3-Atom für die normale Kettenbildung, eine Verknüpfung mit dem Phosphatrest eingehen. Kommt es spontan zu einer solchen Umesterung, wird die Nukleinsäurekette unterbrochen.
 Cytosin |
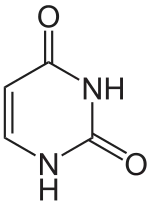 Uracil |
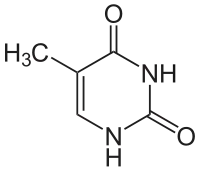 Thymin |
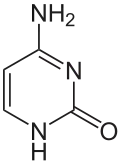
Ein weiterer Unterschied ist, dass in der DNA Thymin verwendet wird, während in der RNA Uracil vorkommt. Durch oxidative Bedingungen oder andere Einwirkungen können Nukleinbasen innerhalb der DNA chemisch verändert werden. So kommt es gelegentlich zu einer Desaminierung (Abspaltung einer NH2-Gruppe, es entsteht stattdessen eine O=Gruppe). In einem Doppelstrang passen dann die Stellen für Wasserstoffbrückenbindungen der gegenüberliegenden Nukleinbasen nicht mehr zusammen und es kommt zu einer partiellen Aufspaltung. Enzyme können veränderte Nukleinbasen ausschneiden und ersetzen oder reparieren. Als Vorlage orientieren sie sich dazu an der zweiten nicht veränderten Nukleinbase. Kommt es nun bei Cytosin zu einer solchen Desaminierung so entsteht Uracil. Würde Uracil auch gewöhnlich in der DNA vorkommen, könnte ein Enzym jetzt nicht mehr unterscheiden, ob das Uracil die falsche Nukleinbase ist oder das gegenüberliegende Guanin (das zuvor mit Cytosin paarte). In diesem Falle könnte eine wichtige Information verändert werden, eine Mutation könnte entstehen. Um dieser Verwechslung zu entgehen, wird in der DNA prinzipiell kein Uracil, sondern Thymin verwendet, Uracil wird in der DNA durch spezifische Enzyme, die Uracil-Glykosylasen erkannt und entfernt. Enzyme können Thymin aufgrund seiner zusätzlichen Methylgruppe einwandfrei erkennen und so ist klar, dass jedes Uracil in der DNA ein kaputtes Cytosin ist. In der RNA ist diese Gefahr der Informationsverfälschung nicht gravierend, da hier Informationen nur kurzfristig gespeichert werden und dazu nicht nur ein RNA-Molekül der jeweiligen Sorte, sondern hunderte vorhanden sind. Sollten einige davon defekt sein, so hat das keine gravierenden Auswirkungen auf den gesamten Organismus, da es genug Ersatz gibt.
Varianten
Es gibt zahlreiche Varianten der obigen Standard-Nukleinsäuren RNA und DNA. Teilweise sind diese natürlichen Ursprungs, darüber hinaus wurden aber auch im Rahmen der Xenobiologie Varianten entwickelt, deren Bausteine auf den ersten Blick gar nicht mehr als Ribo- (im Fall von RNA) oder Desoxyribonukleotide (im Fall von DNA) erkennbar sind. In einzelnen Fällen ist es bis heute Gegenstand der Diskussion, ob eine bestimmte Variante in der Natur vorkommt (oder etwa in der Anfangsphase des Lebens auf der Erde vorkam) oder nicht. Im Prinzip können alle drei Teile eines Nukleinsäurebausteins verändert sein, also:
- die Basen: Neben natürlich vorkommenden Nicht-Standard-Basen (im einfachsten Fall Uracil bei DNA) und Modifikationen (DNA-Methylierung) wurden u. a. folgende künstliche Varianten entwickelt:
- xDNA, xxDNA, yDNA, yyDNA (2006)
- Hachimoji-DNA (2019)
- die Zucker: Xenonukleinsäuren (XNA) haben statt Ribose oder Desoxyribose eine andere Gruppe, die ein anderer Zucker oder Zuckerderivat sein kann, aber nicht muss. Dies sind u. a.:
- die Phosphatgruppe
- Thiophosphat als Abwehrsystem SspABCD–SspE von Bakterien gegen Bakteriophagen,[16][17] und Fomivirsen (ein Phosphorthioat-Oligonukleotid)
- N3′-P5′-Phosphoramidate (NP)
- Morpholino-Phosphoramidate (MF)
- Desoxyadenosinmonoarsenat (dAMAs) siehe GFAJ-1 §Diskussion um den Einbau von Arsen in Biomoleküle: fraglicher Einbau in DNA bei der Halomonas-Spezies GFAJ-1 (siehe auch Halomonas titanicae)
- eine Kombination daraus und weitere spezielle Modifikationen:
- Peptid-Nukleinsäure (englisch Peptide Nucleic Acid, PNA, auch PNS)
- verbrückte Nukleinsäure (englisch locked nucleic acid, LNA) – in Gegensatz zu unverbrückter Nukleinsäure (englisch unlocked nucleic acid, UNA)
- RNA tritt in Lebewesen als D-RNA auf – L-RNA als sog. Spiegelmer kann allerdings synthetisiert werden. Gleiches gilt analog für DNA. L-DNA wird langsamer von Enzymen abgebaut als die natürliche Form, was sie für die Pharmaforschung interessant macht.[18][19]
Siehe auch
Literatur
- Hans Beyer, Wolfgang Walter: Lehrbuch der organischen Chemie. 23. Auflage. Hirzel, Stuttgart 1998, ISBN 3-7776-0808-4.
- Lubert Stryer: Biochemie. 5. Auflage. Spektrum Verlag, 2003, ISBN 3-8274-1303-6.
- Ralf Dahm: Das Molekül aus der Schlossküche. In: MaxPlanckForschung. Nr. 1, 2004, S. 50–55 (mpg.de (Memento vom 11. April 2011 im Internet Archive) [PDF; 1000 kB; abgerufen am 25. Dezember 2019]).
Einzelnachweise
- Eintrag zu Nucleinsäuren. In: Römpp Online. Georg Thieme Verlag, abgerufen am 17. Februar 2016.
- Ulrike Roll: Nukleinsäuren. In: Werner E. Gerabek, Bernhard D. Haage, Gundolf Keil, Wolfgang Wegner (Hrsg.): Enzyklopädie Medizingeschichte. De Gruyter, Berlin/New York 2005, ISBN 3-11-015714-4, S. 1060 f.; hier: S. 1060.
- A. Kossel: Über eine neue Base aus dem Thierkörper. Vortrag in Berichte der Deutschen Chemischen Gesellschaft. Heft 18, 1885, S. 79.
- A. Kossel: Weitere Beiträge zur Chemie des Zellkerns. In: Zeitschrift für physiologische Chemie. Band 10, 1886, S. 248.
- R. Altmann: Über Nucleinsäuren. In: Archiv für Anatomie und Physiologie, Physiologische Abteilung. Leipzig 1889, S. 524–536.
- A. Kossel: Über die chemische Zusammensetzung der Zelle. Vortrag. In: Archiv für Anatomie und Physiologie/Physiologische Abteilung 1891. S. 178.
- A. Kossel, A. Neumann: Über das Thymin, ein Spaltungsproduct der Nucleinsäure. In: Berichte der Deutschen Chemischen Gesellschaft. Band 26, 1893, S. 2753; Darstellung und Spaltungsproducte der Nucleinsäure (Adenylsäure). Vortrag. In: Berichte der Deutschen Chemischen Gesellschaft. Band 27, 1894, S. 2215; Über Nucleinsäure und Thyminsäure. In: Zeitschrift für physiologische Chemie. Band 22, 1896–97, S. 74.
- A. Kossel, H. Steudel: Weitere Untersuchungen über das Cytosin. In: Hoppe-Seyler’s Zeitschrift für physiologische Chemie. Band 38, 1903, S. 49.
- A. Kossel: Über die chemische Beschaffenheit des Zellkerns. Nobelvortrag am 12. Oktober 1910 in Stockholm. In: Münchener Medizinische Wochenschrift. Band 58, 1911, S. 65.
- P. Levene, E. London: The stucture of Thymonucleic acid. In: Journal of Biological Chemistry. 1929. 83. S. 793–802.
- Ramanarayanan Krishnamurthy, Eddy I. Jiménez, Clémentine Gibard: Prebiotic Phosphorylation and Concomitant Oligomerization of Deoxynucleosides to form DNA, in: Angewandte Chemie, 15. Dezember 2020, doi:10.1002/anie.202015910. Dazu:
- Discovery Supports a Surprising New View of How Life on Earth Originated, auf: SciTechDaily vom 28. Dezember 2020. Quelle: Scripps Research Institute
- Discovery boosts theory that life on Earth arose from RNA-DNA mix, auf: ScienceDaily vom 28. Dezember 2020. Quelle: Scripps Research Institute
- David Nield: There's Mounting Evidence That Life on Earth Started With More Than Just RNA, auf: sciencealert vom 1. Januar 2021
- Jianfeng Xu, Václav Chmela, Nicholas J. Green, David A. Russell, Mikołaj J. Janicki, Robert W. Góra, Rafał Szabla, Andrew D. Bond & John D. Sutherland: Selective prebiotic formation of RNA pyrimidine and DNA purine nucleosides, in: Nature Band 582, S. 60–66, 3. Juni 2020, doi:10.1038/s41586-020-2330-9. Dazu:
- Michael Marshall: First life on Earth may actually have been built from both RNA and DNA, auf: NewScientist vom 3. Juni 2020. Magazin: Band 3286 vom 13. Juni 2020
- Philippine Aupy, Lucía Echevarría, Karima Relizani, Fedor Svinartchouk, Luis Garcia, Aurélie Goyenvalle et al.: Identifying and Avoiding tcDNA-ASO Sequence-Specific Toxicity for the Development of DMD Exon 51 Skipping Therapy, in: Molecular Therapy – Nucleic Acids Band 19, S. 371–383, 6. März 2020, Online 26. November 2019, doi:10.1016/j.omtn.2019.11.020.
- Pradeep S. Pallan, Damian Ittig, Annie Héroux, Zdzislaw Wawrzak, Christian J. Leumannb, Martin Egli: Crystal structure of tricyclo-DNA: an unusual compensatory change of two adjacent backbone torsion angles, in: Chemical Communications, Band 7, 2008, doi:10.1039/B716390H.
- Damian Ittig, Anna-Barbara Gerber, Christian Joerg Leumann: Position-dependent effects on stability in tricyclo-DNA modified oligonucleotide duplexes, in: Nucleic Acids Research 39(1), S. 373–380, Januar 2011, doi:10.1093/nar/gkq733, PMID 20719742, PMC 3017593 (freier Volltext)
- Xiaolin Xiong et al.: SspABCD–SspE is a phosphorothioation-sensing bacterial defence system with broad anti-phage activities, in: Nature Microbiology Band 5, S. 917–928, 6. April 2020, doi:10.1038/s41564-020-0700-6, insbes. Fig. 1. Dazu:
- SMART researchers uncover new anti-phage defence mechanisms in bacteria, mit Image 1, auf: EurekAlert! vom 6. Mai 2020, Quelle: Singapore-MIT Alliance for Research and Technology (SMART)
- Hao Yu et al.: DNA backbone interactions impact the sequence specificity of DNA sulfur-binding domains: revelations from structural analyses, in: Nucleic Acids Research, Band 48, Nr. 15, 4. September 2020, S. 8755–8766, doi:10.1093/nar/gkaa574.
- W. Purschke, F. Radtke, F. Kleinjung, S. Klussmann: A DNA Spiegelmer to staphylococcal enterotoxin B, in: Nucleic Acids Research. Band 31, Nr. 12, 2003, S. 3027–3032, doi:10.1093/nar/gkg413, PMID 12799428.
- Gosuke Hayashi, Masaki Hagihara, Kazuhiko Nakatani: Application of L-DNA as a molecular tag, in: Nucleic Acids Symposium Series, Band 49, Nr. 1, 2005, S. 261–262, doi:10.1093/nass/49.1.261, PMID 17150733.