Thermal Shift Assay
Ein thermal shift assay (TSA, synonym Thermofluor Assay oder Differential Scanning Fluorimetry, DSF) ist eine biochemische Methode zur Messung der Änderung der Thermostabilität der Faltung eines Proteins nach Zugabe eines Bindungspartners.[1] Sie ist dadurch eine Methode zur Bestimmung von Protein-Protein-, Protein-DNA-[2] und Protein-Lipid-Interaktionen sowie Interaktionen zwischen Proteinen und niedermolekularen Verbindungen.[3][4]
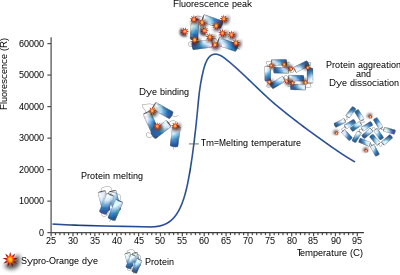
Prinzip
Die Faltung eines Proteins wird oftmals durch Bindung eines weiteren Moleküls stabilisiert. Die Stabilisierung äußert sich unter anderem durch eine erhöhte Thermostabilität des Proteins, bei der die biologische Aktivität des Proteins auch bei vergleichsweise höheren Temperaturen erhalten bleibt und die Denaturierung erst bei höheren Temperaturen einsetzt.[5][6]
Die Durchführung des Thermal Shift Assays erfolgt durch Zugabe von unterschiedlichen Verdünnungsstufen eines Bindungspartners. Zwei Messverfahren haben sich dabei durchgesetzt. Zum Einen ein indirektes Verfahren durch Zusatz eines fluoreszenten Proteinfarbstoffs wie z. B. SYPRO Orange oder 1-Anilinonaphthalen-8-sulfonsäure (ANS).[7] Die Versuchsansätze der Verdünnungsstufen und einer Negativkontrolle werden hierbei in einem Real-Time-Cycler inkubiert, der die Fluoreszenz in den Versuchsansätzen bei zunehmender Temperatur misst. Zum Anderen die direkte Messung der intrinsischen Fluoreszenz, der Eigenfluoreszenz, der Tryptophane (und in vermindertem Maße der Tyrosine), die auf Grund ihrer Polarität vorrangig im Innern einer Proteinstruktur verortet sind. Ein Bindungsereignis hat in der Regel eine Auswirkung auf die Struktur oder Faltung des Proteins, was eine Änderung der Positionen der Tryptophane, bzw. Tyrosine innerhalb der Struktur zur Folge haben kann.[8] Solch eine Positionsänderung verursacht eine Änderung der Fluoreszenz, welche durch die Änderung der Umgebungsbedingungen (Hydratation, Elektronendichte) begründet ist. Besonders deutlich wird dieser Effekt, wenn ein Tryptophan oder Tyrosin an dem Bindungsereignis beteiligt ist. Dadurch erhält man in vielen Fällen zusätzlich zur thermischen Stabilisierung noch Informationen zu strukturellen Änderungen während eines Bindungsvorganges.
Eine Variante der Methode namens CETSA (cellular thermal shift assay) verwendet Zellkulturen anstatt gereinigter Proteine, wodurch der erste Versuchsabschnitt, die Bindung eines Bindungspartners an ein Protein, in vivo erfolgen kann.[9][10] Die bei der Denaturierung verwendeten Puffer beeinflussen die Änderung der Thermostabilität eines Proteins.[11] Durch den vergleichsweise geringen Materialaufwand eignen sich Thermal Shift Assays für Messungen im Hochdurchsatz.[12]
Anwendungen
Thermal Shift Assays werden unter anderem bei der Proteincharakterisierung, beim Protein-Engineering und beim Wirkstoffdesign eingesetzt.[13]
Des Weiteren können Thermal Shift Assays genutzt werden, um geeignete Pufferbedingungen für ein Protein zu finden.[14]
Eine verwandte Methode, um optimale Pufferzusammensetzungen für ein Zielprotein oder Biopharmazeutikum zu identifizieren, ist nanoDSF. nanoDSF misst die Stabilität der Proteinfaltung in thermischen und chemischen Entfaltungsexperimenten. Dabei werden Änderungen der intrinsischen Fluoreszenz von Tryptophan während der Entfaltung detektiert.
Einzelnachweise
- F. H. Niesen, H. Berglund, M. Vedadi: The use of differential scanning fluorimetry to detect ligand interactions that promote protein stability. In: Nature protocols. Band 2, Nummer 9, 2007, ISSN 1750-2799, S. 2212–2221, doi:10.1038/nprot.2007.321, PMID 17853878.
- K. R. Rupesh, A. Smith, P. E. Boehmer: Ligand induced stabilization of the melting temperature of the HSV-1 single-strand DNA binding protein using the thermal shift assay. In: Biochemical and biophysical research communications. [elektronische Veröffentlichung vor dem Druck] Nummer 4, November 2014, ISSN 1090-2104, doi:10.1016/j.bbrc.2014.10.145, PMID 25449284, PMC 4254511 (freier Volltext).
- K. Huynh, C. L. Partch: Analysis of protein stability and ligand interactions by thermal shift assay. In: Current protocols in protein science / editorial board, John E. Coligan.. [et al.]. Band 79, 2015, ISSN 1934-3663, S. 28.9.1–28.9.14, doi:10.1002/0471140864.ps2809s79, PMID 25640896.
- S. N. Krishna, C. H. Luan, R. K. Mishra, L. Xu, K. A. Scheidt, W. F. Anderson, R. C. Bergan: A fluorescence-based thermal shift assay identifies inhibitors of mitogen activated protein kinase kinase 4. In: PloS one. Band 8, Nummer 12, 2013, ISSN 1932-6203, S. e81504, doi:10.1371/journal.pone.0081504, PMID 24339940, PMC 3855329 (freier Volltext).
- Daniel E. Koshland: Application of a Theory of Enzyme Specificity to Protein Synthesis. In: Proceedings of the National Academy of Sciences. Band 44, Nummer 2, Februar 1958, ISSN 0027-8424, S. 98–104, PMID 16590179, PMC 335371 (freier Volltext).
- K. Linderstrøm-Lang, J. A. Schellman: Protein structure and enzyme activity. In: The Enzymes (1959). Band 1, Heft 2, S. 443–510.
- J. J. Lavinder, S. B. Hari, B. J. Sullivan, T. J. Magliery: High-throughput thermal scanning: a general, rapid dye-binding thermal shift screen for protein engineering. In: Journal of the American Chemical Society. Band 131, Nummer 11, März 2009, ISSN 1520-5126, S. 3794–3795, doi:10.1021/ja8049063, PMID 19292479, PMC 2701314 (freier Volltext).
- Anders O. Magnusson, Anna Szekrenyi, Henk-Jan Joosten, James Finnigan, Simon Charnock: nanoDSF as screening tool for enzyme libraries and biotechnology development. In: The FEBS Journal. Band 286, Nr. 1, 2019, ISSN 1742-4658, S. 184–204, doi:10.1111/febs.14696.
- D. Martinez Molina, R. Jafari, M. Ignatushchenko, T. Seki, E. A. Larsson, C. Dan, L. Sreekumar, Y. Cao, P. Nordlund: Monitoring drug target engagement in cells and tissues using the cellular thermal shift assay. In: Science. Band 341, Nummer 6141, Juli 2013, ISSN 1095-9203, S. 84–87, doi:10.1126/science.1233606, PMID 23828940.
- R. Jafari, H. Almqvist, H. Axelsson, M. Ignatushchenko, T. Lundbäck, P. Nordlund, D. Martinez Molina: The cellular thermal shift assay for evaluating drug target interactions in cells. In: Nature protocols. Band 9, Nummer 9, September 2014, ISSN 1750-2799, S. 2100–2122, doi:10.1038/nprot.2014.138, PMID 25101824.
- L. Reinhard, H. Mayerhofer, A. Geerlof, J. Mueller-Dieckmann, M. S. Weiss: Optimization of protein buffer cocktails using Thermofluor. In: Acta crystallographica. Section F, Structural biology and crystallization communications. Band 69, Pt 2 Februar 2013, ISSN 1744-3091, S. 209–214, doi:10.1107/S1744309112051858, PMID 23385769, PMC 3564630 (freier Volltext).
- A. Ciulli, C. Abell: Fragment-based approaches to enzyme inhibition. In: Current Opinion in Biotechnology. Band 18, Nummer 6, Dezember 2007, ISSN 0958-1669, S. 489–496, doi:10.1016/j.copbio.2007.09.003, PMID 17959370.
- J. K. Kranz, C. Schalk-Hihi: Protein thermal shifts to identify low molecular weight fragments. In: Methods in enzymology. Band 493, 2011, ISSN 1557-7988, S. 277–298, doi:10.1016/B978-0-12-381274-2.00011-X, PMID 21371595.
- Reinhard L., Mayerhofer H., Geerlof A., Mueller-Dieckmann J., Weiss M.S.: Optimization of protein buffer cocktails using Thermofluor.. In: Acta Crystallogr Sect F Struct Biol Cryst Commun.. 69, Nr. 2, Januar 2013, S. 209–214. doi:10.1107/S1744309112051858. PMID 23385769.