Proteinsequenzierung
Die Proteinsequenzierung bezeichnet biochemische Methoden zur Bestimmung der Aminosäuresequenz von Proteinen oder Peptiden. Sie ist als Proteincharakterisierung von wesentlicher Bedeutung in der Proteomik.
De-Novo-Sequenzierung
Wenn keine Datenbank-Daten benutzt werden bzw. vorhanden sind, wird die Sequenzierung De-Novo genannt. Die Proteinsequenzierung per massenspektrometrischer Analyse erfolgt durch zusätzliche Fragmentierung der Peptide und einer Auftrennung in einem Reflektron.
Edman-Abbau
Der Edman-Abbau ist die klassische Methode zum Sequenzieren von Peptiden und Proteinen, er wird heutzutage aber nur noch selten verwendet. Im Edman-Abbau wird in einem zyklischen Prozess in jedem Reaktionszyklus die N-terminale Aminosäure derivatisiert, abgespalten und als Phenylthiohydantoin-Aminosäure per HPLC identifiziert.[1]
Statt des Edman-Abbaus wird das Protein aus massenspektrometrischen Fragmentspektren sequenziert. Der Vorteil von massenspektrometrischen Verfahren gegenüber dem Edman-Abbau liegen in erster Linie in einem geringeren Probenbedarf und einer deutlich verkürzten Analysezeit.[2] Außerdem kann die Probe auch in geringerer Reinheit vorliegen und es können auch N-terminal blockierte (z. B. durch Acetylierung, Formylierung) Peptide untersucht werden.
Schlack-Kumpf-Abbau
Beim Schlack-Kumpf-Abbau wird C-terminal sequenziert.
Nanoporen-Sequenzierung
Bei einem Durchtritt von einzelnen linearisierten Proteinen durch Nanoporen kann über die Veränderung der elektrischen Leitfähigkeit an der Nanopore die Aminosäureabfolge bestimmt werden. Diese Methode ist eine Weiterentwicklung der DNA-Sequenzierung mit Nanoporen.[3]
Protein-Identifikation
Indirekte Sequenzierung
Um die Aminosäuresequenz eines Proteins zu bestimmen, wird meist entweder die Gensequenz der DNA aus einer DNA-Sequenzierung oder aus einer Datenbank sequenzierter Genome wie tBLAST in silico in eine Proteinsequenz übersetzt. Durch molekulare Displays kann die Gensequenz eines Proteins erhalten werden. Daneben eignen sich auch noch massenspektrometrische Verfahren und der (eher historisch verwendete) Edman-Abbau zur direkten Identifizierung eines Proteins.
Massenspektrometrische Proteinanalytik
Die wichtigste Methode zur direkten Sequenzierung von Proteinen ist die Massenspektrometrie mit Hilfe des Peptidmassenfingerprint.[4] Ihre Bedeutung wuchs in den letzten Jahren stark, auch in Zusammenhang mit der sich verbessernden Computertechnik. Prinzipiell können mit Massenspektrometrie Proteine jeder Größe sequenziert werden, die Berechnung der Sequenz gestaltet sich jedoch mit zunehmender Größe immer schwieriger. Zudem sind Peptide aufgrund ihrer besseren Löslichkeit leichter zu präparieren. Das Protein wird daher zunächst mit einer Endoprotease verdaut und die resultierende Lösung durch HPLC separiert.
Im Massenspektrometer müssen die Peptide zunächst ionisiert werden. Eine der beiden häufig verwendeten Methoden zur Ionisation ist die Elektrospray-Ionisation, die andere die Matrix-unterstützte Laserdesorption/Ionisation (MALDI). Bei erster wird die Lösung durch eine dünne Düse mit hohem, positivem Potential (positiver Ladung) ins Massenspektromer gesprüht. Die Tröpfchen zerfallen im Vakuum, bis nur noch einzelne Ionen vorliegen. Die Peptide fragmentieren dann, das Masse-zu-Ladung-Verhältnis der Fragmente wird gemessen. Das resultierende Fragment-Spektrum wird mit Programmen analysiert und mit Datenbanken abgeglichen. Der Prozess wird meist anschließend mit einem anders verdauten Protein wiederholt, um aus den Fragmenten das ursprüngliche Protein rekonstruieren zu können.
Für die Analyse der Fragment-Spektren ist hilfreich, dass Fragmentierungen in erster Linie an den Peptidbindungen auftreten (b- und y-Fragmente in der folgenden Abbildung):
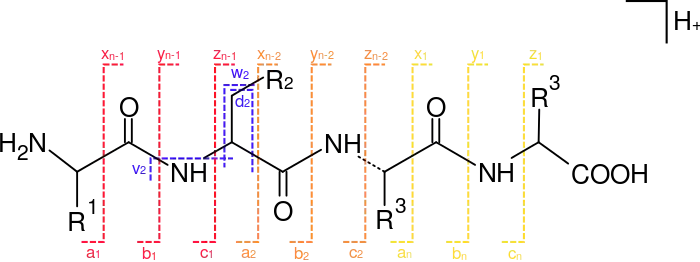
Die Abbildung stellt verschiedene Möglichkeiten für die Bildung von Fragmenten aus einem (protonierten) Peptid bestehend aus n Aminosäuren dar. Die Nomenklatur der verschiedenen Fragmente – z. B. b1-Fragment oder y4-Fragment (= yn-1-Fragment bei n=4 Aminosäuren) – geht auf Vorschläge von Roepstorff, Fohlman und Biemann zurück.[5][6] Die Fragmente der N-terminalen Serie werden mit a, b und c bezeichnet, die der C-terminalen Serie mit x, y und z. Im Index wird zudem die Zahl der enthaltenen Aminosäuren angegeben. Die seltener auftretenden Fragmente der Aminosäure-Seitenketten werden mit d, v und w gekennzeichnet. Letztere Fragmente können die Unterscheidung von Leucin und Isoleucin in der Kette ermöglichen.[7]
Einzelnachweise
- Pehr Edman, Geoffrey Begg: A protein sequenator. In: European Journal of Biochemistry. 1967, S. 80-91. doi:10.1111/j.1432-1033.1967.tb00047.x.
- M. Wilm, Shevchenko, A., Houthaeve, T., Breit, S., Schweigerer, L., Fotsis, T., & Mann, M.: Femtomole sequencing of proteins from polyacrylamide gels by nano-electrospray mass spectrometry. In: Nature. 379, Nr. 6564, 1996, S. 466-469. doi:10.1038/379466a0.
- Y. Yang, R. Liu, H. Xie, Y. Hui, R. Jiao, Y. Gong, Y. Zhang: Advances in nanopore sequencing technology. In: Journal of nanoscience and nanotechnology. Band 13, Nummer 7, Juli 2013, ISSN 1533-4880, S. 4521–4538, PMID 23901471.
- Joshua J. Coon: Collisions or Electrons? Protein Sequence Analysis in the 21st Century. In: Anal. Chem.. 81, Nr. 9, 13. April 2009, S. 3208–3215. doi:10.1021/ac802330b.
- P. Roepstorff, Fohlman: Proposal for a common nomenclature for sequence ions in mass spectra of peptides. Biomed Mass Spectrom. In: Biomedical Mass Spectrometry. 11, Nr. 11, 1984, S. 601. doi:10.1002/bms.1200111109.
- Biemann, K. (1992) Mass spectrometry of peptides and proteins. Annu Rev Biochem 61: 977-1010.
- Christian Schmelzer: Massenspektrometrische Charakterisierung von Proteinhydrolysaten: Verdaustudien an beta-Casein und Strukturuntersuchungen an Elastin. 2007 (uni-halle.de [PDF]).
Literatur
- Friedrich Lottspeich, Joachim W. Engels, Solodkoff, Zettlmeier, Lay: Bioanalytik. 3. Auflage, Spektrum, Heidelberg, 2012, ISBN 978-3-8274-0041-3.
- Hubert Rehm, Thomas Letzel: Der Experimentator: Proteinbiochemie / Proteomics. 6. Auflage, Spektrum, Heidelberg 2009, ISBN 978-3-8274-2312-2.