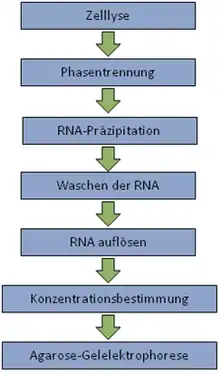
RNA-Extraktion
Die RNA-Extraktion umfasst biochemische Methoden zur Extraktion von RNA aus Zellen. Die isolierte RNA kann in zahlreichen Methoden zur Analyse der Genexpression genutzt werden.[1] Die RNA-Extraktion ist eine der Methoden zur RNA-Reinigung.
RNasen
RNA ist im Vergleich zu DNA relativ empfindlich für abbauende Enzyme (bei RNA sind das RNasen), die ubiquitär vorkommen.[2][3] Zudem wird RNA in Zellen zügig abgebaut, weshalb Zellen zur späteren RNA-Extraktion bei −80 °C gelagert werden.[3] RNasen sind Enzyme, die RNA in kleinere Fragmente spalten. Sie katalysieren die Mg2+-unabhängige Hydrolyse von Phosphodiesterbindungen im Phosphatrückgrat der RNA. RNasen besitzen eine hohe Thermostabilität und können auch nach Autoklavierung noch aktiv sein. Deshalb erfordert der Umgang mit RNA mehr Sorgfalt als das Arbeiten mit der sehr viel stabileren DNA. Um RNA-Abbau zu vermeiden, sollten RNA und RNasen frühzeitig voneinander getrennt werden und verhindert werden, RNasen aus der Umgebung in die Probe einzubringen. Darum sollten Einweghandschuhe getragen werden, weil RNasen von allen Organismen produziert werden und daher auch im menschlichen Schweiß auf der Hautoberfläche und in Staubpartikeln vorhanden sind. Außerdem ist es sinnvoll, einen bestimmten Satz an Verbrauchsmaterialien (Pipetten, Pipettenboxen usw.) nur für RNA-Versuche zu verwenden. Zudem sollte spezielles RNase-freies Wasser verwendet werden (DEPC-Wasser).
Zellaufschluss
Beim Zellaufschluss werden RNasen zügig denaturiert,[3] meist durch Chaotrope wie Guanidiniumthiocyanat. Oftmals wird dem Aufschlusspuffer zusätzlich ein Thiol wie z. B. 10 millimolar Mercaptoethanol oder Dithiothreitol hinzugefügt, um RNasen durch die Spaltung ihrer Disulfidbrücken zu inaktivieren.[4][5] Anschließend wird die RNA von den übrigen zellulären Bestandteilen getrennt. Auf diese Weise erhält man Gesamt-RNA. Diese Gesamt-RNA lässt sich entweder direkt für weitere Experimente benutzen (z. B. Northern Blot, Reverse Transkription in cDNA), oder sie kann als Ausgangssubstanz für die Isolierung von mRNA verwendet werden.
Extraktionen
Die meisten RNA-Extraktionen basieren nach dem Zellaufschluss auf drei unterschiedlichen Verfahren, der Zwei-Phasen-Extraktion,[3] oder der Fällung, letztere eventuell mit zusätzlicher selektiver Adsorption an eine RNA-bindende Matrix.[6][7] Die RNA-Extraktionsverfahren ähneln denen der DNA-Extraktion. Die Verfahren werden zum Teil auch miteinander kombiniert.[8] Meistens folgt auf eine der Methoden eine abschließende Isopropanol- oder Ethanolfällung.
Phenol-Chloroform-Isoamylalkohol-Extraktion
Nach einem Zellaufschluss kann eine RNA-Extraktion mit einer Lösung aus Phenol, Chloroform und Isoamylalkohol in einem Volumenverhältnis von 25:24:1 durchgeführt werden. Dabei bilden sich nach Zugabe zu den Zellen zwei Phasen, eine wässrige und eine organische Phase. Die RNA sammelt sich in der wässrigen Phase. Anschließend wird die wässrige Phase einer Isopropanol- oder Ethanolfällung der RNA unterzogen.
Trizol-Extraktion
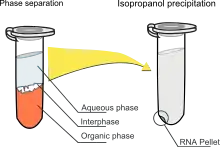
Bei dieser Methode nach Piotr Chomczynski und Nicoletta Sacchi wird mit einem speziellen Reagenz (Handelsnamen und Varianten sind z. B. Trizol, TRI Reagent, Trisure, TriFast, STAT-60, RNAzol oder DNAzol) gearbeitet.[9] Damit ist es möglich, Zellen zu lysieren und gleichzeitig RNA und DNA aus Zellen oder Geweben zu gewinnen. Dieses Verfahren basiert auf der sogenannten single-step-Methode nach Chomczynski und Sacchi. Trizol enthält neben Phenol und Chloroform Guanidiniumthiocyanat, wodurch die Zellen lysiert und gleichzeitig RNasen und andere Enzyme inaktiviert werden. Wenn zu wenig Trizol im Verhältnis zur Probe verwendet wird, verschieben sich Ionenstärke und pH-Wert, wodurch die Reinheit der isolierten RNA gesenkt wird.[3] Bei Blut, Blutplasma und Zellkulturmedium können aufgrund ihrer höheren Pufferwirkung Verschiebungen des pH-Werts auftreten.[3]
Nach Zugabe des Chloroforms bzw. des Trizols sind drei Phasen zu erkennen. Die obere wässrige Phase enthält RNA, die Interphase DNA und die untere Chloroformphase Proteine. Die RNA in der wässrigen Phase wird anschließend mit Isopropanol oder Ethanol präzipitiert. Nach zwei Waschschritten wird die RNA wird der Niederschlag getrocknet. Zu starkes Trocknen verhindert allerdings, dass die RNA vollständig in Lösung geht nach Zugabe von RNase-freiem Wasser (DEPC-Wasser).[3] Aufgrund der Verwendung giftiger und flüchtiger Chemikalien ist eine Laborabzugshaube notwendig. Im Vergleich zur Festphasenadsorption ist die Zwei-Phasen-Extraktion kostengünstiger, aber sie dauert mit 4 Stunden länger.
„Nonidet P-40“-Methode
Diese Methode ist nicht für Gewebe geeignet und dient zur Gewinnung von mRNA aus dem Zytosol. Der Vorteil dieser Methode besteht darin, dass die Zellkerne intakt bleiben und somit zusätzlich noch die Möglichkeit besteht DNA zu isolieren. Das Prinzip beruht auf dem nichtionischen Detergenz Nonidet P-40 (synonym Triton-X100, Octoxinol 9), welches zu den Zellen gegeben wird und sich die DNA (Zellkerne) nach der Zentrifugation als Pellet absetzt. RNA, Proteine und Zelltrümmer bleiben in Lösung. Anschließend erfolgt wie bei der single-step-Methode die Isolierung mit Phenol/Chloroform.
Adsorption an feste Phasen
Als weitere Möglichkeit der RNA-Isolierung stehen viele Kit-Systeme der Festphasenextraktion von verschiedenen Firmen und in zahlreichen Ausführungen zur Verfügung. Bei diesen Kit-Systemen verwendet man kleine Säulchen, die RNA spezifisch binden. Das Säulenbett bzw. die RNA-bindende Matrix besteht oftmals aus Kieselgel. Für die Reinigung von mRNA aus RNA können Oligo-dT-Säulen verwendet werden, die den Poly-A-Schwanz von mRNA binden.[10]
Catrimox-14-Extraktion
RNA und DNA bilden mit manchen kationischen Tensiden unlösliche Komplexe.[11] Bei der Catrimox-14-Extraktion werden die Zellen mit dem kationischen Tensid Catrimox-14 (Tetradecyltrimethylammoniumoxalat) und Guanidiniumthiocyanat lysiert. Nach einer Zentrifugation und einem Waschschritt erfolgt eine Extraktion mit Phenol, Chloroform und Isoamylalkohol (25:24:1 Vol.). Die wässrige Phase wird anschließend einer Ethanolfällung unterzogen.[12][13] Analog erfolgt die CTAB-Methode zur RNA- oder DNA-Extraktion,[14] welche bei pflanzlichen Ausgangsmaterial eine höhere Reinheit und weniger RNA-Abbau aufweist als die Single-Step-Methode.[15]
Konzentrationsbestimmung per Photometrie
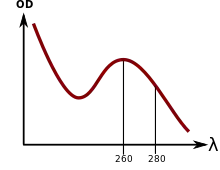
Bei der photometrischen Konzentrationsbestimmung misst man die Extinktion bei λ=260 nm (auch Absorption, A260, Optische Dichte, OD260), dem lokalen Absorptionsmaximum von Nukleinsäuren (DNA, RNA), und bei λ=280 nm (OD280), dem lokalen Absorptionsmaximum von Proteinen. Ob die Probe mit genomischer DNA oder Proteinen verunreinigt ist, kann durch den Quotienten aus OD260 und OD280 ermittelt werden. Bei reiner RNA sollte das Verhältnis ungefähr bei 2,0 liegen. Liegt der Wert unterhalb ist die Probe mit Protein, genomischer DNA und/oder aromatischen Substanzen kontaminiert. In diesem Fall sollte die RNA erneut gereinigt werden. Da eine OD260 von 1 dabei 40 µg/ml RNA entspricht, lässt sich die RNA-Konzentration mit folgender Formel berechnen:
Konzentration [µg/ml] = OD260 × 40 µg/ml × Verdünnungsfaktor
RNA-Integrität per Agarose-Gelelektrophorese
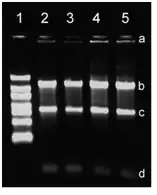
a: genomische DNA
b: 28S-rRNA
c: 18S-rRNA
d: 5S-rRNA
Mit Hilfe der Agarose-Gelelektrophorese können Nukleinsäuren ihrer Größe nach aufgetrennt werden, wobei kleine Fragmente schneller wandern als größere. Die Methode basiert auf den Wanderungseigenschaften der Nukleinsäuren, die durch ihre negativ geladenen Phosphatgruppen bei angelegter elektrischer Spannung in Richtung Anode (Pluspol) wandern. Dafür wird ein Agarosegel verwendet, das anschließend durch verschiedene Farbstoffe (z. B. Methylenblau) angefärbt werden kann. Dadurch kann die RNA sichtbar gemacht und fotografiert werden. Bei intakter RNA sind bei dieser Probe im Gel zwei deutlich getrennte Banden, die 28S- und 18S-Bande ribosomaler RNA zu erkennen. Das 2:1-Verhältnis der Fluoreszenzintensitäten der 28S- und 18S-rRNA-Bande ist ein Zeichen dafür, dass die mRNA nicht abgebaut ist. Die 5S-Bande ist meist kaum oder gar nicht zu sehen.
Geschichte
Die Geschichte der RNA-Extraktion ist mit der Entwicklung der DNA-Extraktion verbunden, da beide chemische Eigenschaften der Nukleinsäuren aufweisen.
Literatur
- C Wagener, O Müller: Molekulare Onkologie: Entstehung, Progression, klinische Aspekte. 3., komplett aktualisierte und erw. Aufl. Thieme, Stuttgart [u. a.] 2009. ISBN 978-3131035134.
- Friedrich Lottspeich, Haralabos Zorbas (Hrsg.): Bioanalytik. Spektrum Akademischer Verlag, Heidelberg u. a. 1998, ISBN 3-8274-0041-4.
- Cornel Mülhardt: Der Experimentator: Molekularbiologie/Genomics. Sechste Auflage. Spektrum Akademischer Verlag, Heidelberg 2008, ISBN 3-82-742036-9.
- J. Sambrook, T. Maniatis, D. W. Russel: Molecular cloning: a laboratory manual. 3rd edition (2001), Cold Spring Harbor Laboratory Press. ISBN 0-87969-577-3.
Einzelnachweise
- L. V. Madabusi, G. J. Latham, B. F. Andruss: RNA extraction for arrays. In: Methods in enzymology. Band 411, 2006, S. 1–14, doi:10.1016/S0076-6879(06)11001-0, PMID 16939782.
- S. C. Tan, B. C. Yiap: DNA, RNA, and protein extraction: the past and the present. In: Journal of biomedicine & biotechnology. Band 2009, 2009, S. 574398, doi:10.1155/2009/574398, PMID 20011662, PMC 2789530 (freier Volltext).
- Piotr Chomczynski, William Wilfinger, Karol Mackey: Single-Step Method of Total RNA Isolation by Guanidine–Phenol Extraction. In: eLS (2013). doi:10.1002/9780470015902.a0003799.pub2.
- K. Mommaerts, I. Sanchez, F. Betsou, W. Mathieson: Replacing β-mercaptoethanol in RNA extractions. In: Analytical biochemistry. Band 479, Juni 2015, S. 51–53, doi:10.1016/j.ab.2015.03.027, PMID 25841674.
- S. E. Zale, A. M. Klibanov: Why does ribonuclease irreversibly inactivate at high temperatures? In: Biochemistry. Band 25, Nummer 19, September 1986, S. 5432–5444, PMID 3778869.
- S. N. Peirson, J. N. Butler: RNA extraction from mammalian tissues. In: Methods in molecular biology (Clifton, N.J.). Band 362, 2007, S. 315–327, doi:10.1007/978-1-59745-257-1_22, PMID 17417019.
- I. E. Jordon-Thaden, A. S. Chanderbali, M. A. Gitzendanner, D. E. Soltis: Modified CTAB and TRIzol protocols improve RNA extraction from chemically complex Embryophyta. In: Applications in plant sciences. Band 3, Nummer 5, Mai 2015, S. , doi:10.3732/apps.1400105, PMID 25995975, PMC 4435465 (freier Volltext).
- I. M. Bird: Extraction of RNA from cells and tissue. In: Methods in molecular medicine. Band 108, 2005, S. 139–148, PMID 16028681.
- P. Chomczynski, N. Sacchi: Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. In: Anal Biochem. (1987) 162(1):156-9. PMID 2440339.
- I. M. Bird: Extraction of RNA from cells and tissue. In: Methods in molecular medicine. Band 108, 2005, S. 139–148, doi:10.1385/1-59259-850-1:139, PMID 16028681.
- S. K. Dutta, A. S. Jones, M. Stacey: The separation of desoxypentosenucleic acids and pentosenucleic acids. In: Biochimica et Biophysica Acta. Band 10, Nummer 4, April 1953, S. 613–622, PMID 13059025.
- C. E. Dahle, D. E. Macfarlane: Isolation of RNA from cells in culture using Catrimox-14 cationic surfactant. In: BioTechniques. Band 15, Nummer 6, Dezember 1993, S. 1102–1105, PMID 8292344.
- C. E. Dahle, D. E. Macfarlane: Isolating RNA with the cationic surfactant, Catrimox-14. In: Methods in molecular biology (Clifton, N.J.). Band 86, 1998, S. 19–21, doi:10.1385/0-89603-494-1:19, PMID 9664447.
- Yolanda M. Camacho-Villasana, Neftali Ochoa-Alejo, Linda Walling, Elizabeth A. Bray: An improved method for isolating RNA from dehydrated and nondehydrated chili pepper (Capsicum annuum L.) plant tissues. In: Plant Molecular Biology Reporter. 20, 2002, S. 407, doi:10.1007/BF02772128.
- K. Shahrokhabadi, R.T. Afshari, H. Alizade, J.T. Afshari, G.R. Javadi: Compared Two Methods for Isolating RNA from Freezing and Nonfreezing Bread Wheat (Triticum aestivum L. ) Plant Tissues. In: Asian Journal of Plant Sciences. 7, 2008, S. 505, doi:10.3923/ajps.2008.505.509.