DNA-bindende Proteine
Eigenschaften
DNA-bindende Proteine kommen in allen Lebewesen und DNA-Viren vor. Sie besitzen mindestens eine Proteindomäne, welche an DNA binden kann. Die Bindung kann dabei an verschiedenen funktionellen Gruppen erfolgen. Das Rückgrat der DNA besteht aus sich abwechselnden Phosphat- und Desoxyribose-Einheiten. Durch die Phosphatgruppen ist die DNA proportional zur Kettenlänge mit negativen Ladungen versehen, an die unter anderem Proteindomänen mit positiv-geladenen Aminosäuren (z. B. Lysin, Arginin) oder Proteindomänen mit negativ geladenen Aminosäuren mit komplexierten Kationen binden können (z. B. Mg2+- oder Zn2+-Komplexe).[4] Da das Desoxyribosephosphat-Rückgrat sich ständig wiederholt, kann ein ausschließlich an das Rückgrat bindendes Protein nicht an eine bestimmte DNA-Sequenz binden (keine Sequenzspezifität). Eine sequenzspezifische Bindung erfolgt durch zumindest teilweise Bindung an eine bestimmte Folge von Nukleinbasen, teilweise kann auch das Rückgrat gebunden werden.
Sequenzspezifität
DNA-bindende Proteine ohne Sequenzspezifität sind z. B. die Polymerasen, Helicasen und generell Proteine, die an der DNA entlanggleiten (z. B. mit DNA-Klammer). Sequenzspezifisch DNA-bindende Proteine sind z. B. Transkriptionsfaktoren, die an definierten Stellen (Promotor) eine Genexpression auslösen.[5] Aufgrund des höheren Anteils an Nukleinbasen in der Oberfläche binden sequenzspezifische DNA-bindende Moleküle eher in der großen Furche der DNA-Doppelhelix.[6] Während manche sequenzspezifisch DNA-bindende Proteine bevorzugt einzelsträngige DNA binden (z. B. Einzelstrang-bindendes Protein), binden andere doppelsträngige DNA (die meisten) und einige wenige auch Heteroduplexe aus DNA und RNA (z. B. Telomerase, Reverse Transkriptasen, RNase H).
Bindung einzelsträngiger DNA
Einzelsträngige DNA kommt in Eukaryoten dauerhaft an den Telomeren vor und vorübergehend unter anderem bei der Replikation, der Transkription, der Rekombination und der DNA-Reparatur vor,[7] z. B. das Einzelstrang-bindende Protein und manche DNA-Reparaturenzyme.[8][9]
Bindung doppelsträngiger DNA
Doppelsträngige DNA mit komplementärer Basenpaarung bildet eine Doppelhelix aus (B-DNA). Diese DNA-Doppelhelix besitzt eine große und eine kleine Furche. Die kleine Furche besitzt einen kleineren Anteil der Nukleinbasen in der Oberfläche des Moleküls, weshalb sie sich weniger für eine Sequenz-spezifische Bindung eignet. Verschiedene DNA-bindende Moleküle wie Lexitropsine, Netropsin,[10] Distamycin, Hoechst 33342, Pentamidin, DAPI oder SYBR Green I binden sequenzunabhängig an die kleine Furche doppelsträngiger DNA.[11] Doppelstrang-bindende Proteine sind unter anderem Histone[12] bzw. DNA-bindendes Protein H-NS,[13] High-Mobility-Group-Proteine,[14][15] DNA-Polymerasen, DNA-abhängige RNA-Polymerasen, Helicasen, Topoisomerasen, Gyrasen, Ligasen, Polynukleotid-Kinasen, Nukleasen, manche DNA-Reparaturenzyme. Sequenzspezifisch dsDNA-bindende Proteine sind unter anderem Transkriptionsfaktoren und manche Endonukleasen.[16][17] Prokaryotische Transkriptionsfaktoren sind meistens kleiner als die von ihnen kontrollierten Genexpressionsprodukte, während eukaryotische Transkriptionsfaktoren meistens größer als deren kontrollierten Produkte sind und gelegentlich mehrere Kopien einer DNA-bindenden Domäne besitzen.[18]
DNA-bindende Proteindomänen
Typische Proteindomänen bei dsDNA-bindenden Proteinen sind die Zinkfingerdomäne, die AT-Haken, die DNA-Klammer und das Helix-Turn-Helix-Motiv zur DNA-Bindung oder die Leucinzipperdomäne (bZIP) zur Dimerisierung. Bei einzelsträngiger DNA wurde unter anderem die OB-Faltungsdomäne beschrieben.[19]
Identifikation
Methoden zur Bestimmung von Protein-DNA-Wechselwirkungen (gebundene DNA-Sequenz, DNA-bindende Proteine) sind z. B. EMSA, DNase Footprinting Assay, Filterbindungstest, DPI-ELISA, DamID, SELEX, ChIP, ChIP-on-Chip oder ChIP-Seq und DAP-Seq.[20][21]
Modellierung
Verschiedene Ansätze zur molekularen Modellierung wurden beschrieben.[22][23] Ebenso wurden Algorithmen zur Bestimmung der gebundenen DNA-Sequenz entwickelt.[24]
Modifikation
Im Zuge eines Proteindesigns können z. B. Zinkfingerproteine oder TALENs entworfen werden. Mit der CRISPR/Cas-Methode können anhand einer komplementären RNA-Sequenz entsprechende DNA-Sequenzen gebunden werden.
Formen
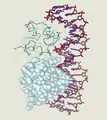 Cro im Komplex mit DNA
Cro im Komplex mit DNA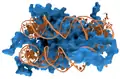 DNA (orange) mit Histonen (blau)
DNA (orange) mit Histonen (blau)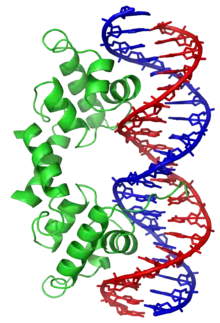

 DNA-Klammer
DNA-Klammer
Weblinks
- Abalone tool for modeling DNA-ligand interactions.
- DBD database of predicted transcription factors Uses a curated set of DNA-binding domains to predict transcription factors in all completely sequenced genomes
- MeSH DNA-bindende Proteine
Einzelnachweise
- A. A. Travers: DNA-protein interactions. Springer, London 1993, ISBN 978-0-412-25990-6.
- C. O. Pabo, R. T. Sauer: Protein-DNA recognition. In: Annu. Rev. Biochem. Band 53, Nr. 1, 1984, S. 293–321, doi:10.1146/annurev.bi.53.070184.001453, PMID 6236744.
- R. E. Dickerson: The DNA helix and how it is read. In: Sci Am. Band 249, Nr. 6, 1983, S. 94–111, doi:10.1038/scientificamerican1283-94.
- K. Luger, A. Mäder, R. Richmond, D. Sargent, T. Richmond: Crystal structure of the nucleosome core particle at 2.8 A resolution. In: Nature. Band 389, Nr. 6648, 1997, S. 251–260, doi:10.1038/38444, PMID 9305837.
- Z. Li, S. Van Calcar, C. Qu, W. Cavenee, M. Zhang, B. Ren: A global transcriptional regulatory role for c-Myc in Burkitt's lymphoma cells. In: Proc Natl Acad Sci USA. Band 100, Nr. 14, 2003, S. 8164–8169, doi:10.1073/pnas.1332764100, PMID 12808131, PMC 166200 (freier Volltext).
- C. Pabo, R. Sauer: Protein-DNA recognition. In: Annu Rev Biochem. Band 53, Nr. 1, 1984, S. 293–321, doi:10.1146/annurev.bi.53.070184.001453, PMID 6236744.
- T. H. Dickey, S. E. Altschuler, D. S. Wuttke: Single-stranded DNA-binding proteins: multiple domains for multiple functions. In: Structure (London, England : 1993). Band 21, Nummer 7, Juli 2013, S. 1074–1084, doi:10.1016/j.str.2013.05.013. PMID 23823326. PMC 3816740 (freier Volltext).
- C. Iftode, Y. Daniely, J. Borowiec: Replication protein A (RPA): the eukaryotic SSB. In: Crit Rev Biochem Mol Biol. Band 34, Nr. 3, 1999, S. 141–180, doi:10.1080/10409239991209255, PMID 10473346.
- J. Kur, M. Olszewski, A. Dugoecka, P. Filipkowski: Single-stranded DNA-binding proteins (SSBs) – sources and applications in molecular biology. In: Acta biochimica Polonica. Band 52, Nummer 3, 2005, S. 569–574. PMID 16082412.
- C. Zimmer, U. Wähnert: Nonintercalating DNA-binding ligands: specificity of the interaction and their use as tools in biophysical, biochemical and biological investigations of the genetic material. In: Prog. Biophys. Mol. Biol. Band 47, Nr. 1, 1986, S. 31–112, doi:10.1016/0079-6107(86)90005-2, PMID 2422697.
- P. B. Dervan: Design of sequence-specific DNA-binding molecules. In: Science. Band 232, Nr. 4749, April 1986, S. 464–471, doi:10.1126/science.2421408, PMID 2421408.
- K. Sandman, S. Pereira, J. Reeve: Diversity of prokaryotic chromosomal proteins and the origin of the nucleosome. In: Cell Mol Life Sci. Band 54, Nr. 12, 1998, S. 1350–1364, doi:10.1007/s000180050259, PMID 9893710.
- R. T. Dame: The role of nucleoid-associated proteins in the organization and compaction of bacterial chromatin. In: Mol. Microbiol. Band 56, Nr. 4, 2005, S. 858–870, doi:10.1111/j.1365-2958.2005.04598.x, PMID 15853876.
- J. Thomas: HMG1 and 2: architectural DNA-binding proteins. In: Biochem Soc Trans. Band 29, Pt 4, 2001, S. 395–401, doi:10.1042/BST0290395, PMID 11497996.
- R. Grosschedl, K. Giese, J. Pagel: HMG domain proteins: architectural elements in the assembly of nucleoprotein structures. In: Trends Genet. Band 10, Nr. 3, 1994, S. 94–100, doi:10.1016/0168-9525(94)90232-1, PMID 8178371.
- L. Myers, R. Kornberg: Mediator of transcriptional regulation. In: Annu Rev Biochem. Band 69, Nr. 1, 2000, S. 729–749, doi:10.1146/annurev.biochem.69.1.729, PMID 10966474.
- B. Spiegelman, R. Heinrich: Biological control throughs regulated transcriptional coactivators. In: Cell. Band 119, Nr. 2, 2004, S. 157–167, doi:10.1016/j.cell.2004.09.037, PMID 15479634.
- V. Charoensawan, D. Wilson, S. A. Teichmann: Genomic repertoires of DNA-binding transcription factors across the tree of life. In: Nucleic acids research. Band 38, Nummer 21, November 2010, S. 7364–7377, doi:10.1093/nar/gkq617. PMID 20675356. PMC 2995046 (freier Volltext).
- N. W. Ashton, E. Bolderson, L. Cubeddu, K. J. O'Byrne, D. J. Richard: Human single-stranded DNA binding proteins are essential for maintaining genomic stability. In: BMC molecular biology. Band 14, 2013, S. 9, doi:10.1186/1471-2199-14-9. PMID 23548139. PMC 3626794 (freier Volltext).
- H. L. Brand, S. B. Satbhai, H. Ü. Kolukisaoglu, D. Wanke: Limits And Prospects Of Methods For The Analysis Of DNA-Protein Interaction. Bentham eBook, S. 124–148, abgerufen am 21. Oktober 2020 (englisch).
- M. F. Carey, C. L. Peterson, S. T. Smale: Experimental strategies for the identification of DNA-binding proteins. In: Cold Spring Harbor protocols. Band 2012, Nummer 1, Januar 2012, S. 18–33, doi:10.1101/pdb.top067470. PMID 22194258.
- V. B. Teif, K. Rippe: Statistical-mechanical lattice models for protein-DNA binding in chromatin. In: Journal of Physics: Condensed Matter. 2010, arxiv:1004.5514.
- K. C. Wong, T. M. Chan, C. Peng, Y. Li, Z. Zhang: DNA Motif Elucidation using belief propagation. In: Nucleic Acids Research. Advanced Online June 2013; doi:10.1093/nar/gkt574. PMID 23814189
- G. D. Stormo: DNA binding sites: representation and discovery. In: Bioinformatics. Band 16, Nummer 1, Januar 2000, S. 16–23. PMID 10812473.
- Created from PDB 1LMB
- Created from PDB 1RVA