Proteinbiosynthese
Proteinbiosynthese (PBS) ist die Neubildung von Proteinen in Zellen. Bei diesem für alle Lebewesen zentralen Prozess wird nach Vorgabe genetischer Information ein Protein aus Aminosäuren aufgebaut.
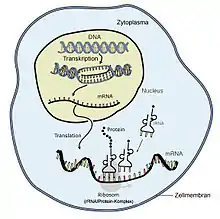
Die Synthese eines Proteins aus seinen Bausteinen, den proteinogenen Aminosäuren, findet im Rahmen der Genexpression an den Ribosomen statt. Die ribosomale Proteinsynthese wird auch als Translation bezeichnet, da hierbei die Basenfolge einer messenger-RNA (mRNA) in die Abfolge von Aminosäuren eines Peptids übersetzt wird. Dies geschieht, indem fortlaufend jedem Codon der mRNA das entsprechende Anticodon einer transfer-RNA (tRNA) zugeordnet wird und deren jeweils einzeln transportierte Aminosäure an die benachbarte gebunden wird (Peptidbindung), sodass eine Kette mit charakteristischer Aminosäuresequenz entsteht. Dieses Polypeptid kann sich im umgebenden Medium zu einem strukturierten Gebilde dreidimensionaler Form auffalten, dem nativen Protein. Häufig wird es durch Abspaltungen, Umbauten und Anbauten danach noch verändert, posttranslational modifiziert.
Während bei prokaryoten Zellen (Procyten) die ringförmige DNA frei im Zytosol vorliegt und die ribosomale Proteinsynthese zumeist unmittelbar und prompt mit der gerade eben erstellten mRNA erfolgt, sind die Verhältnisse bei eukaryoten Zellen (Eucyten) komplizierter. Für das auf mehrere Chromosomen verteilte Genom ist hier mit dem Zellkern (Nukleus) ein eigenes Kompartiment geschaffen, in dessen Karyoplasma auch die Transkription stattfindet. Die primär gezogene RNA-Kopie (hnRNA) wird zunächst stabilisiert, überarbeitet und auf den Kernexport vorbereitet, bevor sie als mRNA eine Kernpore passiert und ins Zytoplasma gelangt, das die Untereinheiten der Ribosomen enthält. Diese räumliche Aufteilung und der mehrschrittige Prozessweg erlauben somit zusätzliche Weisen, eine (hn)RNA posttranskriptional zu modifizieren und darüber die Genexpression zu regulieren beziehungsweise bestimmte RNA-Vorlagen von der Proteinbiosynthese auszuschließen (Gen-Stilllegung).
Einige Arten von Bakterien, Archaeen und Pilzen können über ribosomale Proteinsynthese besondere Proteine aufbauen, die als Multienzymkomplexe eine nichtribosomale Peptidsynthese ermöglichen (NRPS).[1][2]
Transkription
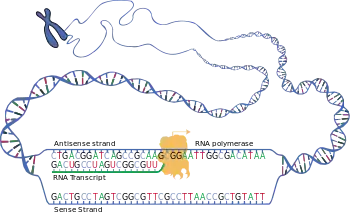
Im ersten Schritt für eine Proteinbiosynthese in einer Zelle werden Abschnitte von Genen auf der doppelsträngigen DNA aufgesucht, abgelesen und in einzelsträngige RNA-Moleküle umgeschrieben. Bei diesem Vorgang werden der vorliegenden Folge von Nukleinbasen der DNA (Adenin, Guanin, Cytosin, Thymin) komplementär die Nukleinbasen von RNA-Bausteinen (Uracil, Cytosin, Guanin, Adenin) zugeordnet. In dem dann zum Strang verknüpften RNA-Transkript kommt Ribose anstelle der Desoxyribose und Uracil anstatt Thymin vor. Die genetische Information ist in der Basenfolge enthalten, ein Codogen auf der DNA wird transkribiert zu einem Codon auf der Boten- oder Messenger-Ribonukleinsäure, kurz mRNA genannt.
Für die Transkription eines Gens ist neben mehreren anderen Faktoren eine RNA-Polymerase nötig als Enzym, das den fortlaufenden Aufbau des RNA-Polymers abhängig von der DNA-Vorlage katalysiert. Die der Vorlage basenpaarend zugeordneten Ribonukleosidtriphosphate (UTP, CTP, GTP und ATP) als Bausteine werden – jeweils unter Abspaltung zweier Phosphatgruppen der Triphosphate – miteinander zum Polynukleotid einer RNA verknüpft. Dabei kann es unterschiedliche Typen der RNA-Polymerase geben für die Transkription von Genen, die mittels einer mRNA für Proteine codieren, und für die anderer Gene, beispielsweise für die Bildung einer rRNA oder einer tRNA.
Bei Eukaryoten findet die Transkription im Zellkern statt; daher muss die mRNA aus dem Kern in das Cytosol exportiert werden, da dort die Translation durchgeführt wird. Prokaryoten hingegen haben kein Kernkompartiment, die Transkription findet hier neben der Translation im Zellplasma statt.
Posttranskriptionale Modifikation
- Spleißen
- Bei Eukaryoten müssen anschließend an die reine Transkription noch die in der dabei entstandenen prä-mRNA enthaltenen nicht-codierenden Introns herausgeschnitten werden, sodass nur noch die benötigten Exons übrig bleiben. Diesen Vorgang nennt man Spleißen. Zum Erkennen der Introns dienen Consensussequenzen. Beim Spleißen binden unterschiedliche snRNPs im Bereich der Introns und Exon-Intron-Übergänge. Diese führen unter Bildung des Spleißosoms zur Spaltung der Phosphodiesterbindungen und damit dem Herausschneiden der Introns. Gleichzeitig werden die Exons ligiert. Spleißen kommt auch bei rRNA und tRNA vor.
- Capping
- Währenddessen findet außerdem das sogenannte Capping statt, bei dem die Stabilität der RNA erhöht wird. Dabei wird eine sogenannte 5'-Cap-Struktur angehängt, wobei das 5' Ende der sich in Synthese befindlichen prä-mRNA zu einer Struktur umgewandelt wird, die als „Cap“ bezeichnet wird und die mRNA vor der Verdauung durch 5'-Exonucleasen und Phosphatasen schützt.
- Polyadenylierung
- Bei der Polyadenylierung werden die Poly(A)-Schwänze an das neu entstandene 3'-Ende der RNA (bis zu 250 Nukleotiden lang) angehängt. Dieser Poly(A)-Schwanz erleichtert den Export der mRNA in das Cytoplasma und schützt außerdem das 3'-Ende vor einem enzymatischen Abbau.
- RNA-Edition
- Bei der RNA-Edition werden einzelne oder mehrere Nukleinbasen des RNA-Moleküls nach der Transkription verändert (modifiziert), eingefügt (insertiert) oder ausgeschnitten (deletiert). Beispielsweise kann das Editing so auf der mRNA ein neues Stopcodon ergeben, das stromaufwärts des vormaligen liegt; die Translation bricht dann hier ab und es wird die kürzere Isoform eines Proteins gebildet. RNA-Editieren kommt nur bei einigen Organismen, Zellen oder Zellorganellen vor und ist oft auf besondere Nukleotidsequenzen beschränkt.
Translation
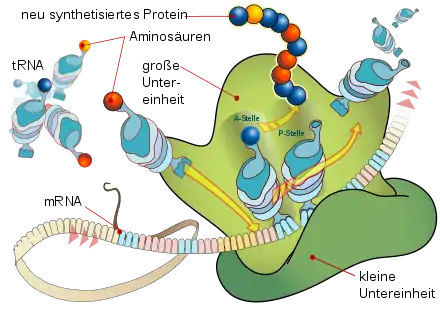
Unter Translation versteht man die Übersetzung der Basensequenz der mRNA in die Aminosäuresequenz des Proteins, die an den Ribosomen geschieht. In der mRNA bilden jeweils drei aufeinander folgende Basen, ein Basentriplett, innerhalb des offenen Leserahmens ein Codon, welches für eine Aminosäure codiert (siehe hierzu genetischer Code). Am Ribosom werden die Codons entsprechend ihrer Abfolge in Aminosäuren translatiert und diese sequentiell zu einem Polypeptid verknüpft.
Zur Ausbildung einer Peptidbindung zwischen zwei Aminosäuren müssen sie in räumliche Nähe zueinander gebracht werden. Dazu wird die Oberfläche einer großen supramolekularen Struktur benötigt. Diese Aufgabe erfüllen die Ribosomen, zusammengesetzt aus einer kleinen und einer großen Untereinheit, welche zwei nebeneinanderliegende Bindungsstellen formt: die A-Stelle und die P-Stelle.
Da es keine strukturelle Verwandtschaft zwischen einem Codon und der dazugehörigen Aminosäure gibt, wird ein Zwischenstück benötigt, das einerseits die Aminosäure bindet und andererseits das zugehörige Codon auf der mRNA erkennt. Diese vermittelnde Funktion übernehmen Transfer-Ribonukleinsäure-Moleküle, verschiedene tRNAs, als Aminosäuren-„Transporter“ mit Erkennungsregion. Sie besitzen zwei auseinanderliegende exponierte Bindungsstellen: die Aminosäurebindungsstelle und das Anticodon. Die Aminosäurebindungsstellen der tRNAs werden durch die Aminoacyl-tRNA-Synthetasen spezifisch mit der passenden Aminosäure beladen. Die tRNA erkennt mit dem Anticodon das komplementäre Codon auf der mRNA und bindet sich spezifisch daran.
Der Translationsprozess als solcher lässt sich in drei Phasen unterteilen: die Initialphase, Elongationsphase und schließlich die Termination:
- Initialphase
- Erreicht eine zuvor synthetisierte mRNA ein Ribosom, so wandert die kleine Untereinheit des Ribosoms solange an der mRNA entlang, bis sie auf das Startcodon AUG stößt. Die dazu passende Methionin-tRNA mit dem Anticodon UAC heftet sich an das Codon (Initiationskomplex).
- Elongationsphase
- Unter Spaltung von GTP lagert sich nun auch die große Untereinheit des Ribosoms an und die Elongation beginnt.
- Die Methionin-tRNA befindet sich bei der Initiationsphase auf der P-Bindungsstelle, sodass sich in der A-Bindungsstelle die nächste tRNA anlagern kann. Eine Peptidyltransferase verknüpft das Methionin der ersten tRNA mit der Aminosäure der nachfolgenden tRNA; diese Bildung eines Dipeptids findet in der A-Bindungsstelle statt. Schließlich wandern die Ribosomeneinheiten um ein Basentriplett weiter.
- Die tRNA mit dem Dipeptid befindet sich nun auf der P-Bindungsstelle, von welcher es die allererste, nun unbeladene tRNA verdrängt hat, und an die freie A-Bindungsstelle kann sich wieder die nächste tRNA anlagern, deren Anticodon komplementär zum Basentriplett des mRNA-Stranges passt.
- Termination
- Trifft ein sich an der mRNA entlang bewegendes Ribosom auf eines der drei Stoppcodons, kommt es zunächst zum Stillstand der Translation, da keine passenden tRNA-Moleküle vorhanden sind, welche für eine Aminosäure codiert sind (Suppression).[3] An ihre Stelle treten so genannte Terminations- oder Release-Faktoren (RFs), die an die A-Stelle binden und die Substratspezifität der Peptidyl-Transferase dahingehend verändern, dass ein Wassermolekül anstelle einer AA-tRNA aktiviert wird.[3] Durch dessen nucleophilen Angriff auf die Bindung zwischen Peptidkette und tRNA kommt es schließlich zur Freisetzung des synthetisierten Proteins und zur Trennung der mRNA vom Ribosom.[3]
Co- und Posttranslationale Modifikation
Die Polypeptidketten einiger Proteine werden schon während der Translation (cotranslational) durch spezielle Enzyme verändert, in den meisten Fällen aber werden Proteine erst nach Abschluss der Translation (posttranslational) modifiziert. Während Chaperone den formgebenden Prozess der Proteinfaltung beeinflussen, von dem auch die Assoziation zu Proteinkomplexen abhängt, verfügt eine Zelle daneben über eine Vielzahl an Möglichkeiten, die Struktur eines Proteins spezifisch abzuwandeln, derart auch funktionell andere Proteinspezies zu schaffen und so durch Modifikationen das Proteom zu erweitern.
Zu diesen Modifikationen gehören die Abspaltung von einzelnen endständigen Aminosäuren oder auch die längerer Peptidsequenzen bei Präkursor-Proteinen, die Einführung zusätzlicher Bindungen, z. B. Disulfidbrücken zwischen Cysteinresten, oder funktioneller Gruppen, wie Hydroxylierungen von Aminosäuren (Prolin zu 4-Hydroxyprolin durch die Prolyl-4-Hydroxylase, Lysin zu Hydroxylysin durch die Lysylhydroxylase), sowie Oxidationen (z. B. kovalente Quervernetzungen mittels Lysinresten durch die Lysyloxidase), Carboxylierungen oder Decarboxylierungen und zahlreiche weitere. Beispielsweise entstehen durch Glykosylierungen Glykoproteine, durch Acylierungen und Prenylierungen Lipoproteine.
Die einzelnen Schritte von Modifizierungen werden jeweils durch besondere Enzyme katalysiert, deren Vorkommen oft auf bestimmte Organellen, Zellen oder Gewebe beschränkt ist. Außerdem kann die Abfolge modifizierender Schritte bzw. deren zeitlicher Verlauf variiert werden, abhängig von Zellmilieu, Entwicklungsphase oder Umgebungsbedingungen. Das Kollagenmolekül etwa durchläuft eine Reihe posttranslationaler Modifikationen, von denen einige erst im Extrazellularraum stattfinden.
Proteintargeting und Proteintransport
Da viele Proteine als Zielort (englisch target) nicht das Zytosol, sondern den Extrazellularraum, die Zellmembran, die Organellen wie Chloroplasten, Mitochondrien, Peroxisomen, Zellkern oder Endoplasmatisches Retikulum haben, hat die Zelle verschiedene Mechanismen, die Proteine dorthin zu verbringen. Diese Proteine enthalten meist eine N- oder auch C-terminale Signalsequenz, die je nach Targetmechanismus sehr unterschiedlich aufgebaut sein kann. In einigen Fällen gibt es keine terminale Signalsequenz, sondern interne Signale der Peptidkette, die über den Zielort des Proteins bestimmt.
- Proteine, deren Ziel das Endoplasmatische Retikulum (ER) ist, tragen eine spezifische N-terminale Sequenz, die von einem Protein-RNA-Komplex, dem Signal Recognition Particle (SRP), erkannt wird. Der SRP-Peptid-Ribosom-Komplex wird dann zum Endoplasmatischen Retikulum rekrutiert, wo er erkannt und gebunden wird. Die Translation wird durch die Membran fortgesetzt. Durch die anheftenden Ribosomen entsteht der Eindruck eines „rauen ERs“. Siehe Cotranslationaler Proteintransport. Im Endoplasmatischen Retikulum findet die Qualitätskontrolle des neu synthetisierten Proteins statt.
- Proteine, die in die Chloroplasten verbracht werden müssen, besitzen eine N-terminale Signalsequenz, die gewöhnlich früh phosphoryliert wird. Die Proteine Hsp70, 14-3-3 und Toc64 können weiterhin durch Interaktion mit dem Protein-Vorläufer eine Rolle bei der Erkennung und Weiterleitung spielen. Der Protein-Precursor-Komplex wird nach der Ankunft auf der Oberfläche des Chloroplasten von Rezeptorstrukturen des Translokonapparates der äußeren Chloroplastenmebran (Translocon Of Outer Chloroplast Membrane, TOC) erkannt. Unter GTP-Hydrolyse wird das Protein dann in den Intermembranraum importiert oder direkt durch den Translokonapparat (TIC) der inneren Chloroplastenmembran in das Stroma importiert. Für den Import in die Membran oder das Lumen der Thylakoide werden mindestens 4 Wege genutzt, die als Sec-abhängig, SRP-abhängig, delta-pH/Tat-abhängig oder spontan bezeichnet werden.
- Für das Mitochondrium wurden für Hefe- und Tierzellen bislang drei verschiedene Import-Wege beschrieben:
- Der Präsequenz-Importweg, dessen Proteine eine N-terminale amphiphile alpha-Helix tragen. Diese Proteine sind meist für die Matrix, die innere Membran oder den Intermembranraum bestimmt.
- Der Carrier-Protein-Importweg für Proteine der inneren Membran, welche verschiedene interne Signale tragen.
- Der Importweg der Proteine der äußeren Hüllmembran, der zur Integration von Proteinen mit beta-Fass-Motiv genutzt wird. Auch hier liegen sequenzinterne Signale vor.
- Alle drei Importwege beginnen am mitochondrialen Translokonapparat in der äußeren Membran (TOM), welcher verschiedene Rezeptoren besitzt. So erkennen die Rezeptoren Tom20 und Tom22 das N-terminale Signal und leiten das Vorläufer-Protein an die Pore Tom40 weiter. Der Rezeptor Tom70 erkennt die internen Signale der Proteine, die für die äußere Membran bestimmt sind.
- Nach dem Import in den Intermembranraum trennen sich die Wege: Die Proteine mit beta-Fass-Motiv, welche für die äußere Membran bestimmt sind, werden durch den SAM-Komplex (Sorting and assembly machinery) in die Membran integriert. Die Proteine der anderen beiden Importwege werden zu verschiedenen TIM-Komplexen dirigiert: Proteine mit Präsequenz werden von dem TIM23-Komplex erkannt, Proteine für die innere Membran dagegen vom TIM22-Komplex.
- Die Präsequenz wird durch das Enzym MPP (englisch mitochondrial processing peptidase) entfernt.
Neben den oben beschriebenen Signalsequenzen ermöglicht eine Glykosylierung ein Targeting für den Einbau in die Zellmembran bzw. für die Exozytose. Beide Wege führen meist über Golgi-Vesikel.
Weblinks
Einzelnachweise
- Matthias Strieker, Alan Tanović, Mohamed A. Marahiel: Nonribosomal peptide synthetases: structures and dynamics. In: Current opinion in structural biology. Band 20, Nummer 2, April 2010, S. 234–240, doi:10.1016/j.sbi.2010.01.009, PMID 20153164.
- Gavin J. Williams: Engineering polyketide synthases and nonribosomal peptide synthetases. In: Current opinion in structural biology. Band 23, Nummer 4, August 2013, S. 603–612, doi:10.1016/j.sbi.2013.06.012, PMID 23838175.
- Translation. (spektrum.de [abgerufen am 15. Juni 2018]).