CRISPR/Cas-Methode
Die CRISPR/Cas-Methode (von englisch Clustered Regularly Interspaced Short Palindromic Repeats - gruppierte kurze palindromische Wiederholungen mit regelmäßigen Abständen und CRISPR-associated - CRISPR-assoziiertes Protein) ist eine molekularbiologische Methode, um DNA gezielt zu schneiden und zu verändern (Genome Editing). Gene können mit dem CRISPR/Cas-System eingefügt, entfernt oder ausgeschaltet werden,[1] auch Nukleotide in einem Gen können geändert werden.[2] Aufgrund der einfachen Durchführung, der Skalierbarkeit hinsichtlich unterschiedlicher Zielsequenzen und der geringen Kosten wird die CRISPR/Cas-Methode zunehmend in der Forschung eingesetzt.[3][4] Gleichzeitig gibt es beim aktuellen Stand der Forschung noch Probleme bei der Spezifität durch off-target-Effekte, also Auswirkungen am Genom außerhalb der Schnittstelle.
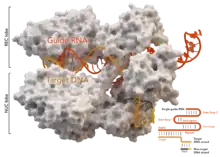
Die wissenschaftliche Grundlage zur Entwicklung der CRISPR/Cas-Methode wurde durch die Entdeckung und Erforschung der CRISPR-Sequenzen und des damit verbundenen CRISPR/Cas-Systems im Immunsystem verschiedener Bakterien und Archaea gelegt. Die erste wissenschaftliche Dokumentation zur Entwicklung und zum Einsatz der Methode wurde 2012 durch eine Arbeitsgruppe um Emmanuelle Charpentier und Jennifer Doudna veröffentlicht. Die wissenschaftliche Fachzeitschrift Science erklärte die CRISPR-Methode zum Breakthrough of the Year 2015.[5] Für ihre Arbeiten an der CRISPR/Cas-Methode wurden die beiden Wissenschaftlerinnen mit dem Nobelpreis für Chemie 2020 ausgezeichnet.
Prinzip
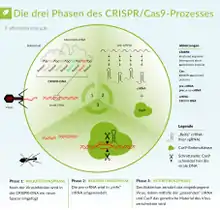
Die CRISPR/Cas-Methode basiert auf einem adaptiven antiviralen Abwehrmechanismus von Bakterien, dem CRISPR.[7][8] Sie wird als Methode verwendet, um DNA an einer bestimmbaren DNA-Sequenz zu schneiden. Dadurch können beispielsweise durch zwei Schnitte DNA-Sequenzen entfernt – oder es kann im Anschluss an einen Schnitt eine andere DNA-Sequenz an der Schnittstelle eingefügt werden.
Das DNA-schneidende Enzym namens Cas (von englisch CRISPR-associated‚ CRISPR-assoziiert‘) bindet eine bestimmte RNA-Sequenz. Auf diese RNA-Sequenz folgt eine weitere RNA-Sequenz, die per Basenpaarung an eine DNA mit komplementärer Sequenz binden kann. Die RNA dient hierbei als Brücke zwischen Cas und der zu schneidenden DNA. Durch die Verkettung des Enzyms Cas, der RNA und der DNA wird das DNA-schneidende Enzym Cas in die räumliche Nähe der gebundenen DNA gebracht, woraufhin das Enzym die (indirekt gebundene) DNA schneidet. Im Sonderfall einer Einfügung von DNA in die Schnittstelle wird eine weitere DNA hinzugegeben, die an ihren beiden Enden jeweils überlappende Sequenzen für eines der beiden Enden der Schnittstelle aufweist. Die einzufügende DNA wird durch die zelleigene DNA-Reparatur mit den Enden der Schnittstelle verbunden. Bei einer Variante des Systems (Prime Editing) ist keine einzufügende DNA für eine Einfügung nötig, da die einzufügende Sequenz durch eine RNA-Verlängerung codiert wird.
Eigenschaften
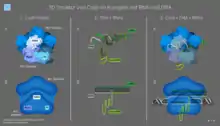
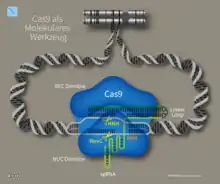
Cas-Proteine können als Ribonukleoproteine bestimmte RNA-Sequenzen binden. Die Endonuklease Cas9 (veraltet auch Cas5, Csn1 oder Csx12) kann eine bestimmte RNA-Sequenz (crRNA repeat, Sequenz GUUUUAGAGCU(A/G)UG(C/U)UGUUUUG)[9] binden und in der unmittelbaren Umgebung DNA schneiden. In Bakterien folgt auf die crRNA repeat-Sequenz eine crRNA-spacer-Sequenz, die an die Ziel-DNA bindet. Beide Teile werden zusammen als crRNA bezeichnet. Die crRNA-spacer-Sequenz dient in der Funktion eines variablen Adapters, die komplementär zur Ziel-DNA ist und an die Ziel-DNA bindet. Die crRNA-repeat-Sequenz bildet eine RNA-Sekundärstruktur und wird dann von Cas9 gebunden,[10] wodurch eine Änderung der Proteinfaltung von Cas9 erfolgt und die Ziel-DNA von der crRNA-spacer-Sequenz durch Basenpaarung gebunden wird.[11] Bei der CRISPR/Cas-Methode wird die crRNA-spacer-Sequenz geändert, um an eine andere Ziel-DNA zu binden. Die Sequenz des crRNA-spacer bestimmt, welche Ziel-DNA-Sequenz gebunden wird.
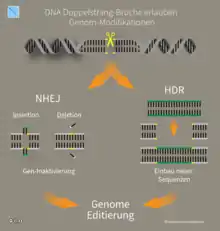
Neben Cas9 und der crRNA ist das Vorhandensein eines PAM-Motivs (englisch protospacer adjacent motif ‚Angrenzendes Motiv an den Protospacer‘) mit der Sequenz NGG (mit N als beliebige Nukleinbase gefolgt von zwei Guaninen) in der Ziel-DNA notwendig für eine Aktivierung von Cas9.[12] Der Schnitt der DNA erfolgt drei Nukleotide vor dem PAM. Zudem ist noch eine weitere RNA notwendig, die tracrRNA, von engl. trans-acting CRISPR RNA. Die tracrRNA ist teilweise komplementär zur crRNA, weshalb sie aneinander binden. Die tracrRNA bindet an eine Vorläufer-crRNA, bildet eine RNA-Doppelhelix und wird durch die RNase III in die aktive Form überführt.[13][1][14] Die beiden RNA-Stränge der crRNA und der tracrRNA können auch in einem einzelnen, teilweise selbsthybridisierenden RNA-Strang untergebracht werden (sgRNA ‚single guide RNA‘).[1] Durch diese Komponenten wird die DNA gebunden und von der Endonukleasefunktion nahe der Bindungsstelle an beiden Strängen der Ziel-DNA geschnitten. In lebenden Organismen folgt nach einem doppelsträngigen DNA-Schnitt eine DNA-Reparatur. Die Reparatur des erzeugten Doppelstrangbruchs erfolgt durch homology-directed repair (HDR) oder durch non-homologous end joining (NHEJ).
Eine alternative Methode mit gleicher Spannbreite möglicher DNA-Zielsequenzen ist das CRISPR/Cpf1-System und das CRISPR/Cas12b-System. Alternativ zum CRISPR/Cas-System können teilweise Transcription Activator-like Effector Nucleases (TALEN) und Zinkfingernukleasen (ZFN) verwendet werden. Jedoch sind diese beiden Methoden mit höherem Aufwand zur Anpassung an unterschiedliche Zielsequenzen verbunden, da zuerst ein Proteindesign zur Änderung der Bindungsspezifität notwendig ist, während die Bindungsspezifität bei Cas-Proteinen je nach Verwendung durch die gebundene crRNA bzw. sgRNA erfolgt und somit leichter zu ändern ist. Alle diese Methoden werden umgangssprachlich als Genschere bezeichnet.
Komponenten
| Komponente | Funktion |
|---|---|
| crRNA | Enthält die Guide RNA, die das richtige Segment der Wirts-DNA lokalisiert, zusammen mit einer Region, die an die tracrRNA bindet (im Allgemeinen in Form einer Haarnadelschleife) und einen aktiven Komplex bildet. |
| tracrRNA | Bindet an crRNA und bildet einen aktiven Komplex. |
| sgRNA | Single-guide RNAs sind kombinierte RNAs, die aus einer tracrRNA und mindestens einer crRNA bestehen. |
| Cas9 (oder Alternative) | Ein Enzym, dessen aktive Form die DNA verändern kann. Es gibt viele Varianten mit unterschiedlichen Funktionen (z. B. Einzelstrangeinkerbung, Doppelstrangbruch, DNA-Bindung) aufgrund der Funktion jedes Enzyms zur Erkennung von DNA-Stellen. |
| Reparaturvorlage | DNA-Molekül, das im DNA-Reparaturprozess der Wirtszelle als Vorlage dient und das Einfügen einer spezifischen DNA-Sequenz in das von Cas9 geteilte Wirtssegment ermöglicht. |
Alternativen zu Cas9
| Protein | Hauptanwendung / Charakteristiken | Jahr/e |
|---|---|---|
| Cas12 | Cas12a ist kleiner und einfacher als Cas9; Cas12b u. a. für das Genome Engineering bei Pflanzen.[15][16] | |
| Cas13 | für RNA-Editing[17] | |
| Cas3[18][19] | 2019 | |
| CasMINI | Etwa doppelt so kompakt wie die gebräuchlicheren Cas9 und Cas12a.[20][21] | 2021 |
Unterschiede zwischen Cas9, Cpf1 und Cas12b
| Eigenschaft | Cas9[22] | Cpf1[22] | Cas12b[23] |
|---|---|---|---|
| Struktur | 2 RNA notwendig oder 1 sgRNA | 1 RNA notwendig (keine tracrRNA) | 2 RNA notwendig oder 1 sgRNA |
| Überhang | keinen (blunt end) | sticky end | sticky end |
| Schnittstelle | proximal zur Erkennungssequenz | distal zur Erkennungssequenz | distal zur Erkennungssequenz |
| Zielsequenz | G-reiches PAM | T-reiches PAM | T-reiches PAM |
| Zelltyp | teilende Zellen | ruhende Zellen | unbekannt |
Das CRISPR/Cas-System kann durch Zugabe eines DNA-Oligonukleotids anstatt eines RNA-Oligonukleotids auch zur Veränderung von RNA verwendet werden.[24][25] Der Effekt der Unterdrückung eines Gens durch Verwendung von CRISPR/Cas besitzt verschiedene Ähnlichkeiten mit demjenigen der RNA-Interferenz, da bei beiden bakteriellen Abwehrmechanismen kurze RNA-Stücke von etwa 18 bis 20 Nukleotiden die Bindung an das Ziel vermitteln.[26]
Anpassung an die Zielsequenz
Wird an eine crRNA repeat-Sequenz anstatt der natürlich vorkommenden crRNA-spacer-Sequenz eine andere, zu einer DNA-Zielsequenz komplementäre RNA-Sequenz angefügt und diese crRNA zu einer tracrRNA hinzugegeben, schneidet Cas9 die DNA nahe der geänderten Zielsequenz. Die an die Ziel-DNA bindende Sequenz besteht aus 20 Nukleotiden, von denen vor allem die 12 an das PAM angrenzenden Nukleotide für die Bindungsspezifität entscheidend sind.[27] Durch das Cas9 mit den entsprechend geänderten RNA-Sequenzen kann sequenzspezifisch doppelsträngige, teilweise komplementäre DNA geschnitten werden, wodurch gezielte Deletionen erzeugt werden können. Die gleichzeitige Änderung mehrerer DNA-Zielsequenzen wird als Multiplex Genome Editing bezeichnet.
Nukleotidsubstitution
Einzelne Cytidine können durch ein Fusionsprotein aus Cas9 ohne doppelsträngige Endonuklease-Aktivität mit einer Cytidin-Deaminase in Thymidin umgewandelt werden.[28] Entsprechend kann mit einem Fusionsprotein, das die Adenosin-Deaminase enthält, ein Adenosin in Guanosin verändert werden.[29] In beiden Fällen entstehen Substitutionsmutationen. Da bei diesen Methode nur einzelne Basen verändert werden, ohne dass die DNA geschnitten wird, wird dieses Verfahren als Base Editing bezeichnet.
Insertionen
Durch Doppelstrangbrüche kann die Häufigkeit einer homologen Rekombination deutlich erhöht werden,[30] wodurch ein sequenzspezifischer Schnitt durch Cas9 auch zum Einfügen von DNA-Sequenzen (z. B. im Zuge der Gentherapie) verwendet werden kann, sofern die anschließend einzufügende DNA an beiden Enden jeweils eine zur jeweiligen Zielsequenz überlappende Sequenz aufweist.[31]
Einbringung in Zellen
Durch Transformation oder Transfektion von einem Vektor können Lebewesen mit dem CRISPR/Cas-System "ergänzt" werden, die es natürlicherweise nicht besitzen, z. B. manche Bakterienstämme,[32] Bäckerhefe,[33] Taufliegen,[34] Zebrabärblinge,[35] Mäuse[36] und Menschen.[37][38] Üblicherweise werden Gene von Cas mit Kernlokalisierungssignal und sgRNA per Plasmid in einen Organismus eingefügt, alternativ kann auch der Komplex aus Cas9 mit einer sgRNA in Zellen eingeschleust werden.[39]
Für ein Genome Editing in der Keimbahn werden als Methoden zur Einschleusung des CRISPR/Cas9 die Elektroporation und die Mikroinjektion eingesetzt.
Spezifität
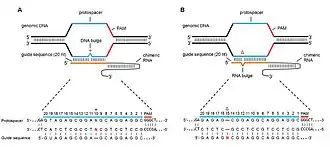
Bei der Erzeugung von Doppelstrangbrüchen an unerwünschten Stellen können unerwünschte Mutationen entstehen, die als off-target-Effekte bezeichnet werden.[40][41][42][43] Durch einen unspezifischen Schnitt können Gene in ihrer Funktion gestört werden. Unter die off-target-Effekte fallen Punktmutationen (Basenänderungen),[44] Deletionen (Entfernungen von DNA-Sequenzen),[45][46] Insertionen (Einfügungen von DNA-Sequenzen),[43] Inversionen (Einfügungen von DNA-Sequenzen mit umgekehrter Abfolge)[43] und Translokationen (Verbindungen mit anderen DNA-Strängen).[47][46] Es wurde beschrieben, dass teilweise über 50 % der Schnitte off-target erfolgten.[40][45] Die Bindung der sgRNA an eine Zielsequenz toleriert auch Basenfehlpaarungen, wodurch sich die Zahl der möglichen Bindungsstellen auf mehrere tausend erhöht, wodurch experimentelle und sicherheitstechnische Probleme entstehen,[48][40] die typisch für unspezifische Mutationen sind. Experimentelle Probleme sind irreführende und nicht reproduzierbare Ergebnisse.[49] Als sicherheitstechnisches Problem wird eine mögliche Entstehung von Krebs angesehen.[50] Die Ursachen für unspezifische Schnitte lassen sich in Basenfehlpaarungen und Schleifenbildungen (der sgRNA oder der Ziel-DNA) einteilen.[45]
Im Jahr 2015 hat ein Team chinesischer Forscher 86 menschlichen Embryonen die korrekte Version des Beta-Globin-Gens injiziert, um den der Beta-Thalassämie zugrunde liegenden Defekt zu korrigieren; dabei wurde festgestellt, dass das korrekte Gen an der richtigen Stelle nur in vier Fällen nachgewiesen werden konnte, in den vier Stellen lag aber ein Mosaik vor.[51]
Erhöhung der Spezifität
Zur Minimierung unspezifischer DNA-Schnitte und der daraus potentiell folgenden Mutagenese werden verschiedene Ansätze zur Erhöhung der Spezifität untersucht,[52][53] wie Proteindesign der Cas9 oder Ersatz der Endonukleaseaktivität von Cas9 durch Kombination mit anderen Endonukleasen.[54] Entsprechend wurden verschiedene Methoden zur Detektion solcher Mutationen entwickelt.[55][56][47][57][58] Ebenso wurden Computerprogramme und Datenbanken entwickelt, um off-target-Effekte vorherzusagen.[59][60]
Paired Nickases
Die Mutation von Asparaginsäure zu Alanin an der Position 10 in Cas9 (kurz: D10A) oder von Histidin zu Alanin an der Position 840 (H840A) inaktiviert die doppelsträngige Endonukleasefunktion des gebundenen DNA-Stranges unter Erhalt der RNA-DNA-Bindungsfunktion und einer einzelsträngigen Endonukleasefunktion.[61][62] Zudem wird die Spezifität durch die Affinität der RNA-DNA-Bindung an Cas9 bestimmt.[63][64][65] Durch Verwendung zweier verschiedener sgRNA, deren an die Ziel-DNA bindende Sequenzen geringfügig versetzt sind, entstehen zwei verschieden bindende Cas-RNA-Komplexe, wodurch die Spezifität des Schnitts erhöht werden kann.[61][66] Dabei werden zwei geringfügig verschiedene Cas9-sgRNA-Komplexe mit unterschiedlicher Spezifität gebildet (paired nickases). Zudem entstehen durch die versetzten Einzelstrangbrüche sticky ends, die eine Insertion einer DNA mit komplementären sticky ends erleichtern. Einzelstrangbrüche werden durch die Basenexzisionsreparatur[61] und die HDR geschlossen,[67] die weniger Mutationen als die Reparatur per NHEJ erzeugen.[61]
Proteindesign von Cas9
Die Mutation D1135E (Änderung von Asparaginsäure zu Glutaminsäure an der Position 1135) von SpCas9 verändert die Bindungsspezifität für das protospacer adjacent motif (PAM) und senkt die Anzahl unspezifischer Schnitte.[68] Auch die HF1-Mutante von Cas9 führt zu einer Minderung unspezifischer DNA-Schnitte.[69]
dCas9-FokI
Die Kombination von Cas9 mit inaktivierter Nukleasefunktion (dCas9) mit der Endonuklease FokI wurde entwickelt, um die Spezifität des DNA-Schnitts zu erhöhen.[54][70][71] FokI wird nur als Homodimer aktiv. Dadurch wird die Anzahl unspezifischer Schnitte auf 1/10.000 gemindert.
BhCas12b v4
Das Protein Cas12b (alter Name C2c1, ein Cas des Typs V, Klasse II) aus Bacillus hisashii, abgekürzt BhCas12b, ist mit 1108 Aminosäuren kleiner als SpCas9 (1368 Aminosäuren) und hat daher auch ein kleineres Gen, was die Verwendung in viralen Vektoren (insbesondere AAV-Vektoren) erleichtert.[72] Allerdings erzeugt der Wildtyp von BhCas12b bei 37 °C nur Einzelstrangbrüche.[72] Daher wurde eine Mutante von BhCas12b namens BhCas12b v4 entwickelt (K846R/S893R/E837G), die auch bei der Körpertemperatur von Säugetieren Doppelstrangbrüche und weniger unspezifische Schnitte erzeugt.[72]
dCas9-RT: Prime Editing
Eine deaktivierte Cas9 (dCas9) – die noch über eine RNA an DNA binden kann, aber ohne DNA zu schneiden – wurde als Fusionsprotein mit einer reversen Transkriptase (RT) kombiniert.[73] Reverse Transkriptasen verwenden eine RNA-Vorlage, um DNA herzustellen – sie sind RNA-abhängige DNA-Polymerasen. Zusätzlich wird eine prime editing guide RNA (pegRNA) benötigt, die sowohl die crRNA-repeat-Sequenz für die Bindung von Cas9 und eine an die Ziel-DNA bindende RNA-Sequenz als auch eine RNA-Vorlagensequenz zur Änderung der DNA-Sequenz (analog zu einem Primer) enthält.[73] Dadurch können Insertionen, Deletionen und alle 12 möglichen Punktmutationen durchgeführt werden, ohne dass mutationsanfällige Doppelstrangbrüche entstehen und im Falle einer Insertion ohne Notwendigkeit einer einzufügenden DNA-Sequenz.[73]
Regulation
Die Verwendung von Anti-CRISPR-Proteinen wurde zur konditionalen Hemmung von CRISPR/Cas sowie zur zeitlichen, örtlichen (zelltypspezifischen) oder zellzyklusabschnittsspezifischen Steuerung der CRISPR-Cas-Methode vorgeschlagen.[74] Da CRISPR/Cas DNA schneidet, solange es aktiv ist, kann eine zeitliche Begrenzung auch unspezifische Schnitte begrenzen, die zu unerwünschten Mutationen führen können.[74]
Beispielsweise werden Anti-CRISPR-Proteine zu einem gewünschten Zeitpunkt induziert[75] oder es werden Fusionsproteine von Anti-CRISPR-Proteinen mit lichtgesteuerten Proteinen zur zeitlichen Steuerung der Methode verwendet.[76] Die Kombination lichtsensitiver Proteine mit Anti-CRISPR-Proteinen ermöglicht eine Aktivierung von CRISPR-Cas nur während einer Bestrahlung mit Licht, beispielsweise die Kombination des Anti-CRISPR-Proteins AcrIIA4 (ein Hemmstoff von Cas9) mit dem lichtsensitiven Protein LOV2 aus Avena sativa (Saathafer), die auch CASANOVA genannt wird.[76] Durch die Verwendung zelltypspezifischer Promotoren vor den Genen von Cas und sgRNA kann die örtliche Wirkung gesteuert werden. Außerdem wurde durch die Kombination der Gene von Anti-CRISPR-Proteinen (AcrIIC1, AcrIIC3 und AcrIIC1) mit Bindungsstellen für miRNAs, die nur in bestimmten Geweben (Leberzellen und Herzmuskelzellen) vorkommen und dort die Bildung des Anti-CRISPR-Proteins hemmen, eine gewebespezifische Steuerung für Leber- und Herzmuskelzellen ermöglicht.[77] Durch die Verwendung zellzyklusabschnittsspezifischer Promotoren (Promotoren von Cyclinen) kann eine Wirkung auf einen Abschnitt im Zellzyklus begrenzt werden.
Typen
Es existieren mehr als 40 verschiedene Cas-Proteinfamilien.[78] Die Familien können in zwei Klassen mit sechs Typen (Klasse I mit Typen I, III und IV sowie Klasse II mit Typen II, V und VI) und weiter in mehr als 30 Subtypen[79] eingeteilt werden.[80][81] Bei Klasse I besteht der Proteinanteil aus mehreren Proteinen in einem Proteinkomplex (Effektorkomplex), während Klasse II nur ein Protein (Effektorprotein) verwendet.[80] Typ I und II binden und schneiden doppelsträngige DNA und III-A dsDNA sowie RNA,[82] während Typ III-B einzelsträngige RNA oder DNA[83] bindet und schneidet.[81][84] Bei allen Typen erfolgt die Bildung des spacers in Bakterien durch Cas1 und Cas2.[84] Bei den Typen I-A und I-E erfolgt der DNA-Schnitt durch Cas3, während bei Typ II Cas9, bei Typ III-A Csm6 und bei Typ III-B Cmr4 den Schnitt bewirkt.[84] Die Typen I und III sind strukturell verwandt, was einen gemeinsamen Ursprung nahelegt.[81] Das helikale Protein Cas des Typs III besitzt mehrere β-hairpins, die in Abständen von sechs Nukleotiden die Doppelhelix der crRNA und der Ziel-RNA für den Schnitt auseinanderdrücken.[81]
Das Cas9 stammt meistens entweder aus Streptococcus pyogenes (SpCas9) oder Staphylococcus aureus (SaCas9), wobei die kodierende DNA-Sequenz von SaCas9 etwa 1000 Basenpaare kürzer ist.[85] Cas9 gehört zum Typ II und wird vermehrt eingesetzt, da der Proteinanteil nur aus einem Protein besteht, wodurch die Klonierung und Überexpression weniger aufwändig ist. Cas-Proteine des Typs I sind dagegen Proteinkomplexe aus mehreren kleinen Proteinen.[86]
Klassifizierung
Aufgrund der evolutionären Entwicklung von Pathogenen und der daraus resultierenden Vielfalt an Erregern müssen sich antivirale Abwehrmechanismen den verschiedenen Pathogenen anpassen können. Dieses Phänomen beschreibt man in der englischsprachigen Literatur als evolutionary arms race (deutsch evolutionäres Wettrüsten).[87][88] Aufgrund dessen haben CRISPR/Cas-Systeme als wichtige Akteure der antiviralen Abwehrmechanismen sich ebenfalls den evolutionären Änderungen angepasst. Dies resultierte in einer Erhöhung der Diversität hinsichtlich der Genzusammensetzung des cas-Operons, der Architektur des CRISPR-Genlocus und der Gensequenzen (auch innerhalb der Cas-Core-Gene). Bei den Cas-Core-Genen handelt es sich um die cas-Gene cas1 bis cas6, die innerhalb vieler CRISPR/Cas-Systemvarianten konserviert sind.[78]
Eine einfache und rationale Klassifizierung von CRISPR/Cas-Systemen erweist sich aus folgenden Gründen als vorteilhaft:
- Erklärung der Herkunft und Evolution der Diversität von CRISPR/Cas-Systemen
- Mitverfolgung und Integration neu entdeckter CRISPR/Cas-Systemvarianten
- kohärente Annotation von CRISPR/Cas-Genloci in mikrobiellen Genomen
Die seit den Anfängen der CRISPR-Forschung verwendete Klassifizierung von CRISPR/Cas-Systemen anhand der Phylogenese des Cas1-Proteins[89] erwies sich nach weiteren Entdeckungen von Genomen mit CRISPR/Cas-Genloci als problematisch, weil die Organisation und Phylogenese der Gene des Effektormoduls von der Phylogenese des Cas1-Proteins abwich. Unter einem Effektormodul versteht man eine Gruppe von cas-Genen, die zur Identifizierung von genetischem Material dient. Des Weiteren existiert ein Adaptationsmodul, welches ebenfalls cas-Gene umfasst und mithilfe von Effektorproteinen zur Protospacer-Auswahl beiträgt, die in das bakterielle Genom integriert werden können.[90][91] Ursache für die phylogenetischen Abweichungen ist wahrscheinlich die Rekombination zwischen den Effektor- und Adaptationsmodulen (englisch module shuffling).[92][93] Somit entschied man, dass charakteristische Merkmale des Effektormoduls zum Klassifizierungsmerkmal von CRISPR/Cas-Systemen wurde.
| Klasse | Typ | Ziel | Operon-Organisation1 | Subtyp | Bakterienstamm |
|---|---|---|---|---|---|
I |
I |
DNA |
cas6, cas11, cas7, cas5, cas8a1, cas3’, cas3’’, cas2, cas4, cas1, cas4, CRISPR | I-A |
Archaeoglobus fulgidus AF1859, AF1870–AF1789 |
DNA |
cas6, cas8b1, cas7, cas5, cas3, cas4, cas1, cas2, CRISPR | I-B |
Clostridium kluyveri CKL_2758–CKL_2751 | ||
DNA |
cas3, cas5, cas8c, cas7, cas4, cas1, cas2, CRISPR | I-C |
Bacillus halodurans BH0336–BH0342 | ||
DNA |
cas3, cas8u2, cas7, cas5, cas6, cas4, cas1, cas2, CRISPR | I-G |
Geobacter sulfurreducens GSU0051–GSU0054, GSU0057–GSU0058 | ||
DNA |
cas3’, cas3’’, cas10d, cas7, cas5, cas6, cas4, cas1, cas2, CRISPR | I-D |
Cyanothece sp. 8802 Cyan8802_0527–Cyan8802_0520 | ||
DNA |
cas3, cas8e, cas11, cas7, cas5, cas6, cas1, cas2, CRISPR | I-E |
Escherichia coli K12 ygcB–ygbF | ||
DNA |
cas1, cas2, cas3, cas8f, cas5f1, cas7f1, cas6f, CRISPR | I-F1 |
Yersinia pseudotuberculosis YPK_1644–YPK_1649 | ||
| tnsA, tnsB, tnsC, tnsD, cas8f3/cas5f3, cas7f3, cas6f, CRISPR | I-F3 |
Vibrio crassostreae J5 20 VCR20J5_310088–VCR20J5_310108 | |||
DNA |
cas1, cas2, cas3, cas7f2, cas5f2, cas6f, CRISPR | I-F2 |
Shewanella putrefaciens CN–32 Sputcn32_1819–Sputcn32_1823 | ||
IV |
Plasmide [95] |
dinG, cas6, cas8-like, cas7, cas5, CRISPR | IV-A |
Thioalkalivibrio sp. K90mix TK90_2699–TK90_2703 | |
Plasmide [95] |
cysH-like, cas8-like, cas11, cas7, cas5, CRISPR | IV-B |
Rhodococcus jostii RHA1 RHA1_ro10069–RHA1_ro10072 | ||
DNA? |
LS, cas11, cas7, cas5, CRISPR | IV-C |
Thermoflexia bacterium D6793_05715–D6793_05700 | ||
III |
DNA/RNA |
cas6, cas10, cas11, cas7, cas5, cas7, csm6, cas1, cas2, CRISPR | III-A |
Staphylococcus epidermidis SERP2463–SERP2455 | |
RNA? |
cas10, cas7, cas5, cas11, cas7, cas7, csx19, cas7, CRISPR | III-D |
Synechocystis sp. 6803 sll7067–sll7063 | ||
RNA? |
TPR + caspase, cas7(3), cas11, RT, cas1, cas2, CRISPR | III-E |
Candidatus Scalindua brodae SCABRO_02601,SCABRO_02597, SCABRO_02593,SCABRO_02595 | ||
DNA? |
cas10, cas5, cas11, cas7, CRISPR | III-F |
Thermotoga lettingae TMO Tlet_0097–Tlet_0100 | ||
DNA/RNA |
cas7, cas7, cas10, cas7, cas11, cas5, CRISPR | III-C |
Methanothermobacter thermautotrophicus MTH328–MTH323 | ||
DNA/RNA |
cas7, cas10, cas5, cas7, cas11, cas6, cas7, CRISPR | III-B |
Pyrococcus furiosus PF1131–PF1124 | ||
II |
II |
DNA |
cas9, cas1, cas2, cas4, tracrRNA, CRISPR | II-B |
Legionella pneumophila str. Paris lpp0160–lpp0163 |
DNA |
cas9, cas1, cas2, csn2, tracrRNA, CRISPR | II-A |
Streptococcus thermophilus str0657–str0660 | ||
DNA |
cas9, cas1, cas2, tracrRNA, CRISPR | II-C1 |
Neisseria lactamica 020-06 NLA_17660–NLA_17680 | ||
DNA |
cas9, tracrRNA, CRISPR, cas4, cas2, cas1 | II-C2 |
Micrarchaeum acidiphilum ARMAN-1 BK997_03320–BK997_03335 | ||
V |
DNA |
cas12a, cas4, cas1, cas2, CRISPR | V-A |
Francisella cf. novicida Fx1 FNFX1_1431–FNFX_1428 | |
DNA |
cas12e, cas4, cas1, cas2, tracrRNA, CRISPR | V-E |
Deltaproteobacteria bacterium A2Z89_08250–A2Z89_08265 | ||
DNA |
cas12b1, cas4, cas1, cas2, tracrRNA, CRISPR | V-B1 |
Alicyclobacillus acidoterrestris N007_06525–N007_06535 | ||
DNA |
cas4, cas1, cas2, cas12b2, tracrRNA, CRISPR | V-B2 |
Planctomycetes bacterium RBG_13_46_10 A2167_01675–A2167_01685 | ||
DNA |
cas12i, CRISPR | V-I |
Freshwater metagenome (JGI) Ga0208225_100001036 | ||
| cas12h, CRISPR | V-H |
Hypersaline lake sediment metagenome (JGI) Ga0180438_100006283 | |||
DNA |
cas1, cas12c, CRISPR | V-C |
Oleiphilus sp. A3715_16885–A3715_16890 | ||
DNA |
cas1, CRISPR, cas12d | V-D |
Bacterium CG09_39_24 BK003_02070–BK003_02075 | ||
DNA |
cas1, cas2, cas4, cas12f1, CRISPR | V-F1 |
Uncultured archaeon NDOCEIEL_00008–NDOCEIEL_00011 | ||
DNA |
c2c10, CRISPR | V-F1 (V-U3) |
Bacillus thuringiensis HD-771 BTG_31928 | ||
DNA |
cas12f2, CRISPR, cas1, cas2, cas4 | V-F2 |
Uncultured archaeon ICDLJNLD_00049–ICDLJNLD_00052 | ||
| c2c8, CRISPR | V-U2 |
Cyanothece sp. PCC 8801 PCC8801_4127 | |||
| c2c9, CRISPR | V-U4 |
Rothia dentocariosa M567 HMPREF0734_01291 | |||
| cas1, cas2, cas4, cas12f3, CRISPR | V-F3 |
Candidatus Micrarchaeota archaeon COU37_03050–COU37_03065 | |||
| c2c4, CRISPR | V-U1 |
Gordonia otitidis GOOTI_RS19525 | |||
| cas12g, CRISPR, tracrRNA | V-G |
Hot springs metagenome FLYL01000025.1 (182949..185252) | |||
| tnsB, tnsC, tniQ, cas12k, tracrRNA, CRISPR | V-K (V-U5) |
Cyanothece sp. PCC 8801 PCC8801_2993–PCC8801_2997 | |||
VI |
RNA |
cas13a, cas1, cas2, CRISPR | VI-A |
Leptotrichia shahii B031_RS0110445 | |
RNA |
WYL, cas13d, cas1, cas2, CRISPR | VI-D |
Ruminococcus bicirculans RBI_RS12820 | ||
RNA? |
cas13c, CRISPR | VI-C |
Fusobacterium perfoetens T364_RS0105110 | ||
RNA |
cas13b1, csx28, CRISPR | VI-B1 |
Prevotella buccae HMPREF6485_RS00335–HMPREF6485_RS00340 | ||
RNA |
csx27, cas13b2, CRISPR | VI-B2 |
Bergeyella zoohelcum HMPREF9699_02005–HMPREF9699_02006 |
Anwendungen

Das CRISPR/Cas-System kann unter anderem zum Genome Editing (Deletionen/Gen-Knockout[96] und Insertionen) und damit auch zur Gentherapie verwendet werden.[97][98]
Problematisch für humane Anwendungen könnte jedoch sein, dass das Immunsystem die Endonuklease Cas9, die bakteriellen Ursprungs ist, als Antigen erkennt. Über 80 % aller gesunden Menschen zeigen sowohl eine Antikörper-basierte humorale Immunantwort als auch eine auf T-Gedächtniszellen basierte zelluläre Immunantwort. Ursache hierfür ist, dass die meisten Menschen im Laufe ihres Lebens schon einmal mit Bakterien wie S. pyogenes oder S. aureus in Kontakt kamen und sich so entsprechende Antikörper und Gedächtniszellen bilden konnten.[99] Körperzellen, deren Genom mit CRISPR/Cas9 editiert wurde, können prinzipiell von cytotoxischen T-Zellen erkannt und durch Apoptose vernichtet werden. Der therapeutische Effekt wäre somit nicht mehr gegeben. Diese Problematik wird aktuell (Stand 2018) intensiv diskutiert.[100][101][102]
Weiterhin wird das CRISPR/Cas-System zur Entfernung der Genome von Krankheitserregern chronischer Infektionskrankheiten wie des Hepatitis-B-Virus[103] und des HIV[104][105] eingesetzt. Die gezielte Veränderung einzelner Gene wird bei der Charakterisierung von Onkogenen und somit zur Untersuchung der Tumorentstehung verwendet.[106][107] Das CRISPR/Cas-System wird zur Untersuchung der Funktionen von teilweise unbekannten Genen eingesetzt.[108] Zudem wird es zur Korrektur von Mutationen bei der Erzeugung induzierter pluripotenter Stammzellen[109] und embryonaler Stammzellen verwendet.[110][111] Weitere Anwendungen werden untersucht.[112] Im März 2020 setzten Wissenschaftler erstmals CRISPR-Cas9 in einem menschlichen Körper ein. Sie versuchen in einer klinischen Studie mittels Genome Editing das Sehvermögen eines Patienten mit Lebersche Kongenitale Amaurose wiederherzustellen, nachdem Tests in menschlichen Zellen, Mäusen und Affen erfolgreich verliefen und sie eine offizielle Genehmigung erhielten. Sie injizieren dazu drei Tropfen mit Milliarden Viren unter die Retina des Patienten. Die Änderung der DNA ist permanent und – anders als beim Human Germline Engineering – nicht vererbbar.[113][114][115][116][117] Im Juni 2020 wurden vorläufige Ergebnisse der, im Frühjahr 2019 gestarteten, ersten klinischen Studie zur Behandlung von vererbten genetischen Erkrankungen mittels CRISPR-Cas9 außerhalb von China veröffentlicht. Sie deuten auf einen Erfolg der Behandlung („CTX001“) hin.[118][119] 2021 wurde die erste, kleine klinische Studie intravenöser CRISPR-Genbearbeitung im Menschen mit erfolgversprechenden Ergebnissen beendet.[120][121]
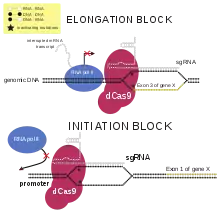
Mit dem CRISPR/Cas-System wurden unter anderem Bakteriophagen-resistente Bakterienstämme erzeugt, und dies durch eine adaptive Resistenz bei Hinzufügen entsprechender RNA bei industriell wichtigen Bakterien, z. B. in der Milch- oder Weinindustrie. Durch ein mutiertes Cas ohne funktionsfähige Endonuklease (dCas9) kann ein DNA-bindendes Protein erzeugt werden, das unter anderem analog zur RNAi zu einem Knockdown von endogenen Genen führt,[122] z. B. durch Transformation mit einem Plasmid, aus dem Cas9 und eine CRISPR-RNA transkribiert wird (CRISPRi).[123] Ebenso kann im Zuge der CRISPRa mittels dCas9 als Fusionsprotein mit einem Aktivator an einer anderen, bestimmbaren Stelle im Genom eine Transkription eingeleitet werden. Anhand charakteristischer CRISPR-Sequenzen können CRISPR/Cas enthaltende Bakterienstämme identifiziert werden (spoligotyping). Ein grün fluoreszierendes Protein kann mit einer Cas-Mutante als Fusionsprotein zur Markierung von DNA-Sequenzen (auch repetitiver Sequenzen wie Telomere) verwendet werden.[124] Durch das CRISPR/Cas-System konnte die Herstellungszeit von Mäusen mit komplexen Genomveränderungen von bis zu zwei Jahren auf wenige Wochen verkürzt werden.[36]
Pflanzenzüchtung
Die CRISPR-Methode eröffnet Pflanzenzüchtern die Möglichkeit, Sorten von Nutzpflanzen auf eine leichtere, effizientere und flexiblere Art zu verändern.[125] Für mehrere Nutzpflanzen liegen bereits Studien vor, aber es sind noch keine CRISPR-Pflanzen auf dem Markt.[126][127][128]
Caribou Biosciences, die 2011 von Doudna und Charpentier gegründete Firma, ging eine strategische Partnerschaft mit DuPont Pioneer ein. Abhängig vom Ausgang des Patentstreits könnte DuPont daher die Rechte an der Anwendung der Methode bei wichtigen Nutzpflanzen wie Mais, Raps und Sojabohne erhalten und Caribou für kleinere Märkte wie Obst und Gemüse. Unklar ist auch, wie sich der letztendliche Patentinhaber hinsichtlich der Lizenzierung der Methode verhalten wird.[126][127] Im September 2016 vergab das Broad-Institut eine (nicht exklusive) Lizenz zur Anwendung der Methode an Monsanto. Von der Lizenz ausgeschlossen sind Gene Drive sowie Anwendungen bei Tabak und die Herstellung steriler Pflanzen.[129]
Gleichzeitig besteht weiterhin Unklarheit bezüglich der Regulierung von CRISPR-Pflanzen (ausschließlich mit Deletionen, ohne Insertionen) in verschiedenen Ländern. Die für die mögliche zukünftige kommerzielle Nutzung von CRISPR-Pflanzen in der Landwirtschaft entscheidende Frage ist, ob diese rechtlich als gentechnisch veränderte Pflanzen angesehen werden, sofern nur etwas aus dem Genom entfernt und kein Transgen eingefügt wurde, da gentechnisch veränderte Pflanzen insbesondere in der EU sehr streng reguliert sind.[126][127][128] Das US-Landwirtschaftsministerium verkündete bereits im April 2016, zwei mit der CRISPR-Methode hergestellte Organismen, einen nicht-bräunenden Champignon und einen Wachsmais mit verändertem Stärkegehalt, nicht zu regulieren.[130] 2021 gingen in Japan Tomaten mit per CRISPR fünffach erhöhtem Gehalt an, möglicherweise leicht beruhigend wirkendem,[131] GABA auf den öffentlichen Markt.[132]
Tierzüchtung
Durch die CRISPR/Cas-Methode kann zum Beispiel das Geschlecht von Tieren beeinflusst werden. In der Schweinemast könnte dadurch das Problem des Ebergeruchs angegangen werden.[133]
Bekämpfung von Insekten
Entdeckungsgeschichte
Siehe auch: CRISPR#Entdeckung und Eigenschaften
Die Entdeckung und Erforschung der CRISPR-Sequenzen und des damit verbundenen CRISPR/Cas-Systems im Immunsystem verschiedener Bakterien und Archaea erfolgte in mehreren Schritten seit es im Jahr 1987 zum ersten Mal beschrieben wurde.[51] Obwohl die Funktion von CRISPR/Cas noch nicht bekannt war, wurde es zu Beginn der 1990er Jahre für das sog. Spoligotyping (die Genom-Typisierung von Bakterien-Isolaten über die Erkennung der unterschiedlichen Spacer innerhalb der direct-repeat-region) genutzt.[51]
Vor allem in den frühen 2000ern wurden die Zusammenhänge zwischen den CRISPR-Sequenzen der DNA und den cas-Genen sowie ihre Bedeutung in der Immunabwehr der Bakterien identifiziert. Ab 2008 war bekannt, dass das adaptive CRISPR DNA bindet.[134]
Im Jahr 2011 zeigte eine Arbeitsgruppe um Emmanuelle Charpentier und Jennifer Doudna, dass mittels des CRISPR/Cas-Systems der Mikroorganismen spezifische DNA-Ziele in vitro geschnitten werden können.[51][135] Sie reichten ihre wissenschaftliche Arbeit am 8. Juni 2012 bei der Fachzeitschrift Science ein, wo sie am 28. Juni veröffentlicht wurde.[1] Parallel zu ihnen arbeitete eine Arbeitsgruppe um Virginijus Šikšnys an der Methode, die bereits im April 2012 ihre Arbeit bei Cell einreichten; diese wurde jedoch abgelehnt, wenngleich die Herausgeber von Cell dem Artikel nachträglich eine große Bedeutung zuschrieben.[136] Im Mai reichten Šikšnys und Kollegen das Papier in den Proceedings of the National Academy of Sciences (PNAS) ein, wo es am 4. September online veröffentlicht wurde.[137]
Doudna und Charpentier beschrieben, wie sich in einem Bakterium gezielt Abschnitte aus dem Erbgut entfernen lassen. Dem Neurowissenschaftler Feng Zhang vom Massachusetts Institute of Technology gelang es später, die CRISPR-Methode nicht nur im Bakterium anzuwenden, sondern für alle Zellen zu optimieren.[138] Der Leiter des Broad Institute und Vorgesetzte von Feng Zhang, Eric Lander, verfasste im Januar 2016 einen Artikel über die Anteile der verschiedenen Wissenschaftler an der Entdeckung des CRISPR/Cas-Systems,[139] der aufgrund von einseitiger Darstellung und eines vermuteten Interessenskonflikts kritisiert wurde.[140][141][142]
Charpentier und Doudna erhielten 2014 für die Entdeckung der CRISPR/Cas-Methode den mit drei Millionen Dollar für jeden Preisträger dotierten Breakthrough Prize in Life Sciences[143] des Jahres 2015 und wurden mit zahlreichen weiteren Preisen bedacht. Im Jahr 2020 erhielten sie den Nobelpreis für Chemie. Innerhalb der ersten fünf Jahre nach Veröffentlichung ist die Methode eine Standardmethode in Laboren geworden und es wurden darüber in diesen fünf Jahren 2.500 wissenschaftliche Veröffentlichungen publiziert.[144]
Patentstreit
Sowohl Doudna und Charpentier (University of California, Berkeley) als auch Zhang (Broad) beantragten grundlegende Patente auf die CRISPR/Cas-Methode. Doudna und Charpentier reichten ihren Antrag beim United States Patent and Trademark Office (USPTO) im Mai 2012, Zhang im Dezember 2012 ein, und Zhang beantragte ein Schnellverfahren. Zhang wurden im Mai 2014 Patentrechte zugesprochen. Das USPTO begründete diese Entscheidung damit, dass Zhang die Methode für alle Zellen tauglich machte.[138] Doudna und Charpentier reichten Klage beim USPTO gegen diese Entscheidung ein. Das Verfahren zur Klärung der Urheberschaft lief im Januar 2016 an. Broad argumentiert, Berkeley habe die Methode zwar für Prokaryoten (Bakterien), aber nicht hinreichend für Eukaryoten (z. B. Mäuse und menschliche Zellen) beschrieben. Berkeley argumentiert, der Schritt von Pro- zu Eukaryoten bedürfe keiner erfinderischen Tätigkeit.[145] Im Februar 2017 entschied das USPTO zugunsten von Broad und lehnte die Klage der University of California mit der Begründung ab, dass die Anwendung der von Doudna und Charpentier beschriebenen CRISPR/Cas-Methode auf Eukaryoten nicht offensichtlich sei.[146]
Auch das Europäische Patentamt (EPA) muss im Patentstreit Entscheidungen fällen. Hinsichtlich des Berkeley-Patentantrags hat das EPA bereits argumentiert, der Antrag habe die Erfindung nicht ausreichend beschrieben, da die Hervorhebung der Rolle der PAM-Sequenzen fehle. Berkeley vertritt die Ansicht, dass diese Rolle für Fachleute offensichtlich ist. Dem Broad-Institut wurden hingegen bereits mehrere Patente an der CRISPR/Cas-Methode zugesprochen, die jedoch von mehreren Seiten angefochten wurden. Kläger verweisen zum Beispiel darauf, dass Broads ursprünglicher Patentantrag einen Wissenschaftler der Rockefeller University als an der Erfindung Mitbeteiligten erwähnte, eine spätere Version des Antrags jedoch nicht.[145]
Risiken
Die Methode ist eine verhältnismäßig einfache, preiswerte, leicht verfügbare, punktgenaue und effiziente Technik. In den ersten Jahren war nicht geregelt, ob reine Deletionen, die auch durch zufällige Mutagenese im Rahmen einer Züchtung entstehen können (jedoch bei der Züchtung nicht zielgerichtet), als Gentechnik zu bewerten sind. Kritiker weisen darauf hin, dass es beispielsweise internationaler Standards und Vorsorgemaßnahmen bedürfe, um Wildwuchs und Missbrauch vorzubeugen. Hier bestehen auch Befürchtungen vor kriminellen oder terroristischen Anwendungen; das amerikanische FBI beispielsweise beobachtet entsprechende mehr oder weniger private Do-it-Yourself (DIY)-„Garagen-Bastler“ („Bio-Hacker“). In Bezug auf den Eingriff in die menschliche Keimbahn durch Verwendung einer CRISPR/Cas-Methode auf menschlichen Keimzellen in Verbindung mit einer In-vitro-Fertilisation im Rahmen einer Gentherapie gibt es bioethische Bedenken.[147] Der frankokanadische Neurologe Jean-François Gariépy warnt in der Monografie The revolutionary phenotype vor einer Genmodifizierung von Menschen. Aufgrund der Komplexität von Genmodifikationen sei eine auf Computerprogramme gestützte Änderung des menschlichen Erbguts unumgänglich. Ausgehend von der RNA-Welt-Hypothese, wonach RNA-basierte Lebensformen durch DNA-basierte abgelöst wurden, argumentiert Gariépy, dass eine solche Vorgehensweise auf lange Sicht ebenfalls zu einer revolutionären Umwandlung der menschlichen Lebensform mit nicht absehbaren Folgen führen könne.[148] Der Deutsche Ethikrat warnt vor „unerwünschten gesundheitlichen Folgen“ angesichts noch unausgereifter Verfahren. Zudem bestünden nicht absehbare ethische Folgen.[149]
Recht
Der Europäische Gerichtshof (EuGH) entschied am 25. Juli 2018, dass grundsätzlich auch mit der CRISPR/Cas-Methode (Mutagenese) bearbeitete Pflanzen ohne Fremd-DNA als gentechnisch veränderte Organismen (GVO) anzusehen sind und grundsätzlich den in der GVO-Richtlinie vorgesehenen Verpflichtungen unterliegen (Az. C-528/16). Geklagt hatte die französische Bauerngewerkschaft Confédération paysanne und weitere acht Verbände gegen die französische Regierung.[150]
Das Urteil wurde von Umweltschützern gelobt, während Naturwissenschaftler die Auswirkungen kritisch beurteilten, da die Entfernung von DNA auch durch klassische Methoden der Züchtung erreicht würde, wie eine Behandlung mit radioaktiver Strahlung oder mit erbgutverändernden Chemikalien (deren Erzeugnisse nicht als GVO klassifiziert werden), allerdings erfolgen klassische Methoden nach dem Zufallsprinzip.[151] Es sei fraglich, ob von nun an noch Forschungsförderung für entsprechende Projekte gewährt werde. Zudem ist die Zulassung aufwändiger.[151] Auch sei die diesbezügliche Forschung innerhalb der EU für Unternehmen unter den neuen rechtlichen Rahmenbedingungen nicht mehr profitabel. Deshalb werde sie nicht mehr innerhalb der EU erfolgen.[152] Weiterhin wurden nachteilige Auswirkungen auf den Welthandel befürchtet.[153]
Literatur
- M. Belfort, R. P. Bonocora: Homing endonucleases: from genetic anomalies to programmable genomic clippers. In: Methods in molecular biology (Clifton, N.J.). Band 1123, 2014, S. 1–26, doi:10.1007/978-1-62703-968-0_1, PMID 24510256, PMC 4436680 (freier Volltext).
- James Kozubek: Modern Prometheus. Cambridge University Press, 2016, ISBN 978-1-316-78098-5.
Weblinks
- Deutschlandfunk Das Feature, 5. Dezember 2017, Peter Kreysler: Schöpfung mit der Genschere – Die DNA-Revolution
- Leopoldina: Genomchirurgie CRISPR
- youtube.com: Gen-editing mit CRISPR/Cas9: Animation zum Prinzip der Methode auf dem youtube-Kanal der Max-Planck-Gesellschaft, 10. Februar 2016, mit englischen Untertiteln
- spektrum.de, 24. Juni 2015, Heidi Ledford: Werkzeug der Genmanipulation – Gentechnik: CRISPR verändert alles
- wissenschaft.de, 3. Juni 2019, Daniela Albat: Crispr-Babys: Fataler Eingriff?
Einzelnachweise
- Martin Jinek, Krzysztof Chylinski, Ines Fonfara, Michael Hauer, Jennifer A. Doudna, Emmanuelle Charpentier: A Programmable Dual-RNA–Guided DNA Endonuclease in Adaptive Bacterial Immunity. In: Science. Band 337, Nr. 6096, 17. August 2012, ISSN 0036-8075, S. 816–821, doi:10.1126/science.1225829, PMID 22745249.
- H. Ochiai: Single-Base Pair Genome Editing in Human Cells by Using Site-Specific Endonucleases. In: International journal of molecular sciences. Band 16, Nummer 9, 2015, S. 21128–21137, doi:10.3390/ijms160921128, PMID 26404258, PMC 4613245 (freier Volltext) (Review).
- M. M. Mahfouz, A. Piatek, C. N. Stewart: Genome engineering via TALENs and CRISPR/Cas9 systems: challenges and perspectives. In: Plant biotechnology journal. Band 12, Nummer 8, Oktober 2014, S. 1006–1014, doi:10.1111/pbi.12256, PMID 25250853.
- E. Pennisi: The CRISPR craze. In: Science. Band 341, Nummer 6148, August 2013, S. 833–836, doi:10.1126/science.341.6148.833, PMID 23970676.
- Email Science: And Science’s Breakthrough of the Year is … In: news.sciencemag.org. 17. Dezember 2015, abgerufen am 17. Dezember 2015 (englisch).
- Jennifer Doudna, Emmanuelle Charpentier.: The new frontier of genome engineering with CRISPR-Cas9. Science 346(6213):1077 (2014), PMID 25430774, doi:10.1126/science.1258096.
- R. Sorek, V. Kunin, P. Hugenholtz: CRISPR–a widespread system that provides acquired resistance against phages in bacteria and archaea. In: Nature reviews. Microbiology. Band 6, Nummer 3, März 2008, S. 181–186, ISSN 1740-1534. doi:10.1038/nrmicro1793. PMID 18157154.
- D. Rath, L. Amlinger, A. Rath, M. Lundgren: The CRISPR-Cas immune system: biology, mechanisms and applications. In: Biochimie. Band 117, Oktober 2015, S. 119–128, doi:10.1016/j.biochi.2015.03.025, PMID 25868999 (Review).
- G. Gasiunas, R. Barrangou, P. Horvath, V. Siksnys: Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. In: Proceedings of the National Academy of Sciences. Band 109, Nummer 39, September 2012, S. E2579–E2586, ISSN 1091-6490. doi:10.1073/pnas.1208507109. PMID 22949671. PMC 3465414 (freier Volltext).
- V. Kunin, R. Sorek, P. Hugenholtz: Evolutionary conservation of sequence and secondary structures in CRISPR repeats. In: Genome biology. Band 8, Nummer 4, 2007, S. R61, ISSN 1465-6914. doi:10.1186/gb-2007-8-4-r61. PMID 17442114. PMC 1896005 (freier Volltext).
- F. Jiang, D. W. Taylor, J. S. Chen, J. E. Kornfeld, K. Zhou, A. J. Thompson, E. Nogales, J. A. Doudna: Structures of a CRISPR-Cas9 R-loop complex primed for DNA cleavage. In: Science. Band 351, Nummer 6275, Februar 2016, S. 867–871, doi:10.1126/science.aad8282, PMID 26841432.
- S. H. Sternberg, S. Redding, M. Jinek, E. C. Greene, J. A. Doudna: DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. In: Nature. Band 507, Nummer 7490, März 2014, S. 62–67, doi:10.1038/nature13011, PMID 24476820, PMC 4106473 (freier Volltext).
- E. Deltcheva, K. Chylinski, C. M. Sharma, K. Gonzales, Y. Chao, Z. A. Pirzada, M. R. Eckert, J. Vogel, E. Charpentier: CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. In: Nature. Band 471, Nummer 7340, März 2011, S. 602–607, doi:10.1038/nature09886, PMID 21455174, PMC 3070239 (freier Volltext).
- S. J. Brouns: Molecular biology. A Swiss army knife of immunity. In: Science. Band 337, Nummer 6096, August 2012, S. 808–809, doi:10.1126/science.1227253, PMID 22904002.
- Researchers establish new viable CRISPR-Cas12b system for plant genome engineering (en-us). In: phys.org. Abgerufen am 6. April 2020.
- Meiling Ming, Qiurong Ren, Changtian Pan, Yao He, Yingxiao Zhang, Shishi Liu, Zhaohui Zhong, Jiaheng Wang, Aimee A. Malzahn, Jun Wu, Xuelian Zheng, Yong Zhang, Yiping Qi: CRISPR–Cas12b enables efficient plant genome engineering. In: Nature Plants. 6, Nr. 3, March 2020, S. 202–208. doi:10.1038/s41477-020-0614-6. PMID 32170285.
- RNA editing with CRISPR-Cas13. In: Science. 358, Nr. 6366, November 2017, S. 1019–1027. bibcode:2017Sci...358.1019C. doi:10.1126/science.aaq0180. PMID 29070703. PMC 5793859 (freier Volltext).
- CRISPR-Cas3 innovation holds promise for disease cures, advancing science (en). In: Cornell Chronicle. Abgerufen am 24. Oktober 2021.
- Adam E. Dolan, Zhonggang Hou, Yibei Xiao, Max J. Gramelspacher, Jaewon Heo, Sara E. Howden, Peter L. Freddolino, Ailong Ke, Yan Zhang: Introducing a Spectrum of Long-Range Genomic Deletions in Human Embryonic Stem Cells Using Type I CRISPR-Cas. In: Molecular Cell. 74, Nr. 5, 6. Juni 2019, ISSN 1097-2765, S. 936–950.e5. doi:10.1016/j.molcel.2019.03.014.
- Researchers develop an engineered 'mini' CRISPR genome editing system (en). In: phys.org. Abgerufen am 18. Oktober 2021.
- Xiaoshu Xu, Augustine Chemparathy, Leiping Zeng, Hannah R. Kempton, Stephen Shang, Muneaki Nakamura, Lei S. Qi: Engineered miniature CRISPR-Cas system for mammalian genome regulation and editing. In: Molecular Cell. 3. September 2021, ISSN 1097-2765. doi:10.1016/j.molcel.2021.08.008.
- Heidi Ledford: Alternative CRISPR system could improve genome editing. In: Nature. 526, 2015, S. 17, doi:10.1038/nature.2015.18432.
- cas12b - CRISPR-associated endonuclease Cas12b - Alicyclobacillus acidoterrestris (strain ATCC 49025 / DSM 3922 / CIP 106132 / NCIMB 13137 / GD3B) - cas12b gene. In: uniprot.org. 16. Oktober 2013, abgerufen am 24. Januar 2019 (englisch).
- M. R. O’Connell, B. L. Oakes, S. H. Sternberg, A. East-Seletsky, M. Kaplan, J. A. Doudna: Programmable RNA recognition and cleavage by CRISPR/Cas9. In: Nature. Band 516, Nummer 7530, Dezember 2014, S. 263–266, ISSN 1476-4687. doi:10.1038/nature13769. PMID 25274302.
- C. R. Hale, P. Zhao, S. Olson, M. O. Duff, B. R. Graveley, L. Wells, R. M. Terns, M. P. Terns: RNA-guided RNA cleavage by a CRISPR RNA-Cas protein complex. In: Cell. Band 139, Nummer 5, November 2009, S. 945–956, doi:10.1016/j.cell.2009.07.040, PMID 19945378, PMC 2951265 (freier Volltext).
- Rodolphe Barrangou, A. Birmingham, S. Wiemann, R. L. Beijersbergen, V. Hornung, A. v. Smith: Advances in CRISPR-Cas9 genome engineering: lessons learned from RNA interference. In: Nucleic acids research. Band 43, Nummer 7, April 2015, S. 3407–3419, doi:10.1093/nar/gkv226, PMID 25800748, PMC 4402539 (freier Volltext).
- M. Mikami, S. Toki, M. Endo: Precision Targeted Mutagenesis via Cas9 Paired Nickases in Rice. In: Plant & cell physiology. Band 57, Nummer 5, Mai 2016, S. 1058–1068, doi:10.1093/pcp/pcw049, PMID 26936792, PMC 4867050 (freier Volltext).
- A. C. Komor, Y. B. Kim, M. S. Packer, J. A. Zuris, D. R. Liu: Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. In: Nature. Band 533, Nummer 7603, Mai 2016, S. 420–424, doi:10.1038/nature17946, PMID 27096365, PMC 4873371 (freier Volltext).
- Gaudelli, N. M., et al. (2017): Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature 551(7681): 464-471. doi:10.1038/nature24644.
- N. Rudin, E. Sugarman, J. E. Haber: Genetic and physical analysis of double-strand break repair and recombination in Saccharomyces cerevisiae. In: Genetics. Band 122, Nummer 3, Juli 1989, S. 519–534, ISSN 0016-6731. PMID 2668114. PMC 1203726 (freier Volltext).
- P. D. Hsu, E. S. Lander, F. Zhang: Development and Applications of CRISPR-Cas9 for Genome Engineering. In: Cell. Band 157, Nummer 6, Juni 2014, S. 1262–1278, ISSN 1097-4172. doi:10.1016/j.cell.2014.05.010. PMID 24906146. PDF.
- W. Jiang, D. Bikard, D. Cox, F. Zhang, L. A. Marraffini: RNA-guided editing of bacterial genomes using CRISPR-Cas systems. In: Nature Biotechnology. Band 31, Nummer 3, März 2013, S. 233–239, ISSN 1546-1696. doi:10.1038/nbt.2508. PMID 23360965. PMC 3748948 (freier Volltext).
- J. E. DiCarlo, J. E. Norville, P. Mali, X. Rios, J. Aach, G. M. Church: Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. In: Nucleic acids research. Band 41, Nummer 7, April 2013, S. 4336–4343, ISSN 1362-4962. doi:10.1093/nar/gkt135. PMID 23460208. PMC 3627607 (freier Volltext).
- K. J. Beumer, D. Carroll: Targeted genome engineering techniques in Drosophila. In: Methods (San Diego, Calif.). Band 68, Nummer 1, Juni 2014, S. 29–37, doi:10.1016/j.ymeth.2013.12.002, PMID 24412316, PMC 4048800 (freier Volltext) (Review).
- W. Y. Hwang, Y. Fu, D. Reyon, M. L. Maeder, S. Q. Tsai, J. D. Sander, R. T. Peterson, J. R. Yeh, J. K. Joung: Efficient genome editing in zebrafish using a CRISPR-Cas system. In: Nature Biotechnology. Band 31, Nummer 3, März 2013, S. 227–229, ISSN 1546-1696. doi:10.1038/nbt.2501. PMID 23360964. PMC 3686313 (freier Volltext).
- H. Wang, H. Yang, C. S. Shivalila, M. M. Dawlaty, A. W. Cheng, F. Zhang, R. Jaenisch: One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. In: Cell. Band 153, Nummer 4, Mai 2013, S. 910–918, ISSN 1097-4172. doi:10.1016/j.cell.2013.04.025. PMID 23643243. PMC 3969854 (freier Volltext).
- P. Mali, L. Yang, K. M. Esvelt, J. Aach, M. Guell, J. E. DiCarlo, J. E. Norville, G. M. Church: RNA-guided human genome engineering via Cas9. In: Science. Band 339, Nummer 6121, Februar 2013, S. 823–826, ISSN 1095-9203. doi:10.1126/science.1232033. PMID 23287722. PMC 3712628 (freier Volltext).
- Puping Liang, Yanwen Xu, Xiya Zhang, Chenhui Ding, Rui Huang, Zhen Zhang, Jie Lv, Xiaowei Xie, Yuxi Chen, Yujing Li, Ying Sun, Yaofu Bai, Zhou Songyang, Wenbin Ma, Canquan Zhou, Junjiu Huang: CRISPR/Cas9-mediated gene editing in human tripronuclear zygotes. In: Protein & Cell. 6, 2015, S. 363, doi:10.1007/s13238-015-0153-5.
- L. Arora, A. Narula: Gene Editing and Crop Improvement Using CRISPR-Cas9 System. In: Frontiers in plant science. Band 8, 2017, S. 1932, doi:10.3389/fpls.2017.01932, PMID 29167680, PMC 5682324 (freier Volltext).
- Y. Fu, J. A. Foden, C. Khayter, M. L. Maeder, D. Reyon, J. K. Joung, J. D. Sander: High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. In: Nature Biotechnology. Band 31, Nummer 9, September 2013, S. 822–826, doi:10.1038/nbt.2623, PMID 23792628, PMC 3773023 (freier Volltext).
- P. D. Hsu, D. A. Scott, J. A. Weinstein, F. A. Ran, S. Konermann, V. Agarwala, Y. Li, E. J. Fine, X. Wu, O. Shalem, T. J. Cradick, L. A. Marraffini, G. Bao, F. Zhang: DNA targeting specificity of RNA-guided Cas9 nucleases. In: Nature Biotechnology. Band 31, Nummer 9, September 2013, S. 827–832, doi:10.1038/nbt.2647, PMID 23873081, PMC 3969858 (freier Volltext).
- S. W. Cho, S. Kim, Y. Kim, J. Kweon, H. S. Kim, S. Bae, J. S. Kim: Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases. In: Genome research. Band 24, Nummer 1, Januar 2014, S. 132–141, doi:10.1101/gr.162339.113, PMID 24253446, PMC 3875854 (freier Volltext).
- A. Veres, B. S. Gosis, Q. Ding, R. Collins, A. Ragavendran, H. Brand, S. Erdin, C. A. Cowan, M. E. Talkowski, K. Musunuru: Low incidence of off-target mutations in individual CRISPR-Cas9 and TALEN targeted human stem cell clones detected by whole-genome sequencing. In: Cell stem cell. Band 15, Nummer 1, Juli 2014, S. 27–30, doi:10.1016/j.stem.2014.04.020, PMID 24996167, PMC 4082799 (freier Volltext).
- H. Wang, H. Yang, C. S. Shivalila, M. M. Dawlaty, A. W. Cheng, F. Zhang, R. Jaenisch: One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. In: Cell. Band 153, Nummer 4, Mai 2013, S. 910–918, doi:10.1016/j.cell.2013.04.025, PMID 23643243, PMC 3969854 (freier Volltext).
- Y. Lin, T. J. Cradick, M. T. Brown, H. Deshmukh, P. Ranjan, N. Sarode, B. M. Wile, P. M. Vertino, F. J. Stewart, G. Bao: CRISPR/Cas9 systems have off-target activity with insertions or deletions between target DNA and guide RNA sequences. In: Nucleic acids research. Band 42, Nummer 11, Juni 2014, S. 7473–7485, doi:10.1093/nar/gku402, PMID 24838573, PMC 4066799 (freier Volltext).
- T. J. Cradick, E. J. Fine, C. J. Antico, G. Bao: CRISPR/Cas9 systems targeting β-globin and CCR5 genes have substantial off-target activity. In: Nucleic acids research. Band 41, Nummer 20, November 2013, S. 9584–9592, doi:10.1093/nar/gkt714, PMID 23939622, PMC 3814385 (freier Volltext).
- D. Kim, S. Bae, J. Park, E. Kim, S. Kim, H. R. Yu, J. Hwang, J. I. Kim, J. S. Kim: Digenome-seq: genome-wide profiling of CRISPR-Cas9 off-target effects in human cells. In: Nature methods. Band 12, Nummer 3, März 2015, S. 237–43, 1 p following 243, doi:10.1038/nmeth.3284, PMID 25664545.
- P. D. Hsu, E. S. Lander, F. Zhang: Development and applications of CRISPR-Cas9 for genome engineering. In: Cell. Band 157, Nummer 6, Juni 2014, S. 1262–1278, doi:10.1016/j.cell.2014.05.010, PMID 24906146, PMC 4343198 (freier Volltext).
- L. Cong, F. Zhang: Genome engineering using CRISPR-Cas9 system. In: Methods in molecular biology. Band 1239, 2015, S. 197–217, doi:10.1007/978-1-4939-1862-1_10, PMID 25408407.
- A. Eid, M. M. Mahfouz: Genome editing: the road of CRISPR/Cas9 from bench to clinic. In: Experimental & molecular medicine. Band 48, Nummer 10, 10 2016, S. e265, doi:10.1038/emm.2016.111, PMID 27741224, PMC 5099421 (freier Volltext).
- Annika Hardt: Technikfolgenabschätzung des CRISPR/Cas-Systems, Über die Anwendung in der menschlichen Keimbahn. De Gruyter, Berlin, Boston 2019, ISBN 978-3-11-062170-9 (degruyter.com [abgerufen am 9. Oktober 2019]).
- Y. Fu, J. D. Sander, D. Reyon, V. M. Cascio, J. K. Joung: Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. In: Nature Biotechnology. Band 32, Nummer 3, März 2014, S. 279–284, doi:10.1038/nbt.2808, PMID 24463574, PMC 3988262 (freier Volltext).
- K. Standage-Beier, Q. Zhang, X. Wang: Targeted Large-Scale Deletion of Bacterial Genomes Using CRISPR-Nickases. In: ACS synthetic biology. Band 4, Nummer 11, November 2015, S. 1217–1225, doi:10.1021/acssynbio.5b00132, PMID 26451892, PMC 4655420 (freier Volltext).
- S. Q. Tsai, N. Wyvekens, C. Khayter, J. A. Foden, V. Thapar, D. Reyon, M. J. Goodwin, M. J. Aryee, J. K. Joung: Dimeric CRISPR RNA-guided FokI nucleases for highly specific genome editing. In: Nature Biotechnology. Band 32, Nummer 6, Juni 2014, S. 569–576, doi:10.1038/nbt.2908, PMID 24770325, PMC 4090141 (freier Volltext).
- V. Pattanayak, J. P. Guilinger, D. R. Liu: Determining the specificities of TALENs, Cas9, and other genome-editing enzymes. In: Methods in enzymology. Band 546, 2014, S. 47–78, doi:10.1016/B978-0-12-801185-0.00003-9, PMID 25398335, PMC 4440668 (freier Volltext).
- J. Zischewski, R. Fischer, L. Bortesi: Detection of on-target and off-target mutations generated by CRISPR/Cas9 and other sequence-specific nucleases. In: Biotechnology Advances. Band 35, Nummer 1, 2017 Jan – Feb, S. 95–104, doi:10.1016/j.biotechadv.2016.12.003, PMID 28011075.
- S. Q. Tsai, N. T. Nguyen, J. Malagon-Lopez, V. V. Topkar, M. J. Aryee, J. K. Joung: CIRCLE-seq: a highly sensitive in vitro screen for genome-wide CRISPR-Cas9 nuclease off-targets. In: Nature methods. Band 14, Nummer 6, Juni 2017, S. 607–614, doi:10.1038/nmeth.4278, PMID 28459458, PMC 5924695 (freier Volltext).
- P. Cameron, C. K. Fuller, P. D. Donohoue, B. N. Jones, M. S. Thompson, M. M. Carter, S. Gradia, B. Vidal, E. Garner, E. M. Slorach, E. Lau, L. M. Banh, A. M. Lied, L. S. Edwards, A. H. Settle, D. Capurso, V. Llaca, S. Deschamps, M. Cigan, J. K. Young, A. P. May: Mapping the genomic landscape of CRISPR-Cas9 cleavage. In: Nature methods. Band 14, Nummer 6, Juni 2017, S. 600–606, doi:10.1038/nmeth.4284, PMID 28459459.
- R. Singh, C. Kuscu, A. Quinlan, Y. Qi, M. Adli: Cas9-chromatin binding information enables more accurate CRISPR off-target prediction. In: Nucleic acids research. Band 43, Nummer 18, Oktober 2015, S. e118, doi:10.1093/nar/gkv575, PMID 26032770, PMC 4605288 (freier Volltext).
- S. Q. Tsai, Z. Zheng, N. T. Nguyen, M. Liebers, V. V. Topkar, V. Thapar, N. Wyvekens, C. Khayter, A. J. Iafrate, L. P. Le, M. J. Aryee, J. K. Joung: GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. In: Nature Biotechnology. Band 33, Nummer 2, Februar 2015, S. 187–197, doi:10.1038/nbt.3117, PMID 25513782, PMC 4320685 (freier Volltext).
- F. A. Ran, P. D. Hsu, C. Y. Lin, J. S. Gootenberg, S. Konermann, A. E. Trevino, D. A. Scott, A. Inoue, S. Matoba, Y. Zhang, F. Zhang: Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity. In: Cell. Band 154, Nummer 6, September 2013, S. 1380–1389, doi:10.1016/j.cell.2013.08.021, PMID 23992846, PMC 3856256 (freier Volltext).
- K. Ishida, P. Gee, A. Hotta: Minimizing off-Target Mutagenesis Risks Caused by Programmable Nucleases. In: International journal of molecular sciences. Band 16, Nummer 10, 2015, S. 24751–24771, doi:10.3390/ijms161024751, PMID 26501275, PMC 4632775 (freier Volltext) (Review).
- H. O'Geen, A. S. Yu, D. J. Segal: How specific is CRISPR/Cas9 really? In: Current opinion in chemical biology. [elektronische Veröffentlichung vor dem Druck] Oktober 2015, doi:10.1016/j.cbpa.2015.10.001, PMID 26517564.
- T. Koo, J. Lee, J. S. Kim: Measuring and Reducing Off-Target Activities of Programmable Nucleases Including CRISPR-Cas9. In: Molecules and cells. Band 38, Nummer 6, Juni 2015, S. 475–481, doi:10.14348/molcells.2015.0103, PMID 25985872, PMC 4469905 (freier Volltext) (Review).
- Y. Ma, L. Zhang, X. Huang: Genome modification by CRISPR/Cas9. In: The FEBS journal. Band 281, Nummer 23, Dezember 2014, S. 5186–5193, doi:10.1111/febs.1311, PMID 25315507 (Review).
- P. Mali, J. Aach, P. B. Stranges, K. M. Esvelt, M. Moosburner, S. Kosuri, L. Yang, G. M. Church: CAS9 transcriptional activators for target specificity screening and paired nickases for cooperative genome engineering. In: Nature Biotechnology. Band 31, Nummer 9, September 2013, S. 833–838, doi:10.1038/nbt.2675, PMID 23907171, PMC 3818127 (freier Volltext).
- J. Lee, J. H. Chung, H. M. Kim, D. W. Kim, H. Kim: Designed nucleases for targeted genome editing. In: Plant biotechnology journal. Band 14, Nummer 2, Februar 2016, S. 448–462, doi:10.1111/pbi.12465, PMID 26369767.
- B. P. Kleinstiver, M. S. Prew, S. Q. Tsai, V. V. Topkar, N. T. Nguyen, Z. Zheng, A. P. Gonzales, Z. Li, R. T. Peterson, J. R. Yeh, M. J. Aryee, J. K. Joung: Engineered CRISPR-Cas9 nucleases with altered PAM specificities. In: Nature. Band 523, Nummer 7561, Juli 2015, S. 481–485, doi:10.1038/nature14592, PMID 26098369, PMC 4540238 (freier Volltext).
- B. P. Kleinstiver, V. Pattanayak, M. S. Prew, S. Q. Tsai, N. T. Nguyen, Z. Zheng, J. K. Joung: High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects. In: Nature. Band 529, Nummer 7587, Januar 2016, S. 490–495, doi:10.1038/nature16526, PMID 26735016, PMC 4851738 (freier Volltext).
- Guilinger, J. P., Thompson, D. B. & Liu, D. R. (2014). Fusion of catalytically inactive Cas9 to FokI nuclease improves the specificity of genome modification. Nature Biotechnol. 32, 577–582 doi:10.1038/nbt.2909
- N. Wyvekens, V. V. Topkar, C. Khayter, J. K. Joung, S. Q. Tsai: Dimeric CRISPR RNA-Guided FokI-dCas9 Nucleases Directed by Truncated gRNAs for Highly Specific Genome Editing. In: Human gene therapy. Band 26, Nummer 7, Juli 2015, S. 425–431, doi:10.1089/hum.2015.084, PMID 26068112, PMC 4509490 (freier Volltext).
- Jonathan Strecker, Sara Jones, Balwina Koopal, Jonathan Schmid-Burgk, Bernd Zetsche, Linyi Gao, Kira S. Makarova, Eugene V. Koonin & Feng Zhang: Engineering of CRISPR-Cas12b for human genome editing. In: Nature Communications. Band 10, Nummer 1, Januar 2019, S. 212, doi:10.1038/s41467-018-08224-4, PMID 30670702.
- Andrew V. Anzalone, Peyton B. Randolph, Jessie R. Davis, Alexander A. Sousa, Luke W. Koblan, Jonathan M. Levy, Peter J. Chen, Christopher Wilson, Gregory A. Newby, Aditya Raguram, David R. Liu: Search-and-replace genome editing without double-strand breaks or donor DNA. In: Nature., doi:10.1038/s41586-019-1711-4.
- K. L. Maxwell: The Anti-CRISPR Story: A Battle for Survival. In: Molecular cell. Band 68, Nummer 1, Oktober 2017, S. 8–14, doi:10.1016/j.molcel.2017.09.002, PMID 28985512.
- A. Pawluk, N. Amrani, Y. Zhang, B. Garcia, Y. Hidalgo-Reyes, J. Lee, A. Edraki, M. Shah, E. J. Sontheimer, K. L. Maxwell, A. R. Davidson: Naturally Occurring Off-Switches for CRISPR-Cas9. In: Cell. Band 167, Nummer 7, Dezember 2016, S. 1829–1838.e9, doi:10.1016/j.cell.2016.11.017, PMID 27984730, PMC 5757841 (freier Volltext).
- F. Bubeck, M. D. Hoffmann, Z. Harteveld, S. Aschenbrenner, A. Bietz, M. C. Waldhauer, K. Börner, J. Fakhiri, C. Schmelas, L. Dietz, D. Grimm, B. E. Correia, R. Eils, D. Niopek: Engineered anti-CRISPR proteins for optogenetic control of CRISPR-Cas9. In: Nature methods. Band 15, Nummer 11, November 2018, S. 924–927, doi:10.1038/s41592-018-0178-9, PMID 30377362.
- M. D. Hoffmann, S. Aschenbrenner, S. Grosse, K. Rapti, C. Domenger, J. Fakhiri, M. Mastel, K. Börner, R. Eils, D. Grimm, D. Niopek: Cell-specific CRISPR-Cas9 activation by microRNA-dependent expression of anti-CRISPR proteins. In: Nucleic acids research. Band 47, Nummer 13, Juli 2019, S. e75, doi:10.1093/nar/gkz271, PMID 30982889, PMC 6648350 (freier Volltext).
- D. H. Haft, J. Selengut, E. F. Mongodin, K. E. Nelson: A guild of 45 CRISPR-associated (Cas) protein families and multiple CRISPR/Cas subtypes exist in prokaryotic genomes. In: PLoS computational biology. Band 1, Nummer 6, November 2005, S. e60, ISSN 1553-7358. doi:10.1371/journal.pcbi.0010060. PMID 16292354. PMC 1282333 (freier Volltext).
- Kira S. Makarova, Yuri I. Wolf, Eugene V. Koonin: Classification and Nomenclature of CRISPR-Cas Systems: Where from Here?. In: The CRISPR Journal. Band 1, 2018, S. 325, doi:10.1089/crispr.2018.0033.
- H. Nishimasu, O. Nureki: Structures and mechanisms of CRISPR RNA-guided effector nucleases. In: Current opinion in structural biology. Band 43, April 2017, S. 68–78, doi:10.1016/j.sbi.2016.11.013, PMID 27912110.
- D. W. Taylor, Y. Zhu, R. H. J. Staals, J. E. Kornfeld, A. Shinkai, J. van der Oost, E. Nogales, J. A. Doudna: Structures of the CRISPR-Cmr complex reveal mode of RNA target positioning. In: Science. 348, 2015, S. 581, doi:10.1126/science.aaa4535.
- Tomoyuki Numata, Hideko Inanaga, Chikara Sato, Takuo Osawa: Crystal Structure of the Csm3–Csm4 Subcomplex in the Type III-A CRISPR–Cas Interference Complex. In: Journal of Molecular Biology. Band 427, 2015, S. 259, doi:10.1016/j.jmb.2014.09.029.
- Wenyuan Han, Yingjun Li, Ling Deng, Mingxia Feng, Wenfang Peng, Søren Hallstrøm, Jing Zhang, Nan Peng, Yun Xiang Liang, Malcolm F. White, Qunxin She: A type III-B CRISPR-Cas effector complex mediating massive target DNA destruction. Band 45, Nummer 4, In: Nucleic Acids Research., S. 1983–1993, doi:10.1093/nar/gkw1274.
- J. van der Oost, E. R. Westra, R. N. Jackson, B. Wiedenheft: Unravelling the structural and mechanistic basis of CRISPR-Cas systems. In: Nature reviews. Microbiology. Band 12, Nummer 7, Juli 2014, S. 479–492, doi:10.1038/nrmicro3279, PMID 24909109, PMC 4225775 (freier Volltext).
- F. Ann Ran, L. e. Cong, Winston X. Yan, David A. Scott, Jonathan S. Gootenberg, Andrea J. Kriz, Bernd Zetsche, Ophir Shalem, Xuebing Wu, Kira S. Makarova, Eugene V. Koonin, Phillip A. Sharp, Feng Zhang: In vivo genome editing using Staphylococcus aureus Cas9. In: Nature. 520, 2015, S. 186, doi:10.1038/nature14299.
- A. Plagens, H. Richter, E. Charpentier, L. Randau: DNA and RNA interference mechanisms by CRISPR-Cas surveillance complexes. In: FEMS microbiology reviews. Band 39, Nummer 3, Mai 2015, S. 442–463, doi:10.1093/femsre/fuv019, PMID 25934119.
- Adi Stern, Rotem Sorek: The phage-host arms race: Shaping the evolution of microbes. In: BioEssays. Band 33, 2011, S. 43, doi:10.1002/bies.201000071.
- Eugene V. Koonin, Kira S. Makarova, Yuri I. Wolf: Evolutionary Genomics of Defense Systems in Archaea and Bacteria. In: Annual Review of Microbiology. Band 71, 2017, S. 233, doi:10.1146/annurev-micro-090816-093830.
- Kira S. Makarova, Nick V. Grishin, Svetlana A. Shabalina, Yuri I. Wolf, Eugene V. Koonin: A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action. In: Biology Direct. Band 1, S. 7, doi:10.1186/1745-6150-1-7.
- Donghyun Ka, Dong Man Jang, Byung Woo Han, Euiyoung Bae: Molecular organization of the type II-A CRISPR adaptation module and its interaction with Cas9 via Csn2. In: Nucleic Acids Research. 46, 2018, S. 9805, doi:10.1093/nar/gky702.
- Eugene V. Koonin: CRISPR: a new principle of genome engineering linked to conceptual shifts in evolutionary biology. In: Biology & Philosophy. Band 34, 2019, doi:10.1007/s10539-018-9658-7.
- Kira S. Makarova, Yuri I. Wolf, Omer S. Alkhnbashi: An updated evolutionary classification of CRISPR–Cas systems. In: Nature Reviews Microbiology. Band 13, 2015, S. 722, doi:10.1038/nrmicro3569.
- Sanjarbek Hudaiberdiev, Sergey Shmakov, Yuri I. Wolf, Michael P. Terns, Kira S. Makarova, Eugene V. Koonin: Phylogenomics of Cas4 family nucleases. In: BMC Evolutionary Biology. Band 17, 2017, doi:10.1186/s12862-017-1081-1.
- K. S. Makarova, Y. I. Wolf, J. Iranzo, S. A. Shmakov, O. S. Alkhnbashi, S. J. Brouns, E. Charpentier, D. Cheng, D. H. Haft, P. Horvath, S. Moineau, F. J. Mojica, D. Scott, S. A. Shah, V. Siksnys, M. P. Terns, Č. Venclovas, M. F. White, A. F. Yakunin, W. Yan, F. Zhang, R. A. Garrett, R. Backofen, J. van der Oost, R. Barrangou, E. V. Koonin: Evolutionary classification of CRISPR-Cas systems: a burst of class 2 and derived variants. In: Nature reviews. Microbiology. Band 18, Nummer 2, Februar 2020, S. 67–83, doi:10.1038/s41579-019-0299-x, PMID 31857715 (Review).
- Rafael Pinilla-Redondo, David Mayo-Muñoz, Jakob Russel, Roger A. Garrett, Lennart Randau, Søren J. Sørensen, Shiraz A. Shah: Type IV CRISPR–Cas systems are highly diverse and involved in competition between plasmids. In: Nucleic Acids Research. 27. Dezember 2019, doi:10.1093/nar/gkz1197.
- O. Shalem, N. E. Sanjana, F. Zhang: High-throughput functional genomics using CRISPR-Cas9. In: Nature reviews. Genetics. Band 16, Nummer 5, Mai 2015, S. 299–311, doi:10.1038/nrg3899, PMID 25854182, PMC 4503232 (freier Volltext) (Review).
- Y. Wang, Z. Li, J. Xu, B. Zeng, L. Ling, L. You, Y. Chen, Y. Huang, A. Tan: The CRISPR/Cas System mediates efficient genome engineering in Bombyx mori. In: Cell research. Band 23, Nummer 12, Dezember 2013, S. 1414–1416, ISSN 1748-7838. doi:10.1038/cr.2013.146. PMID 24165890. PMC 3847576 (freier Volltext).
- T. R. Sampson, D. S. Weiss: Exploiting CRISPR/Cas systems for biotechnology. In: BioEssays : news and reviews in molecular, cellular and developmental biology. Band 36, Nummer 1, Januar 2014, S. 34–38, ISSN 1521-1878. doi:10.1002/bies.201300135. PMID 24323919.
- D. L. Wagner, L. Amini, D. J. Wendering, L. M. Burkhardt, L. Akyüz, P. Reinke, H. D. Volk, M. Schmueck-Henneresse: High prevalence of Streptococcus pyogenes Cas9-reactive T cells within the adult human population. In: Nature medicine. [elektronische Veröffentlichung vor dem Druck] Oktober 2018, doi:10.1038/s41591-018-0204-6, PMID 30374197.
- J. M. Crudele, J. S. Chamberlain: Cas9 immunity creates challenges for CRISPR gene editing therapies. In: Nature Communications. Band 9, Nummer 1, August 2018, S. 3497, doi:10.1038/s41467-018-05843-9, PMID 30158648, PMC 6115392 (freier Volltext).
- Sascha Karberg: Molekulare Medizin: Rückschlag für Gentherapie mit "Crispr". In: tagesspiegel.de. 22. Februar 2018, abgerufen am 6. November 2018.
- Harald Frater: CRISPR/Cas9: Genschere mit Nebenwirkungen: Cas9-Enzym der Genschere könnte im Körper ungewollte Immunreaktionen auslösen. In: scinexx.de. 2. November 2018, abgerufen am 6. November 2018.
- G. Lin, K. Zhang, J. Li: Application of CRISPR/Cas9 Technology to HBV. In: International journal of molecular sciences. Band 16, Nummer 11, 2015, S. 26077–26086, doi:10.3390/ijms161125950, PMID 26540039 (Review).
- L. Ye, J. Wang, A. I. Beyer, F. Teque, T. J. Cradick, Z. Qi, J. C. Chang, G. Bao, M. O. Muench, J. Yu, J. A. Levy, Y. W. Kan: Seamless modification of wild-type induced pluripotent stem cells to the natural CCR5Δ32 mutation confers resistance to HIV infection. In: Proceedings of the National Academy of Sciences. Band 111, Nummer 26, Juli 2014, S. 9591–9596, doi:10.1073/pnas.1407473111, PMID 24927590, PMC 4084478 (freier Volltext).
- G. Wang, N. Zhao, B. Berkhout, A. T. Das: CRISPR-Cas based antiviral strategies against HIV-1. In: Virus research. [elektronische Veröffentlichung vor dem Druck] Juli 2017, doi:10.1016/j.virusres.2017.07.020, PMID 28760348.
- F. J. Sánchez-Rivera, T. Jacks: Applications of the CRISPR-Cas9 system in cancer biology. In: Nature reviews. Cancer. Band 15, Nummer 7, Juli 2015, S. 387–395, doi:10.1038/nrc3950, PMID 26040603, PMC 4530801 (freier Volltext).
- S. Chen, H. Sun, K. Miao, C. X. Deng: CRISPR-Cas9: from Genome Editing to Cancer Research. In: International journal of biological sciences. Band 12, Nummer 12, 2016, S. 1427–1436, doi:10.7150/ijbs.17421, PMID 27994508, PMC 5166485 (freier Volltext).
- T. Wijshake, D. J. Baker, B. van de Sluis: Endonucleases: new tools to edit the mouse genome. In: Biochimica et Biophysica Acta. Band 1842, Nummer 10, Oktober 2014, S. 1942–1950, doi:10.1016/j.bbadis.2014.04.020, PMID 24794718.
- H. L. Li, P. Gee, K. Ishida, A. Hotta: Efficient genomic correction methods in human iPS cells using CRISPR-Cas9 system. In: Methods (San Diego, Calif.). [elektronische Veröffentlichung vor dem Druck] Oktober 2015, doi:10.1016/j.ymeth.2015.10.015, PMID 26525194.
- T. Horii, I. Hatada: Genome Editing Using Mammalian Haploid Cells. In: International journal of molecular sciences. Band 16, Nummer 10, 2015, S. 23604–23614, doi:10.3390/ijms161023604, PMID 26437403, PMC 4632716 (freier Volltext) (Review).
- E. A. Vasileva, O. U. Shuvalov, A. V. Garabadgiu, G. Melino, N. A. Barlev: Genome-editing tools for stem cell biology. In: Cell death & disease. Band 6, 2015, S. e1831, doi:10.1038/cddis.2015.167, PMID 26203860, PMC 4650720 (freier Volltext) (Review).
- N. Savić, G. Schwank: Advances in therapeutic CRISPR/Cas9 genome editing. In: Translational research : the journal of laboratory and clinical medicine. [elektronische Veröffentlichung vor dem Druck] September 2015, doi:10.1016/j.trsl.2015.09.008, PMID 26470680.
- Doctors use gene editing tool Crispr inside body for first time (en). In: the Guardian, 4. März 2020. Abgerufen am 6. April 2020.
- Doctors try 1st CRISPR editing in the body for blindness. In: AP NEWS, 4. März 2020. Abgerufen am 6. April 2020.
- Franny White: OHSU performs first-ever CRISPR gene editing within human body (en) In: OHSU News. Abgerufen am 12. April 2020.
- Doctors use gene editing tool Crispr inside body for first time (en). In: the Guardian, 4. März 2020. Abgerufen am 6. April 2020.
- Franny White: OHSU performs first-ever CRISPR gene editing within human body (en) In: OHSU News. Abgerufen am 12. April 2020.
- More early data revealed from landmark CRISPR gene editing human trial (en). In: New Atlas.
- CRISPR Therapeutics and Vertex Announce New Clinical Data for Investigational Gene-Editing Therapy CTX001™ in Severe Hemoglobinopathies at the 25th Annual European Hematology Association (EHA) Congress | CRISPR Therapeutics (en) In: crisprtx.gcs-web.com. Abgerufen im 1 July 2020.
- Jocelyn Kaiser: CRISPR injected into the blood treats a genetic disease for first time (en). In: Science | AAAS, 26 June 2021. Abgerufen am 11. Juli 2021.
- Julian D. Gillmore, Ed Gane, Jorg Taubel, Justin Kao, Marianna Fontana, Michael L. Maitland, Jessica Seitzer, Daniel O’Connell, Kathryn R. Walsh, Kristy Wood, Jonathan Phillips, Yuanxin Xu, Adam Amaral, Adam P. Boyd, Jeffrey E. Cehelsky, Mark D. McKee, Andrew Schiermeier, Olivier Harari, Andrew Murphy, Christos A. Kyratsous, Brian Zambrowicz, Randy Soltys, David E. Gutstein, John Leonard, Laura Sepp-Lorenzino, David Lebwohl: CRISPR-Cas9 In Vivo Gene Editing for Transthyretin Amyloidosis. In: New England Journal of Medicine. 26. Juni 2021. doi:10.1056/NEJMoa2107454. PMID 34215024.
- L. S. Qi, M. H. Larson, L. A. Gilbert, J. A. Doudna, J. S. Weissman, A. P. Arkin, W. A. Lim: Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. In: Cell. Band 152, Nummer 5, Februar 2013, S. 1173–1183, ISSN 1097-4172. doi:10.1016/j.cell.2013.02.022. PMID 23452860. PMC 3664290 (freier Volltext).
- M. H. Larson, L. A. Gilbert, X. Wang, W. A. Lim, J. S. Weissman, L. S. Qi: CRISPR interference (CRISPRi) for sequence-specific control of gene expression. In: Nature protocols. Band 8, Nummer 11, November 2013, S. 2180–2196, ISSN 1750-2799. doi:10.1038/nprot.2013.132. PMID 24136345. PMC 3922765 (freier Volltext).
- B. Chen, L. A. Gilbert, B. A. Cimini, J. Schnitzbauer, W. Zhang, G. W. Li, J. Park, E. H. Blackburn, J. S. Weissman, L. S. Qi, B. Huang: Dynamic Imaging of Genomic Loci in Living Human Cells by an Optimized CRISPR/Cas System. In: Cell. Band 155, Nummer 7, Dezember 2013, S. 1479–1491, ISSN 1097-4172. doi:10.1016/j.cell.2013.12.001. PMID 24360272.
- X. Liu, S. Wu, J. Xu, C. Sui, J. Wei: Application of CRISPR/Cas9 in plant biology. In: Acta pharmaceutica Sinica. B. Band 7, Nummer 3, Mai 2017, S. 292–302, doi:10.1016/j.apsb.2017.01.002, PMID 28589077, PMC 5443236 (freier Volltext).
- Huw D. Jones: Are plants engineered with CRISPR technology genetically modified organisms? In: The Biochemist. Band 38, Nr. 3, Juni 2016, S. 14–17.
- Scott M. Schaeffer, Paul A. Nakata: CRISPR/Cas9-mediated genome editing and gene replacement in plants: Transitioning from lab to field. In: Plant Science. Band 240, 2015, S. 130–142, doi:10.1016/j.plantsci.2015.09.011.
- Teodoro Cardi, C. Neal Stewart Jr.: Progress of targeted genome modification approaches in higher plants. In: Plant Cell Reports. Band 35, 2016, S. 1401–1416, doi:10.1007/s00299-016-1975-1.
- Monsanto cuts deal to use CRISPR to engineer food. New Scientist, 23. September 2016.
- Erin Brodwin: The next generation of GMO food is here, and it's technically not a GMO. Business Insider, 18. April 2016.
- Evert Boonstra, Roy de Kleijn, Lorenza S. Colzato, Anneke Alkemade, Birte U. Forstmann, Sander Nieuwenhuis: Neurotransmitters as food supplements: the effects of GABA on brain and behavior. In: Frontiers in Psychology. 6, 6. Oktober 2015. doi:10.3389/fpsyg.2015.01520. PMID 26500584. PMC 4594160 (freier Volltext).
- Tomato In Japan Is First CRISPR-Edited Food In The World To Go On Sale (en). In: IFLScience. Abgerufen am 18. Oktober 2021.
- Stefanie Kurtz et al.: Knockout of the HMG domain of the porcine SRY gene causes sex reversal in gene-edited pigs. In: PNAS. 2021, doi:10.1073/pnas.2008743118.
- L. A. Marraffini, E. J. Sontheimer: CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA. In: Science. Band 322, Nummer 5909, Dezember 2008, S. 1843–1845, doi:10.1126/science.1165771, PMID 19095942, PMC 2695655 (freier Volltext).
- E. Pennisi: The CRISPR craze. In: Science. Band 341, Nummer 6148, August 2013, S. 833–836, doi:10.1126/science.341.6148.833, PMID 23970676.
- Martin Schlak, DER SPIEGEL: Crispr-Cas9: Wie ein litauischer Biochemiker die Chance seines Lebens verpasste. Abgerufen am 7. Oktober 2020.
- G. Gasiunas, R. Barrangou, P. Horvath, V. Siksnys: Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. In: Proceedings of the National Academy of Sciences. Band 109, Nummer 39, September 2012, S. E2579–E2586, doi:10.1073/pnas.1208507109, PMID 22949671, PMC 3465414 (freier Volltext).
- Streit um Ruhm, Ehre und Millionen Dollar, Zeit, 22. Juni 2016.
- E. S. Lander: The Heroes of CRISPR. In: Cell. Band 164, Nummer 1–2, Januar 2016, S. 18–28, doi:10.1016/j.cell.2015.12.041, PMID 26771483.
- Tracy Vence: “Heroes of CRISPR” Disputed. In: The Scientist, 19. Januar 2016.
- Joel Achenbach: Eric Lander talks CRISPR and the infamous Nobel ‘rule of three’. In: The Washington Post, 21. April 2016.
- Stephen S. Hall: The Embarrassing, Destructive Fight over Biotech's Big Breakthrough. In: Scientific American, 4. Februar 2016.
- Recipients Of The 2015 Breakthrough Prizes In Fundamental Physics And Life Sciences Announced. In: breakthroughprize.org. 9. November 2014, archiviert vom Original am 13. September 2016; abgerufen am 7. März 2018 (englisch).
- A. Pawluk: CRISPR: No Sign of Slowing Down. In: Cell. Band 174, Nummer 5, August 2018, S. 1039–1041, doi:10.1016/j.cell.2018.08.010, PMID 30142340.
- Heidi Ledford: Titanic clash over CRISPR patents turns ugly. In: Nature. Band 537, 22. September 2016, S. 460–461, doi:10.1038/537460a.
- Interference 106,048, USPTO, 15. Februar 2017.
- A. Plaza Reyes, F. Lanner: Towards a CRISPR view of early human development: applications, limitations and ethical concerns of genome editing in human embryos. In: Development. Band 144, Nummer 1, 01 2017, S. 3–7, doi:10.1242/dev.139683, PMID 28049687.
- Jean-François Gariépy: The revolutionary phenotype. Hrsg.: Elora Editions. 2019, ISBN 978-1-72986-156-1, S. 93–96.
- Crispr. Chinesische Forscher wegen Genmanipulation zu Haftstrafen verurteilt. In: Zeit Online. 30. Dezember 2019, abgerufen am 13. August 2020.
- Gerichtshof der Europäischen Union: Pressemitteilung Nr. 111/18 – Urteil in der Rechtssache C-528/16. In: Europäischer Gerichtshof. 25. Juli 2018, abgerufen am 27. Juli 2018. bzw. Ergebnisliste C-528/16
- Crispr/Cas-Urteil des EuGH: Der lange Schatten der Ideologien. In: spektrum.de. 25. Juli 2018, abgerufen am 21. Januar 2019.
- Ewen Callaway: CRISPR plants now subject to tough GM laws in European Union. In: Nature. Band 560, Nr. 7716, 25. Juli 2018, ISSN 0028-0836, S. 16–16, doi:10.1038/d41586-018-05814-6 (nature.com [abgerufen am 3. August 2018]).
- Kai Kupferschmidt: EU verdict on CRISPR crops dismays scientists. In: Science. Band 361, Nr. 6401, 3. August 2018, ISSN 0036-8075, S. 435–436, doi:10.1126/science.361.6401.435, PMID 30072518 (sciencemag.org [abgerufen am 3. August 2018]).